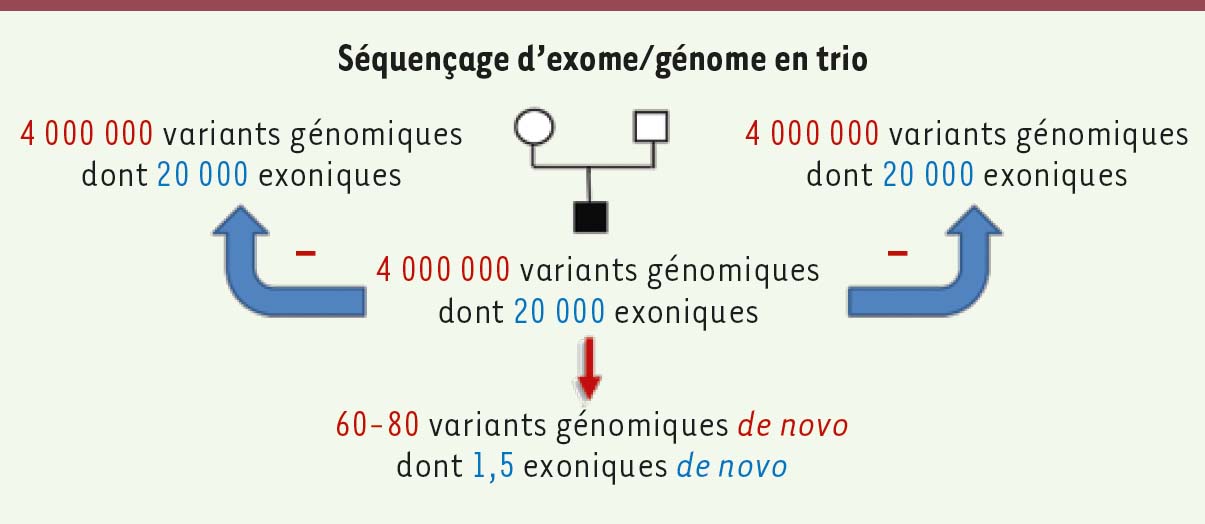
L’exome, qui représente 1,5 % du génome compte 20 000 variations en moyenne par individu, dont 1 000 sont considérées comme rares. Parmi celles-ci, 1,5 en moyenne est survenue de novo. C’est parmi les variations rares que l’on cherche celles qui peuvent être responsables d’une maladie génétique. Le défi n’est pas de les détecter, mais de les interpréter et de les classer.
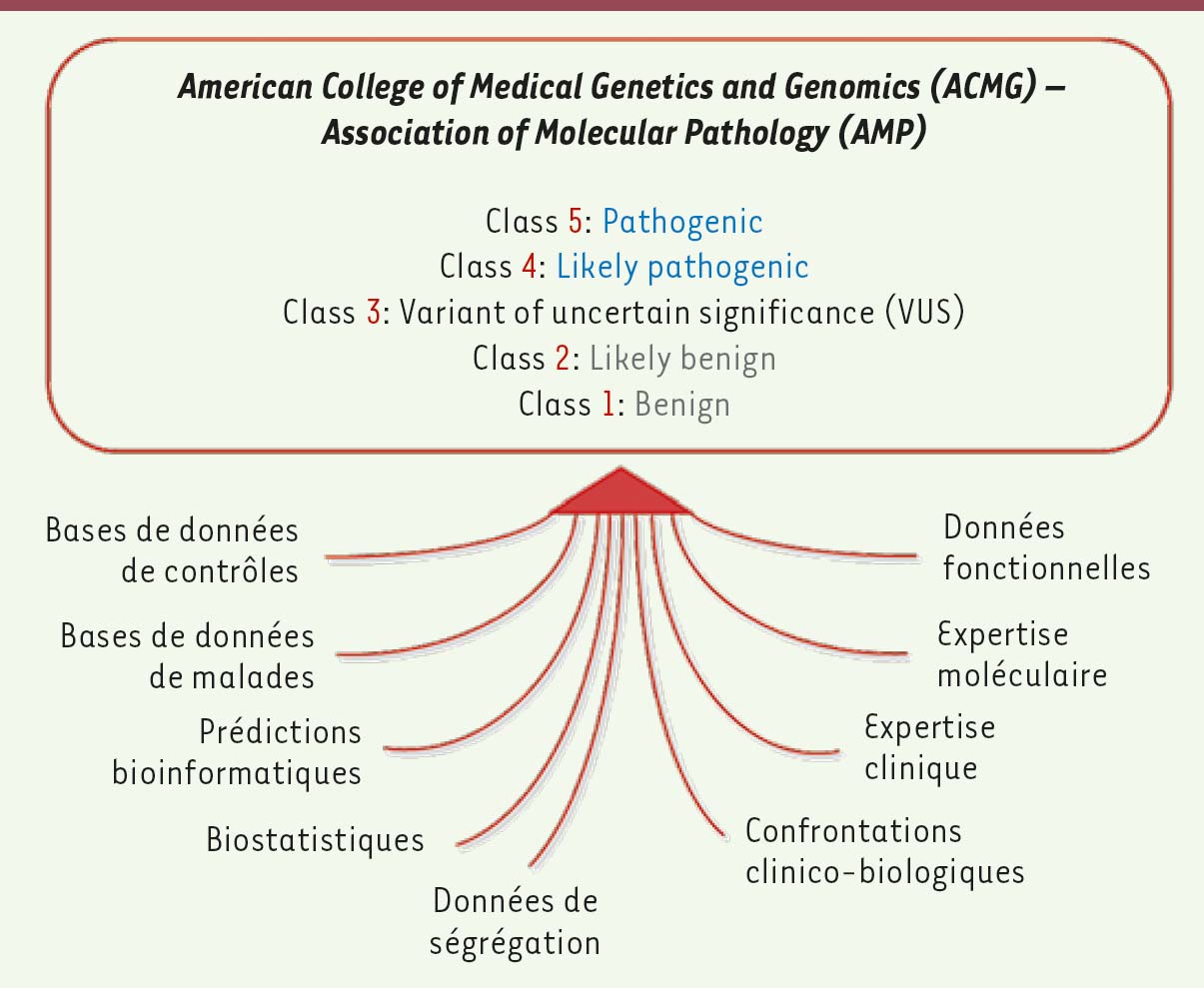
Des recommandations américaines proposent de classer toutes les variations du génome en cinq classes : bénin, probablement bénin, de signification inconnue, probablement pathogène ou pathogène. Il faut alors regrouper un ensemble d’arguments et faire preuve d’une grande expertise pour interpréter ces variants, en apportant une précision au niveau du phénotype du patient, une expertise moléculaire, bioinformatique, biostatistique, des données fonctionnelles et de la littérature (Figure 2).
 | Figure 2 Recommandations de l’ACMG. |
Différentes stratégies peuvent être appliquées. De plus en plus, on va vers l’exome, voire le génome entier pour séquencer l’ensemble du génome et s’intéresser ensuite à un certain nombre de gènes en particulier, ce qui s’appelle des panels de gènes in silico.
Les avantages de l’exome et du génome sont la couverture, le nombre d’exons séquencés au total. Le génome est considéré comme le meilleur outil pour séquencer l’exome. Le génome est également meilleur pour détecter des variants de structure et des variations du nombre de copies (délétions, duplications).
Le séquençage de panels de gènes permet de séquencer de manière plus profonde et de détecter des variations somatiques (post-zygotiques). Lorsqu’il apparaît que le phénotype peut être lié à une mutation somatique dans un gène particulier associé un phénotype spécifique, il est encore intéressant d’avoir recours à des panels.
En conclusion, le séquençage à haut débit représente une révolution dans la recherche, avec l’identification de la diversité du génome humain, la dissection des bases moléculaires des maladies mendéliennes ou complexes. On assiste également à une révolution médicale, du fait de la rapidité du diagnostic, de la diminution de l’errance diagnostique et de l’accès plus rapide à une prise en charge précoce, un suivi adapté, un traitement médical personnalisé et une prise en charge adaptée des familles.
L’avenir de la génomique est le développement et l’accès au séquençage du génome pour les patients. Pour cela, il nous faut mieux comprendre les variations codantes, les variations non codantes, les interactions entre les gènes et avec l’environnement, afin de permettre une médecine de précision basée sur l’interprétation des variations génomiques individuelles.