Vignette (Photo © CNRS-IGS). Coupe ultrafine de particule de Pandoravirus (microscopie électronique).
L’histoire des virus géants a débuté en 2003, avec la publication de la nature virale et la description détaillée de Mimivirus [1, 2]. Par la taille de sa particule (0,7 µm de diamètre) et celle de son génome de plus d’un million de nucléotides, Mimivirus démontrait qu’aucune loi biologique n’interdisait à un virus d’être aussi complexe qu’une cellule bactérienne. Depuis, les découvertes successives ont constitué une véritable saga, avec l’apparition continuelle de nouveaux personnages, de familles (virales) sans cesse recomposées [3] (→) et de multiples épisodes de combats entre les chercheurs tentant de reconstruire leur évolution au travers de récits contradictoires [4-6]. Au-delà d’un simple tumulte interne à la virologie, les virus géants ont initié et continuent d’alimenter une réflexion profonde sur la notion même de virus, d’organisme vivant, et sur le rôle qu’ils auraient pu jouer dans l’émergence et l’évolution du monde cellulaire [4-9]. Au-delà des laboratoires, les virus géants deviennent l’objet de réflexions épistémologiques [10, 11].
(→) Voir la Synthèse de J.M. Claverie et al., m/s n° 12, décembre 2016, page 1087
Au sein des familles distinctes de virus géants connues à ce jour (Tableau I), l’une d’entre elles a récemment été l’objet d’une attention particulière, celle des Pandoraviridae1. Publiée en 2013 [12], la découverte des deux premiers membres de cette nouvelle famille (Pandoravirus salinus et P. dulcis) a eu un grand retentissement à plusieurs titres. Elle a d’abord démontré à la surprise générale que le gigantisme viral n’était pas l’apanage de la seule famille des Mimivirus, les Mimiviridae. Cette deuxième famille de virus géants laissait prévoir l’existence de plusieurs autres familles bien distinctes, dont la découverte ne s’est pas fait attendre [13-15], et dont la liste n’est probablement pas terminée. Les premiers pandoravirus ont aussi démontré que nous n’avions pas atteint la limite de taille des particules virales (plus d’un µm) ni celle de leur génome. Avec 2,5 millions de nucléotides, P. salinus atteignait la complexité génétique de certains parasites eucaryotes, comme les microsporidies. Il reste néanmoins tributaire, comme tous les virus, de la machinerie de traduction de son hôte amibien pour la synthèse de ses propres protéines.
Tableau I.
Liste des familles de virus « géants » découvertes depuis Mimivirus. Ces familles comptent toutes des membres se multipliant dans les amibes du genre Acanthamoeba. Les trois dernières lignes correspondent à des virus dont les particules sont à la limite de visibilité au microscope optique. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Avec seulement deux membres de la famille des Pandoraviridae, il était néanmoins impossible d’aller plus loin dans l’étude des mécanismes évolutifs susceptibles d’engendrer de tels monstres au sein du monde viral. Nous sommes alors retournés sur le terrain, à la recherche d’échantillons environnementaux susceptibles de contenir d’autres souches de cette famille. Nous avons eu la chance d’en isoler 3 nouvelles, dont la comparaison avec nos deux souches initiales et avec une autre décrite par un laboratoire allemand [16] nous a permis de montrer l’insuffisance des scénarios évolutifs qui s’affrontent traditionnellement pour tenter de rendre compte de la complexité et de « l’étrangeté » du génome des virus géants.
Avec de nombreuses nuances, deux types de scénarios principaux s’affrontent : celui développé par les réductionnistes et celui des inflationnistes [4-9]. Les réductionnistes s’appuient sur une loi amplement documentée par de multiples exemples, qui veut que les organismes parasites (et en particulier les bactéries strictement intracellulaires) soient acculés à une dépendance de plus en plus marquée vis-à-vis de leur hôte, par la perte progressive et irréversible des gènes dont la fonction peut être assurée par la cellule qu’ils infectent [17-20]. Selon cette école de pensée, les virus sont le résultat d’une évolution réductive permanente qui les a conduits d’un stade ancestral quasi-cellulaire aux plus petits virus que nous connaissons actuellement. Dans ce contexte, les virus « géants » seraient ceux dont l’évolution a été particulièrement lente. Cette hypothèse rend particulièrement bien compte de la famille des Mimiviridae, dont le génome code de nombreuses fonctions liées à la traduction des protéines, un domaine d’activité considéré (depuis l’énoncé des critères de Lwoff)2 [21] comme le strict apanage du monde cellulaire. Selon cette école, les différentes familles de virus que nous connaissons seraient les résultats de pertes différentielles de gènes à partir de formes ancestrales plus complexes, elles-mêmes issues de lignées proto-cellulaires éventuellement différentes de l’ancêtre commun du monde cellulaire contemporain [4, 7].
Pour les inflationnistes, le scénario est exactement inversé. Les virus ont commencé par être des entités très simples et très petites, leur génome ne codant que les fonctions absolument nécessaires à leur réplication (i.e. une ADN polymérase) et à leur encapsidation. Selon cette école, les différentes familles de virus que nous connaissons sont les résultats d’acquisitions (dites « latérales ») de gènes provenant du monde cellulaire ou d’autres virus [5].
Une école « centriste », prône une théorie intermédiaire selon laquelle l’évolution des virus s’effectue en « accordéon » par une alternance de phases de réduction et d’inflation [22].
Chacune de ces théories, même si elles s’opposent, se situe dans un contexte historique dans lequel les gènes viraux proviennent d’une version ancestrale soit héritée d’un virus parent, soit acquise d’un organisme cellulaire. Pour des raisons différentes, ces deux scénarios ne fournissent pas d’explications satisfaisantes à l’existence des virus géants, ni à la composition de leur génome, en particulier à la proportion énorme de leurs gènes codant des protéines orphelines (ou ORFans), sans homologue dans le monde cellulaire ou viral connu à ce jour.
L’hypothèse « réductionniste » se heurte principalement à la difficulté de rendre compte de la grande diversité génétique observée entre différents membres de la même famille (dont moins de la moitié des gènes peuvent être partagés). Chaque génome viral contemporain étant vu comme un sous-ensemble de gènes sélectionnés à partir d’un génome ancestral plus complexe, la taille prédite de celui-ci devient difficilement soutenable lorsque qu’elle vient à dépasser le nombre de gènes (environ 260) qui sont nécessaires au métabolisme minimal d’une bactérie non parasite [23]. D’un autre côté, comme les nombreux gènes spécifiques à un seul virus sont majoritairement constitués d’ORFans, on ne peut pas non plus en attribuer l’origine à une acquisition latérale.
Selon l’école opposée des inflationnistes, les virus ont commencé tout petit (un ou deux gènes, qui forment le noyau des gènes communs à toutes les familles de virus). Ils ont ensuite évolué en acquérant des gènes de leurs hôtes ou d’autres organismes les environnant. Nous avons ici deux difficultés.
– La première est d’expliquer quelles forces pousseraient ces virus à évoluer à contresens de la réduction que l’on constate chez tous les parasites intracellulaires (et de la majorité des virus qui se satisfont d’une dizaine de gènes).
– La seconde est que l’origine des gènes supposément « acquis » serait passablement mystérieuse étant donné l’énorme proportion d’ORFans qui les caractérisent (jusqu’à 90 % pour les pandoravirus). Si la plupart de ces gènes avait été empruntée, ceux-ci devraient appartenir à des catégories d’organismes dont nous n’avons encore jamais rencontré le moindre représentant3.
L’hypothèse de l’évolution en accordéon ne résout en rien les difficultés précédentes, dont la principale est de rendre compte de l’énorme proportion d’ORFans dans les génomes viraux (de 65 % à 93 %, pour chaque nouvelle famille découverte). C’est à la solution de ce mystère que s’est attaqué notre récent travail sur les Pandoravirus [24].
Après avoir isolé et caractérisé en détail trois nouveaux membres de cette famille (Tableau II), nous avons entrepris une comparaison globale des 6 génomes disponibles. Ce travail a d’abord consisté en une ré-annotation rigoureuse de ces génomes (tirant partie d’analyses transcriptomiques et protéomiques complémentaires) qui a permis d’éliminer tous les gènes (et protéines correspondantes) dont la prédiction pouvait être douteuse, notamment en raison de la proportion inhabituellement élevée de G+C (> 60 %) dans le génome des Pandoravirus4.
Tableau II.
Liste des différents isolats de Pandoravirus caractérisés à ce jour. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
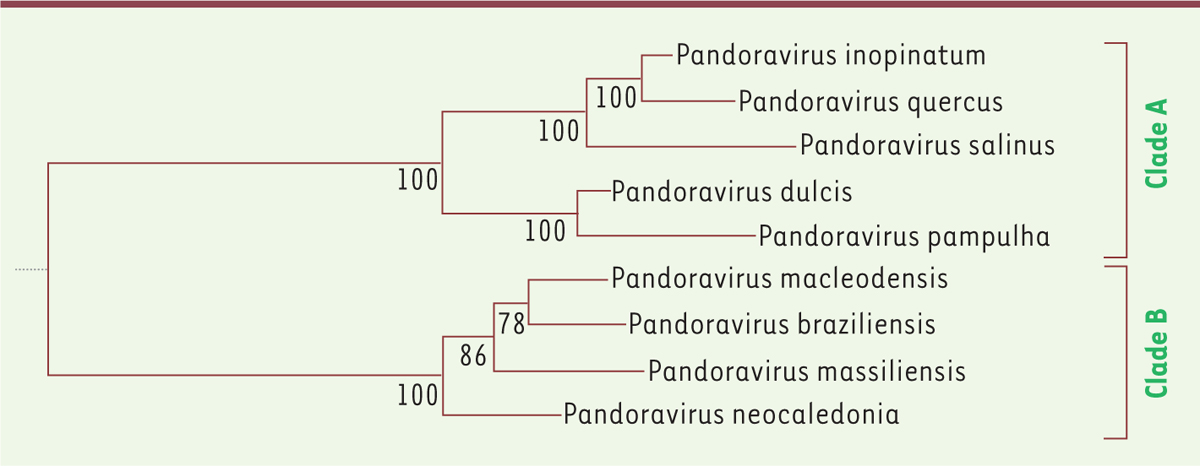
Après avoir éliminé le risque de comptabiliser des gènes faussement prédits comme autant d’ORFans, nous avons procédé à une nouvelle analyse comparative du contenu génique des différents Pandoravirus, dont la famille semble déjà se diviser en deux branches (ou clades), une tendance confirmée par la position de 3 autres isolats décrits depuis la publication de notre étude (Figure 1) [25].
| Figure 1. Classification des Pandoraviridae. Bien qu’encore peu fournie, la famille des Pandoravirus se divise déjà clairement en deux sous-groupes (clades A et B) engagés dans un processus de diversifications distinctes. À partir des similarités constatées entre les séquences de l’ADN polymérase de chacun des virus, cet arbre en reconstitue la généalogie la plus probable. La confiance associée à chaque embranchement est indiquée par un pourcentage. Cet arbre et ces valeurs sont calculés avec un algorithme standard (IQ-Tree [35]). |
Nos analyses ont confirmé que le gigantisme du génome des Pandoravirus (deux fois plus grand que celui des Mimiviridae) n’était pas la conséquence d’une propension anormalement élevée à l’acquisition latérale de gènes, ni à la duplication de gènes préexistants. Pourtant, nous avons constaté que chaque nouvel isolat contribuait pour environ 50 nouveaux gènes (dont 95 % d’ORFans) au « pan génome » des Pandoraviridae. L’origine de ces gènes, bien que leur nombre ait été réduit de moitié par notre annotation très conservatrice, restait donc un mystère.
La solution de cette énigme est alors venue de la découverte d’un gradient de similarité (évalué par 3 statistiques indépendantes) entre les régions intergéniques et les gènes spécifiques d’un virus donné (95 % d’ORFans), puis les gènes spécifiques à chaque clade (90 % d’ORFans), puis les gènes communs à tous les isolats (65 % d’ORFans). En fait, plus un gène est orphelin (partagé uniquement par les Pandoravirus, ou seulement partagé au sein d’un des clades, ou spécifique d’un seul isolat) et plus sa « signature » ressemble à celle des régions non-codantes séparant des gènes successifs [24]. En l’absence d’autres hypothèses capables d’expliquer une telle relation, le scénario le plus plausible que nous proposons est que les gènes/protéines spécifiques à chaque Pandoravirus soient issus d’un processus de création de novo et in situ à partir de séquences d’ADN intergéniques initialement non-codantes.
Une telle hypothèse vient se heurter de plein fouet au paradigme actuel qui veut qu’un peptide résultant d’une séquence aléatoire n’ait qu’une probabilité infime d’exercer une fonction qui, de plus, confèrerait un avantage à son porteur. En l’absence du moindre avantage sélectif, ces nouveaux gènes devraient donc être éliminés avant d’avoir pu évoluer vers la moindre utilité. Le fait qu’ils ne le soient pas suggère que toutes les forces gouvernant l’évolution des Pandoravirus n’ont pas encore été identifiées ou comprises.
Ce scénario, qui rompt avec le concept fondamental que tous les gènes ont une histoire (c’est-à-dire qu’ils sont en continuité avec une forme ancestrale), semble pourtant le seul capable de rendre compte de l’extrême altérité et de la diversité du contenu génétique des Pandoravirus, et peut-être des autres virus géants.
Notons que cette boîte de Pandore conceptuelle a déjà été ouverte depuis longtemps par divers auteurs explorant les génomes d’organismes bien plus standard que les virus géants, comme la levure [26], la plante Arabidopsis thaliana [27], différent animaux [28-31], y compris les primates [32]. Quelle que soit notre incrédulité résiduelle devant le côté magique de ce scénario, il nous faut bien admettre qu’il a bien fallu qu’un jour soient créés de novo les ancêtres de nos gènes, dans des conditions certainement bien moins favorables que celles régnant dans le monde vivant d’aujourd’hui.
Nous ne pouvons qu’espérer que notre étude de la diversité des Pandoravirus et nos spéculations sur leur évolution contribuent à relancer des travaux sur un processus originel étonnamment absent des préoccupations des évolutionnistes contemporains, pour la plupart occupés à construire des arbres dont nous ne comprenons toujours pas les racines.