| |
| Med Sci (Paris). 34(12): 1071–1078. doi: 10.1051/medsci/2018299.Le virus de l’hépatite E Un virus méconnu qui se dévoile Maliki Ankavay,1 Jean Dubuisson,1 and Laurence Cocquerel1* 1Université de Lille, CNRS, Inserm, CHU Lille, Institut Pasteur de Lille, U1019-UMR8204-CIIL- Center for Infection and Immunity of Lille1, rue du Professeur Calmette, F-59000Lille, France |
Vignette (© État d’Urgence Production / DNDi / Fondation Mérieux / Inserm / Institut Pasteur / MSF / Réseau Canopé / Universciences / Réalisateur : Gérard Lafon). Alors que les hépatites virales A, B et C sont connues du grand public, l’hépatite E est généralement méconnue. Cette infection, dont l’agent responsable est le virus de l’hépatite E (HEV, E pour epidemic/enteric), est pourtant maintenant la première cause d’hépatite aiguë et de jaunisse dans le monde. Deux milliards de personnes vivent dans des pays où cette infection est endémique et sont donc susceptibles de contracter une hépatite E. L’Organisation mondiale de la santé (OMS) estimait, en 2017, à 20 millions le nombre d’infections par le HEV chaque année, avec plus de 3,3 millions de cas d’hépatites E aiguës et 44 000 décès liés à cette infection. Le HEV est présent dans toutes les régions du monde, mais sa prévalence est la plus élevée en Asie de l’est et du sud. En France, la séroprévalence est de 22,4 % à l’échelle nationale. Elle est de 52,2 % dans le sud du pays, particulièrement touché par cette infection. Quatre génotypes majeurs (génotypes 1 à 4) du HEV sont pathogènes pour l’homme. Ils présentent des différences épidémiologiques. Les génotypes 1 et 2 sont généralement rencontrés dans les pays en développement et sont présents exclusivement chez l’homme. Leur transmission se fait par voie féco-orale, essentiellement par consommation d’eau contaminée par les particules virales présentes dans les matières fécales. Les génotypes 3 et 4 sont, eux, essentiellement présents dans les pays industrialisés dans lesquels le nombre de cas rapportés a été multiplié par 10 ces dix dernières années, en raison de l’amélioration des outils diagnostiques et de la recherche du virus en cas d’hépatite aiguë [1]. Ils diffèrent des génotypes 1 et 2 par leur transmission, qui est principalement zoonotique. Ils sont en effet présents chez différents animaux domestiques et sauvages qui constituent des réservoirs viraux à partir desquels l’homme peut se contaminer [2]. La consommation de viande de porc ou de gibier, peu ou pas cuite ou sous la forme de salaison, représente une source importante de contamination. Le HEV est ainsi détectable dans 30 % des saucisses préparées à partir de foie de porc, comme les figatelli corses1 [3]. Le virus est en effet présent dans 65 % des élevages de porcs en France, 31 % des animaux se révèlent positifs pour le virus et 4 % des foies entrant dans la chaîne alimentaire sont infectés - une cuisson à cœur à 71 °C pendant 20 minutes permet cependant d’inactiver le virus [4]. Le HEV peut également se transmettre par voie parentérale, verticale et transplantationnelle [5]. L’hépatite E est asymptomatique chez les animaux ; elle est généralement aiguë mais spontanément résolutive chez l’homme et, la plupart du temps, ne nécessite pas d’hospitalisation. L’infection peut cependant engendrer de graves complications, notamment chez les femmes enceintes infectées par le génotype 1 ou 2, chez lesquelles l’hépatite E peut devenir fulminante, atteignant un taux de mortalité situé entre 15 et 25 %. Les personnes ayant une hépatopathie sous-jacente sont également très à risque. L’hépatite E peut également devenir chronique chez les patients immunodéprimés infectés par le génotype 3 [42] (→). (→) Voir la Nouvelle de J. Izopet et N. Kamar, m/s n° 12, décembre 2008, page 1023 Elle est aussi associée à un certain nombre de désordres extra-hépatiques, notamment neurologiques (syndrome de Guillain-Barré et amyotrophie névralgique) et rénaux (glomérulonéphrite) [6]. Bien que l’hépatite E représente un problème majeur de santé publique dans le monde, aucun traitement spécifique n’est aujourd’hui disponible. La Ribavirine peut cependant être utilisée dans la prise en charge de l’hépatite chronique chez les patients immunodéprimés. Un vaccin recombinant (Hecolin®) a été homologué en Chine en 2011 mais pas dans les autres pays, celui-ci n’ayant pas fait l’objet d’essais d’innocuité, d’immunogénicité et d’efficacité chez les enfants, les femmes enceintes, les personnes âgées et les personnes atteintes d’hépatopathie. Le diagnostic de l’hépatite E repose sur la détection du génome viral par amplification (PCR, polymerase chain reaction), la détection des anticorps anti-HEV (Ig [immunoglobuline] M et G) ou de l’antigène de capside. Les anticorps anti-HEV sont détectables dès le début de la symptomatologie : le taux est maximum au bout d’un mois et décroît après 2 à 6 mois pour les IgM ; les IgG persistent de 18 mois à plus de 10 ans. La présence d’IgG anti-HEV témoigne ainsi d’une exposition antérieure au virus. L’infection est considérée comme chronique si l’ARN viral reste détectable plus de 3 mois après l’infection. |
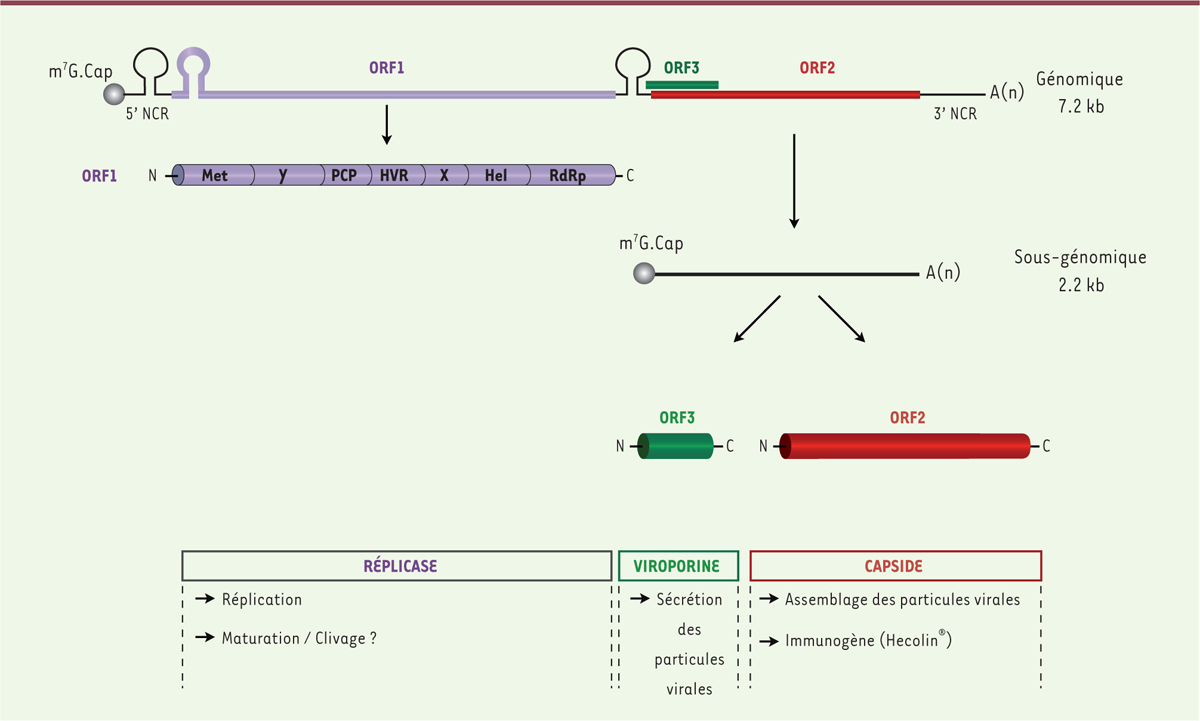
Le virus de l’hépatite E est un virus appartenant au genre Orthohepevirus de la famille des Hepeviridae [7]. C’est un petit virus d’environ 30-34 nm de diamètre qui possède une capside de symétrie icosaédrique [8, 9]. Son génome consiste en une molécule d’ARN monocaténaire de polarité positive ([+]) d’environ 7 200 nucléotides présentant respectivement à ses extrémités 5’ et 3’ une coiffe et une queue poly(A). Cet ARN possède trois cadres ouverts de lecture (ORF), nommés ORF1, ORF2 et ORF3, encadrés par deux régions non codantes (NCR) qui jouent un rôle essentiel dans la réplication et la transcription du génome viral. Les 3 ORF codent respectivement les protéines ORF1, ORF2 et ORF3. Les protéines ORF2 et ORF3 sont produites à partir d’un ARN sous-génomique bicistronique de 2,2 kilobases (ARNsg), dont le promoteur se situerait dans la région de jonction entre l’ORF1 et son codon d’initiation (Figure 1).
 | Figure 1. Organisation du génome du HEV. Le HEV possède un génome à ARN simple brin de polarité positive d’environ 7 200 nucléotides avec respectivement à ses extrémités 5’ et 3’ une coiffe (m7G.Cap) et une queue poly(A). Il présente trois cadres ouverts de lecture (ORF) encadrés par deux régions non codantes (NCR). Ces ORF codent respectivement trois protéines appelées ORF1, ORF2 et ORF3. La protéine ORF1 est une polyprotéine traduite à partir de l’ARN génomique qui est importante pour la réplication virale. Elle possède sept domaines : le domaine méthyl-transférase (Met) ; le domaine Y ; le domaine papaïne cystéine protéase (PCP) ; le domaine hypervariable (HVR) ; le macrodomaine X ; le domaine hélicase (Hel) et le domaine ARN polymérase ARN-dépendante (RdRp). Les protéines ORF2 et ORF3 sont synthétisées à partir d’un ARN sous-génomique bicistronique de 2,2 kb (ARNmSG). L’ORF2 est la protéine de capside virale. La protéine ORF3 joue un rôle important dans la sécrétion des particules virales. N : extrémité amino-terminale ; C : extrémité carboxy-terminale ; m7G.Cap : coiffe ; A(n) : queue poly(A). |
La protéine ORF1, également appelée réplicase, est une polyprotéine non-structurale de 1 693 acides aminés (aa) présentant plusieurs domaines distincts essentiels à la réplication virale (pour revue [10]). Des analyses de bioinformatique de sa séquence ont permis d’identifier des domaines (Figure 1) dont les activités enzymatiques ont ensuite été démontrées : méthyl-transférase (Met), ARN hélicase (Hel) et ARN polymérase ARN-dépendante (RdRp). En revanche, la fonction exacte du macrodomaine X, du domaine Y et de la région hypervariable (HVR) reste à définir. Une activité enzymatique chymotrypsine a été montrée pour le domaine papaïne cystéine protéase (PCP). Le clivage de la polyprotéine en plusieurs polypeptides n’a pas encore pu être confirmé. Néanmoins, il semble que le facteur de coagulation Xa et la thrombine seraient essentiels à la réplication du HEV et pourraient être impliqués dans la maturation de ORF1 [11]. La protéine ORF2 est la protéine de capside. Cette protéine de 660 aa possède un peptide signal N-terminal (correspondant aux 20 premiers aa) et trois sites potentiels de N-glycosylation. L’importance de cette modification post-traductionnelle dans le cycle infectieux du HEV reste à définir. La protéine ORF2 possède trois domaines distincts appelés S (shell), M (middle) et P (protruding). Le domaine P serait la cible majeure des anticorps neutralisants. Il pourrait contenir un domaine de liaison du virus à son récepteur cellulaire. Les monomères de capside s’assembleraient pour former des décamères qui encapsident l’ARN viral. La capside serait constituée de 180 copies disposées en icosaèdre [12-14]. La protéine ORF2 est hautement immunogène. Des études immunologiques et structurales de cette protéine de capside ont d’ailleurs contribué au développement du vaccin Hecolin® en Chine [15]. ORF2 est la protéine virale la mieux caractérisée, essentiellement grâce à des études portant sur des formes tronquées de la protéine produites dans des systèmes d’expression hétérologue utilisant le baculovirus ou la bactérie Escherichia coli. Ce n’est que très récemment que la séquence exacte de la protéine ORF2 associée aux particules virales infectieuses (ORF2i) a été identifiée : elle correspond aux résidus 14 à 660 de la protéine [9]. Au cours de son cycle infectieux, le HEV produit la protéine de capside ORF2 sous trois formes différentes : (1) la forme ORF2i, d’environ 80 kDa, qui est la forme associée aux particules infectieuses. Cette protéine n’est pas glycosylée ; (2) la forme ORF2g (glycosylée), d’environ 90 kDa ; et (3) la forme ORF2c (clivée), d’environ 75 kDa. Ces dernières sont des protéines sécrétées en très grandes quantités mais ne forment pas de particules virales et ne sont pas associées à du matériel infectieux. Elles sont N-glycosylées, O-glycosylées et sialylées. La forme ORF2c est un produit de clivage N-terminal de la protéine ORF2g (données non publiées). ORF2g et ORF2c sont les antigènes viraux majeurs présents dans le sérum des patients infectés par le HEV [9] ; elles pourraient représenter un leurre immunologique. Une étude très récente a en effet confirmé l’existence des différentes formes d’ORF2 et a montré que la forme ORF2g sécrétée pouvait moduler la réponse immunitaire en interférant avec la reconnaissance des particules virales par les anticorps neutralisants [16]. La forme ORF2c n’a pas été identifiée dans cette étude, probablement en raison des faibles quantités de cette protéine synthétisée ou du type cellulaire utilisé. La protéine ORF3 est une phosphoprotéine structurale de 113-115 acides aminés. Elle est constituée de deux domaines hydrophobes N-terminaux (D1 et D2) et de deux domaines riches en prolines (P1 et P2). Cette protéine de 13 kDa est présente à la surface des particules virales circulant dans le sérum de patients infectés ou produites en culture cellulaire [9, 17]. Le rôle exact de l’ORF3 dans le cycle infectieux du HEV n’a pas encore été pleinement établi. Néanmoins, il semble qu’elle ait un rôle critique dans la sécrétion des particules virales et très récemment une activité viroporine lui a été attribuée [18]. ORF3 interagit, via un motif PSAP (Pro-Ser-Ala-Pro) présent dans son domaine P2, avec la protéine Tsg101 (tumor susceptibility gene 101) [19, 20], un élément du complexe ESCRT (endosomal sorting complexes required for transport) important pour la formation des exosomes [43] (→) et requis pour le bourgeonnement de plusieurs virus enveloppés [21]. (→) Voir la Synthèse de S. Le Lay et al., m/s n° 11, novembre 2018, page 936 Bien qu’initialement considéré comme un virus non enveloppé, le HEV, comme le virus de l’hépatite A, est désormais identifié comme un virus quasi-enveloppé mais qui ne présente pas de protéine d’enveloppe à sa surface [22, 23]. Contrairement aux particules retrouvées dans les selles de patients, les virions produits en culture cellulaire et ceux qui circulent dans le sang des patients infectés, appelés eHEV, sont associés à des lipides et à la protéine ORF3. L’ultrastructure des particules enveloppées et non enveloppées a été visualisée récemment par microscopie électronique à transmission [9]. Les particules eHEV, associées à des lipides et à la protéine ORF3, seraient moins infectieuses que des particules non lipidées présentes dans les selles [24, 25], probablement du fait de l’utilisation de voies différentes d’entrée dans la cellule cible [23]. Il est à noter que l’enveloppe des eHEV pourrait protéger le virus des anticorps neutralisants [26]. |
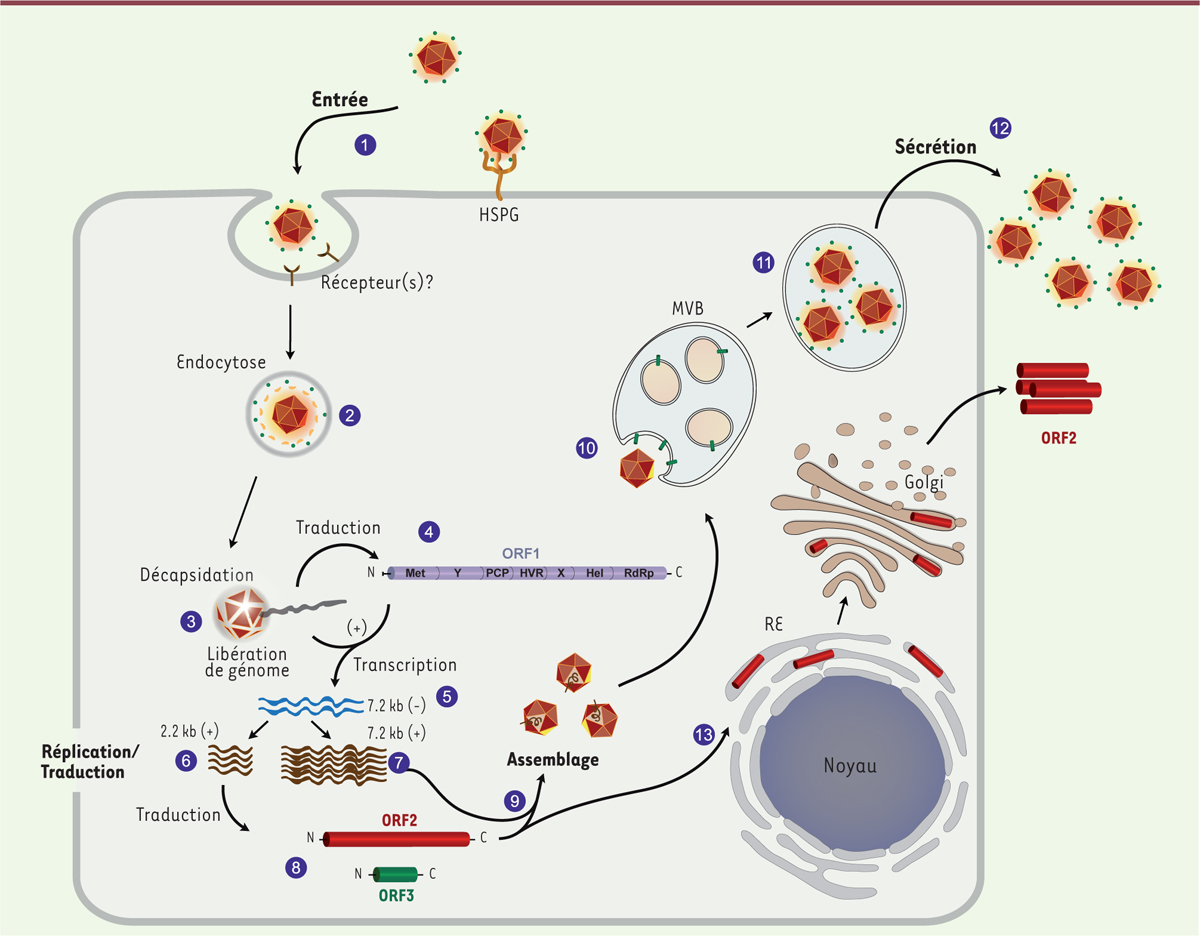
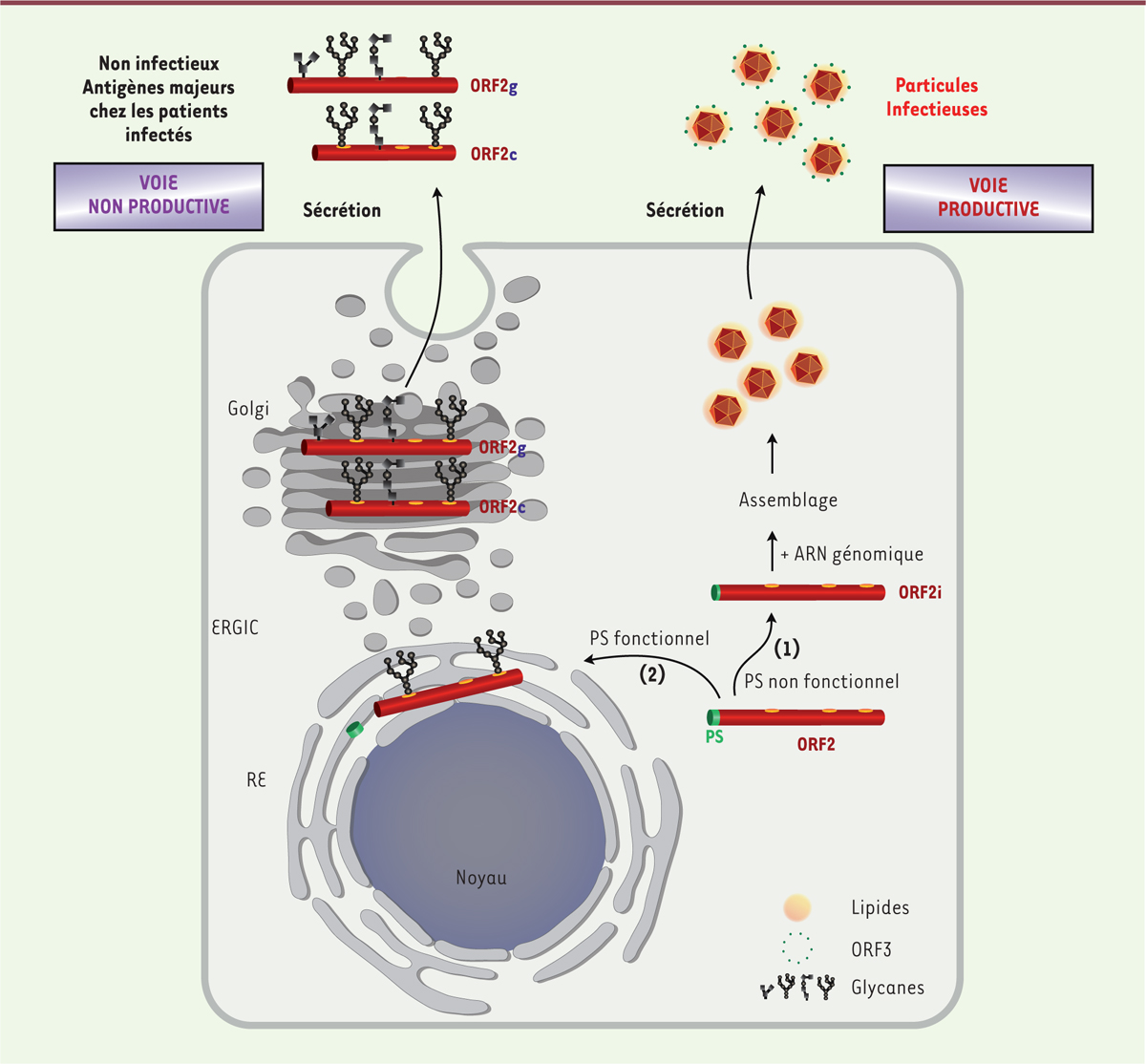
Le cycle infectieux du HEV En raison de la difficulté de l’amplifier en culture cellulaire, les différentes étapes du cycle infectieux du HEV restent encore très mal connues (Figure 2). Néanmoins, il est proposé qu’après son attachement non spécifique à la surface de la cellule cible via des protéoglycanes à héparane sulfate [27], le virus interagirait avec un ou des récepteurs qui restent à identifier. Les particules non enveloppées et les eHEV entreraient dans les cellules par des mécanismes différents [23] : l’entrée des eHEV impliquerait une endocytose, en suivant une voie dépendant de la clathrine, de la dynamine-2, de Rab5/Rab7 et du cholestérol [23, 28, 29] ; le mécanisme d’entrée des particules non enveloppées reste, lui, à définir. En entrant dans la cellule, les lipides de l’enveloppe des eHEV subiraient une dégradation par les lipases des lysosomes libérant la capside [23]. Après décapsidation, dont le mécanisme reste inconnu, l’ARN viral serait alors libéré dans le cytosol où il serait traduit par la machinerie cellulaire pour produire la réplicase ORF1. L’ORF1 serait à l’origine de la synthèse d’ARN simple brin de polarité négative (-) à partir de l’ARN génomique de polarité positive (+). Ces ARN(-) serviraient de matrice pour la synthèse de nouveaux ARN(+) génomiques de 7,2 kb, mais également des ARN sous-génomiques de 2,2 kb, ensuite cappés (coiffés) et polyadénylés. Les ARN sous-génomiques de polarité (+) sont traduits en protéines ORF2 et ORF3. ORF2 est massivement produite et pourrait suivre deux voies [9] (Figure 3) : (1) une voie majeure non-productive, dans laquelle la protéine est transloquée dans la lumière du réticulum endoplasmique (RE) et suivrait la voie de sécrétion où elle est glycosylée et clivée. Cette voie conduit à la production des formes non infectieuses ORF2g et ORF2c, qui sont potentiellement des leurres immunologiques ; (2) une voie productive pour laquelle une fraction de la protéine ORF2, nommée ORF2i, resterait du côté cytosolique des compartiments membranaires de la voie de sécrétion, pour ensuite rejoindre le site d’assemblage des particules virales (ce site n’étant pas défini à ce jour). ORF3, par son activité de viroporine et en interagissant avec différents facteurs cellulaires, favoriserait les étapes tardives du cycle infectieux et la sécrétion des particules virales [30]. Les virions néoformés seraient alors libérés dans le milieu extracellulaire par un mécanisme qui reste à définir mais qui ferait intervenir ORF3 et la voie de sécrétion des exosomes [20, 24, 31-33]. Seules des eHEV sont produites en système de culture cellulaire ou présentes dans le sérum de patients infectés [9]. Il semble donc qu’il puisse exister, dans le tractus intestinal, un processus de délipidation par les acides biliaires et des protéases digestives, plutôt qu’une production naturelle de particules nues.
 | Figure 2. Cycle infectieux du HEV. 1. Le HEV initie son cycle en interagissant de manière peu spécifique avec des protéoglycanes à héparane sulfate (HSPG) présents à la surface de la cellule cible, avant d’interagir avec un ou plusieurs récepteurs spécifiques non encore identifiés. 2. Les particules virales seraient ensuite internalisées dans la cellule par endocytose. 3. Dans le cytoplasme, la dissociation de la capside virale permettrait la libération du génome viral. 4. La protéine ORF1, contenant la réplicase virale, serait immédiatement synthétisée par la machinerie de traduction cellulaire. 5. La présence de la réplicase virale conduirait à la synthèse d’ARN simple brin de polarité négative (-) à partir de l’ARN génomique de polarité positive (+). Ces ARN(-) serviraient de matrice pour la synthèse de nouveaux ARN(+) sous-génomiques d’environ 2,2 kb (6) et génomiques d’environ 7,2 kb (7). 8. Les protéines ORF2 et ORF3 seraient ensuite traduites à partir des ARN sous-génomiques. La protéine ORF2 suivrait deux voies : une voie productive (9) dans laquelle cette protéine s’assemble avec l’ARN(+) génomique pour former des virions ; ces virions (10), présents au niveau des corps multi-vésiculaires (MVB, multivesicular bodies) seraient ensuite enveloppés d’une bicouche lipidique (11) dans laquelle la protéine ORF3 viendrait s’insérer. 12. Les virions seraient ensuite sécrétés grâce à l’activité viroporine de l’ORF3. 13. Dans la voie non productive, la grande majorité de la protéine ORF2 est transloquée dans la lumière du réticulum endoplasmique (RE) pour suivre la voie de sécrétion classique des protéines. Cette voie utilisée par ORF2 n’aboutit pas à la formation de particules virales. |
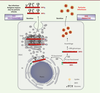 | Figure 3. Modèle des voies de production de la protéine de capside ORF2. La protéine ORF2 pourrait suivre deux voies différentes après sa synthèse. 1. Une voie productive minoritaire dans laquelle une fraction de la protéine ORF2 possède un peptide signal (PS) non fonctionnel et resterait localisée du coté cytosolique. Elle sert alors à encapsider le génome viral pour former des virions qui sont ensuite sécrétés. Cette protéine ORF2 associée aux particules virales est appelée ORF2i. 2. Une voie non productive majoritaire dans laquelle une fraction importante de la protéine ORF2 (ORF2g) possédant un PS fonctionnel est transloquée dans la lumière du RE pour suivre la voie de sécrétion classique des protéines. Elle y subit des modifications post-traductionnelles telles que la N-glycosylation, la O-glycosylation et la sialylation. La protéine ORF2g est ensuite partiellement clivée pour produire l’ORF2c. Les protéines ORF2g et ORF2c ne forment pas de particules virales et sont sécrétées en très grandes quantités dans le sérum des patients infectés par le HEV. ORF2g : ORF2 glycosylée ; ORF2c : ORF2 clivée ; ORF2i : ORF2 infectieuse/intracellulaire ; ERGIC : ER-Golgi intermediate compartment. |
|
Les modèles d’étude du HEV En raison de l’absence, pendant des années, d’un système efficace de culture cellulaire pour produire le HEV, différents outils permettant l’étude des protéines virales ou de certaines étapes du cycle infectieux ont été développés. Des systèmes d’expression hétérologue ont été particulièrement utilisés. Notamment, la protéine de capside ORF2 a été essentiellement étudiée en exprimant des formes tronquées dans des cellules d’insectes ou dans la bactérie Escherichia coli. Ces études ont permis de réaliser des analyses structurales et fonctionnelles de la capside virale. Des réplicons, des unités autonomes de réplication pour lesquelles les ORF2 et 3 sont remplacés par le gène codant une protéine rapporteuse, telle que la GFP (green fluorescent protein) ou la luciférase, sont également particulièrement utiles pour l’analyse de la réplication du HEV [34]. Des anticorps spécifiques des trois protéines virales ont également été produits. Les anticorps anti-ORF2 et anti-ORF3 permettent ainsi de détecter ces protéines en système d’expression hétérologue et infectieux. En revanche, les anticorps anti-ORF1 ne permettent de détecter ORF1, ou ses différents domaines, que lorsque la protéine est surexprimée via un vecteur d’expression plasmidique [35]. Aucun anticorps détectant ORF1 en système réplicatif ou infectieux n’est disponible à ce jour. Bien que plusieurs tentatives d’infection aient été initialement réalisées dans diverses lignées cellulaires, l’amplification du HEV en culture cellulaire s’est avérée très difficile (pour revue [36]). Le succès de l’amplification du HEV a été obtenu avec l’adaptation de souches de génotypes 3 et 4 à la culture cellulaire, les génotypes 1 et 2 restant difficiles voire impossibles à amplifier. En effet, une souche de génotype 3, puis une souche de génotype 4, ont été adaptées par Tanaka et al. à des cultures de cellules d’hépatome humain (PLC/PRF/5) et d’adénocarcinome pulmonaire (A549), et les mutations nécessaires à l’adaptation du virus ont été définies. En 2011, la souche Kernow-C1, de génotype 3, provenant d’un patient chroniquement co-infecté par le VIH (virus de l’immunodéficience humaine) et le HEV a été identifiée pour sa capacité à infecter des lignées cellulaires d’origine humaine, porcine ou de cerf, après six passages en série (Kernow-C1 p6). La capacité accrue de la souche Kernow-C1 p6 à se répliquer in vitro a pour origine une insertion de 57 aa provenant de la protéine ribosomale humaine S17 au sein du domaine HVR de l’ORF1 du virus. Cette souche recombinante (Kernow-C1 p6) contenant la S17 était présente dans l’échantillon fécal originel du patient. Après six passages en série sur des cellules d’hépatome humain (HepG2/C3A), elle est devenue dominante grâce à sa capacité de propagation plus efficace que les autres souches virales présentes dans l’échantillon. L’insertion de la S17 dans une souche de génotype 1 (Sar55) augmente également de façon significative la capacité de ce génotype à être transfecté dans des cellules de hamster BHK-21. Une autre souche de génotype 3 (LBPR-0379) a été identifiée pour son avantage de croissance en culture cellulaire ; elle présente une insertion de la protéine ribosomale S19 dans le domaine HVR de ORF1. Les mécanismes par lesquels ces insertions améliorent la réplication du HEV in vitro et élargissent le spectre d’hôtes du virus sont inconnus et sont d’un intérêt majeur. Ils démontrent la capacité du virus à muter en quasi-espèces et d’acquérir de nouvelles capacités au cours d’une infection chronique. Néanmoins, bien que ces systèmes aient permis de mieux appréhender le cycle infectieux du HEV, ils ne permettent de produire que des titres infectieux faibles, limitant les analyses biochimiques directes des protéines virales et des particules infectieuses. Très récemment, un système utilisant la souche Kernow-C1 p6 et les cellules PLC3, un sous-clone de la lignée PLC/PRF/5, a été décrit [9]. Dans ce système, la protéine ORF2 est produite de manière très précoce et en grandes quantités, et des particules infectieuses sont produites rapidement, dès 4 jours après la transfection. L’amplification puis la purification des particules sur gradient ou coussin d’iodixanol ont permis d’obtenir des titres viraux importants (1 à 5 x 106 FFU[focus forming units]/ml) et de déterminer l’ultrastructure des particules virales. C’est ainsi qu’il a pu être montré que les particules produites en culture cellulaire (HEVcc) présentaient une morphologie similaire à celle des particules isolées à partir de patients infectés [9]. Des essais d’infection d’hépatocytes primaires, humains ou de macaques, ont également été réalisés avec succès [37-39]. Des hépatocytes issus de cellules souches pluripotentes constituent également un bon modèle d’étude du HEV, notamment pour des souches virales primaires non adaptées à la culture cellulaire. Ces cellules semblent physiologiquement plus appropriées pour l’étude de souches primaires et pour l’analyse de la réponse immunitaire et des voies de signalisation induites par l’infection HEV [40, 41]. La compréhension des mécanismes de la pathogenèse du HEV nécessite l’utilisation de modèles animaux. L’infection par HEV est généralement asymptomatique chez la plupart des hôtes zoonotiques qui sont de ce fait peu utilisables en laboratoire. Les primates non humains, comme les singes macaques cynomolgus et rhésus, ont été utilisés avec succès pour étudier la réponse immunitaire à l’infection. Ces modèles sont cependant coûteux et leur utilisation pose des problèmes éthiques. Des modèles de souris au foie humanisé ont récemment été développés [30]. Ceux-ci peuvent se révéler intéressants pour étudier certains aspects de l’infection virale. Cependant, ces souris sont immuno-incompétentes et la reconstitution d’un système immunitaire actif serait nécessaire afin d’évaluer le rôle de la réponse immunitaire dans la pathogenèse de l’infection [44] (→). (→) Voir la Nouvelle de H. Gilgenkrantz, m/s n° 10, octobre 2016, page 812 De nombreux outils ont donc été récemment développés pour étudier le HEV. Ils posent ainsi les jalons qui permettront de mieux comprendre le cycle infectieux et la pathogénicité du virus. Des avancées notables ont été réalisées dans la caractérisation des protéines du HEV, notamment de sa protéine de capside ORF2. Cependant, l’abondante sécrétion de cette protéine sous une forme non associée à du matériel infectieux interroge quant à son rôle potentiel dans le contrôle de la réponse immunitaire. Certaines étapes clés du cycle infectieux du virus restent également à caractériser, notamment les facteurs cellulaires impliqués dans son entrée. Finalement, l’identification de formes chroniques chez les personnes immunodéprimées devrait conduire à évaluer les effecteurs de la réponse immunitaire impliqués dans le contrôle de cette infection virale. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article. |
Nous remercions Sophana Ung pour son aide technique. Maliki Ankavay est allocataire doctoral de l’Agence Nationale de Recherches sur le Sida et les Hépatites virales (ANRS).
|
Footnotes |
1.
Aspinall EJ, Couturier E, Faber M, et al. Hepatitis E virus infection in Europe: surveillance and descriptive epidemiology of confirmed cases, 2005 to 2015 . Euro Surveill. 2017 ; :22.. 2.
Doceul V, Bagdassarian E, Demange A, et al. Zoonotic Hepatitis E Virus: Classification, animal reservoirs and transmission routes . Viruses. 2016; ; 8 : :270.. 3.
Pavio N, Merbah T, Thébault A. Frequent hepatitis E virus contamination in food containing raw pork liver . France. Emerging Infect Dis. 2014; ; 20 : :1925.–1927. 4.
Barnaud E, Rogée S, Garry P, et al. Thermal inactivation of infectious hepatitis E virus in experimentally contaminated food . Appl Environ Microbiol. 2012; ; 78 : :5153.–5159. 5.
Kamar N, Izopet J, Pavio N, et al. Hepatitis E virus infection . Nat Rev Dis Primers. 2017; ; 3 : :17086.. 6.
Pischke S, Hartl J, Pas SD, et al. Hepatitis E virus: Infection beyond the liver? . J Hepatol. 2017; ; 66 : :1082.–1095. 7.
Purdy MA, Harrison TJ, Jameel S, et al. ICTV virus taxonomy profile: Hepeviridae . J Gen Virol. 2017; ; 98 : :2645.–2646. 8.
Bradley DW, Krawczynski K, Cook EH, et al. Enterically transmitted non-A, non-B hepatitis: serial passage of disease in cynomolgus macaques and tamarins and recovery of disease-associated 27- to 34-nm virus-like particles . Proc Natl Acad Sci USA. 1987; ; 84 : :6277.–6281. 9.
Montpellier C, Wychowski C, Sayed IM, et al. Hepatitis E Virus lifecycle and identification of 3 forms of the ORF2 capsid protein . Gastroenterology. 2018; ; 154 : :211.–23e8. 10. Parvez MK. The hepatitis E virus nonstructural polyprotein . Future Microbiology. 2017; fmb-2017-0016.. 11.
Kanade GD, Pingale KD, Karpe YA. Activities of thrombin and factor Xa are essential for replication of Hepatitis E Virus and are possibly implicated in ORF1 polyprotein processing . J Virol. 2018; ; 92 : :17.–15. 12.
Guu TSY, Liu Z, Ye Q, et al. Structure of the hepatitis E virus-like particle suggests mechanisms for virus assembly and receptor binding . Proc Natl Acad Sci USA. 2009; ; 106 : :12992.–12997. 13.
Yamashita T, Mori Y, Miyazaki N, et al. Biological and immunological characteristics of hepatitis E virus-like particles based on the crystal structure . Proc Natl Acad Sci USA. 2009; ; 106 : :12986.–12991. 14.
Xing L, Li TC, Mayazaki N, et al. Structure of hepatitis E virion-sized particle reveals an RNA-dependent viral assembly pathway . J Biol Chem. 2010; ; 285 : :33175.–33183. 15.
Tang X, Yang C, Gu Y, et al. Structural basis for the neutralization and genotype specificity of hepatitis E virus . Proc Natl Acad Sci USA. 2011; ; 108 : :10266.–10271. 16.
Yin X, Ying D, Lhomme S, et al. Origin, antigenicity, and function of a secreted form of ORF2 in hepatitis E virus infection . Proc Natl Acad Sci USA. 2018; ; 3 : :21345.–21312. 17.
Takahashi M, Yamada K, Hoshino Y, et al. Monoclonal antibodies raised against the ORF3 protein of hepatitis E virus (HEV) can capture HEV particles in culture supernatant and serum but not those in feces . Arch Virol. 2008; ; 153 : :1703.–1713. 18.
Ding Q, Heller B, Capuccino JMV, et al. Hepatitis E virus ORF3 is a functional ion channel required for release of infectious particles . Proc Natl Acad Sci U S A. 2017; ; 114 : :1147.–1152. 19.
Surjit M, Oberoi R, Kumar R, et al. Enhanced 1 microglobulin secretion from Hepatitis E Virus ORF3-expressing human hepatoma cells is mediated by the tumor susceptibility gene 101 . J Biol Chem. 2006; ; 281 : :8135.–8142. 20.
Nagashima S, Takahashi M Jirintai, et al. A PSAP motif in the ORF3 protein of hepatitis E virus is necessary for virion release from infected cells . J Gen Virol. 2011; ; 92 : :269.–278. 21.
Votteler J, Sundquist WI. Virus Budding and the ESCRT Pathway . Cell Host Microbe. 2013; ; 14 : :232.–241. 22.
Feng Z, Hensley L, McKnight KL, et al. A pathogenic picornavirus acquires an envelope by hijacking cellular membranes . Nature. 2013; ; 496 : :367.–371. 23.
Yin X, Ambardekar C, Lu Y, et al. Distinct entry mechanisms for non-enveloped and quasi-enveloped hepatitis E virus . J Virol. 2016; ; 90 : :4232.–4242. 24.
Yamada K, Takahashi M, Hoshino Y, et al. ORF3 protein of hepatitis E virus is essential for virion release from infected cells . J Gen Virol. 2009; ; 90 : :1880.–1891. 25.
Shukla P, Nguyen HT, Faulk K, et al. Adaptation of a genotype 3 hepatitis E virus to efficient growth in cell culture depends on an inserted human gene segment acquired by recombination . J Virol. 2012; ; 86 : :5697.–5707. 26.
Feng Z, Lemon SM. Peek-a-boo: membrane hijacking and the pathogenesis of viral hepatitis . Trends in Microbiol. 2014; ; 22 : :59.–64. 27.
Kalia M, Chandra V, Rahman SA, et al. Heparan sulfate proteoglycans are required for cellular binding of the Hepatitis E Virus ORF2 capsid protein and for viral infection . J Virol. 2009; ; 83 : :12714.–12724. 28.
Kapur N, Thakral D, Durgapal H, et al. Hepatitis E virus enters liver cells through receptor-dependent clathrin-mediated endocytosis . J Viral Hepat. 2011; ; 19 : :436.–448. 29.
Holla P, Ahmad I, Ahmed Z, et al. Hepatitis E Virus enters liver cells through a Dynamin-2 . Clathrin and membrane cholesterol-dependent pathway. Traffic. 2015; ; 16 : :398.–416. 30.
Nimgaonkar I, Ding Q, Schwartz RE, et al. Hepatitis E virus: advances and challenges . Nat Rev Gastroenterol Hepatol. 2018; ; 15 : :96.–110. 31.
Nagashima S, Jirintai S, Takahashi M, et al. Hepatitis E virus egress depends on the exosomal pathway, with secretory exosomes derived from multivesicular bodies . J Gen Virol. 2014; ; 95 : :2166.–2175. 32.
Nagashima S, Takahashi M, Jirintai S, et al. Tumour susceptibility gene 101 and the vacuolar protein sorting pathway are required for the release of hepatitis E virions . J Gen Virol. 2011; ; 92 : :2838.–2848. 33.
Emerson SU, Nguyen HT, Torian U, et al. Release of genotype 1 hepatitis E virus from cultured hepatoma and polarized intestinal cells depends on open reading frame 3 protein and requires an intact PXXP motif . J Virol. 2010; ; 84 : :9059.–9069. 34.
Emerson SU, Nguyen H, Graff J, et al. In vitro replication of hepatitis E virus (HEV) genomes and of an HEV replicon expressing green fluorescent protein . J Virol. 2004; ; 78 : :4838.–4846. 35.
Lenggenhager D, Gouttenoire J, Malehmir M, et al. Visualization of hepatitis E virus RNA and proteins in the human liver . J Hepatol. 2017; ; 67 : :471.–479. 36.
Okamoto H.. Culture systems for hepatitis E virus . J Gastroenterol. 2012; ; 48 : :147.–158. 37.
Tam AW, White R, Yarbough PO, et al. In vitro infection and replication of Hepatitis E Virus in primary cynomolgus macaque hepatocytes . Virology. 1997; ; 238 : :94.–102. 38.
Oshiro Y, Yasue H, Takahashi K, et al. Mode of swine hepatitis E virus infection and replication in primary human hepatocytes . J Gen Virol. 2014; ; 95 : :2677.–2682. 39.
Yin X, Li X, Ambardekar C, et al. Hepatitis E virus persists in the presence of a type III interferon response . PLoS Pathog. 2017; ; 13 : :e1006417.. 40.
Wu X, Thi VLD, Liu P, et al. Pan-genotype Hepatitis E Virus replication in stem cell-derived hepatocellular systems . Gastroenterology. 2018; ; 154 : :663.–674. 41.
Helsen N, Debing Y, Paeshuyse J, et al. Stem cell-derived hepatocytes: A novel model for hepatitis E virus replication . J Hepatol. 2016; ; 64 : :565.–573. 42.
Izopet J, Kamar N. Hépatite E. De la transmission zoonotique du virus à l’évolution chronique de l’infection chez l’immunodéprimé . Med Sci (Paris). 2008; ; 24 : :1023.–1025. 43.
Le Lay S. Carmen Martinez M, Andriantsitohaina R. Vésicules extracellulaires, biomarqueurs et bioeffecteurs du syndrome métabolique . Med Sci (Paris). 2018; ; 34 : :936.–943. 44.
Gilgenkrantz H, Gouttenoire J, Mallet V. Un modèle murin pour une infection chronique méconnue : l’hépatite E ! . Med Sci (Paris). 2016; ; 32 : :812.–814. |