| |
| Med Sci (Paris). 34(10): 778–781. doi: 10.1051/medsci/2018214.Dynamique de la chromatine en réponse aux dommages de l’ADN Une histoire multiéchelle Judith Miné-Hattab1,2* and Xavier Darzacq3 1Institut Curie, PSL research university, CNRS, UMR3664, 75005Paris, France 2Sorbonne Université, Institut Curie, CNRS, UMR3664, F-75005Paris, France 3Division of genetics, genomics and development, Department of molecular and cell biology, University of California, Berkeley, Berkeley, CA 947201États-Unis MeSH keywords: Chromatine, Assemblage et désassemblage de la chromatine, Cassures double-brin de l'ADN, Altération de l'ADN, Réparation de l'ADN, Histone, Humains, physiologie, métabolisme |
Dynamique de la chromatine en réponse aux cassures double brins L’information génétique contenue à l’intérieur de nos cellules est codée sous forme d’une molécule : l’ADN. Longue d’environ 2 mètres, celle-ci est compactée dans un noyau d’environ 10 mm de diamètre, soit 200 000 fois plus petit. L’organisation et la dynamique de l’ADN jouent un rôle essentiel dans de nombreux processus biologiques et se trouvent perturbées dans des cellules de tissus malades. Afin de mieux comprendre la dynamique de l’ADN à l’intérieur d’une cellule vivante, en l’occurrence une cellule de bactérie, l’une des approches consiste à suivre, par microscopie de fluorescence le mouvement d’une région spécifique d’ADN appelée « locus ». Pour observer la dynamique d’un locus particulier dans une cellule vivante, il est nécessaire de mettre en place une stratégie d’ingénierie génomique visant à créer un domaine d’ADN visualisable par microscopie. Pour cela, des opérateurs lacO (ou tetO)1 sont introduits au locus d’intérêt, auxquels vont se lier leurs partenaires protéiques LacI (ou TetR) marqués par des fluorophores. Le site d’intérêt est ainsi visible sous la forme d’un point fluorescent en microscopie champ large. En suivant la position de ce point au cours du temps, il est ainsi possible de déterminer la trajectoire suivie par ce locus, et d’en déduire l’espace qu’il explore au sein du noyau. Il a ainsi été montré que la chromatine diffuse différemment selon le niveau de différenciation cellulaire, le cycle cellulaire [1], le niveau d’activité transcriptionnelle, etc. Au cours de la vie d’une cellule, notre génome subit de nombreuses cassures dont les plus génotoxiques sont les cassures double brin [2]. Comment la dynamique de la chromatine est-elle affectée en réponse aux dommages de l’ADN ? Dans une étude précédente [3, 4] nous avions suivi le mouvement de deux loci génomiques homologues en l’absence et en présence de cassure(s) double-brin dans des cellules diploïdes de levure Saccharomyces cerevisiae. Nous avions alors observé une augmentation importante de la dynamique de la chromatine en réponse à une ou plusieurs cassures double brin. Le site endommagé s’est révélé être le plus mobile, le volume nucléaire exploré par ce locus étant dix fois supérieur en présence de dommage. Cependant, la réponse cellulaire déclenchée par une cassure double brin ne se limite pas au site endommagé. En effet, la dynamique de l’ensemble du génome augmente également, et cela de façon proportionnelle au nombre de cassures double brin induites dans un noyau [3]. À la suite de dommages de l’ADN, la cellule déclenche donc une modification de la dynamique chromatinienne qui se traduit par une hyper-mobilité locale très importante, ainsi que par une hyper-mobilité globale plus modérée affectant l’ensemble du génome. Les protéines impliquées dans la recombinaison homologue, Mec12,, Rad93 [5], Rad514, et Sae25, [3], sont nécessaires pour promouvoir l’hyper-mobilité de la chromatine en réponse aux cassures double brin. L’augmentation de mobilité locale et globale de la chromatine semble favoriser l’appariement entre chromosomes homologues, celui-ci étant dix fois plus fréquent après l’induction d’une cassure double-brin [3]. De nombreuses études (voir la revue [6]), ont également montré que le changement de mobilité observé chez la levure est un mécanisme conservé à travers les organismes [7, 8, 9]. Chez les mammifères, l’augmentation de mobilité à la suite de dommages de l’ADN n’est cependant pas observée dans toutes les études, ces variations étant probablement la conséquence du type et du nombre de cassures, de la région chromatinienne endommagée, et de la voie de signalisation utilisée pour réparer ces cassures. (→) (→) Voir la Nouvelle de J. Miné-Hattab et R. Rothstein, m/s n° 8-9 août-septembre 2012, page 714 En réponse aux cassures double brin, le coefficient de diffusion de la chromatine reste inchangé [3, 5] : le site endommagé explore un espace nucléaire plus large sans pour cela augmenter sa vitesse de déplacement. Afin de mieux comprendre les modifications affectant la chromatine à la suite des dommages de l’ADN, nous avons récemment étudié la dynamique de la chromatine à différentes échelles spatio-temporelles [10]. |
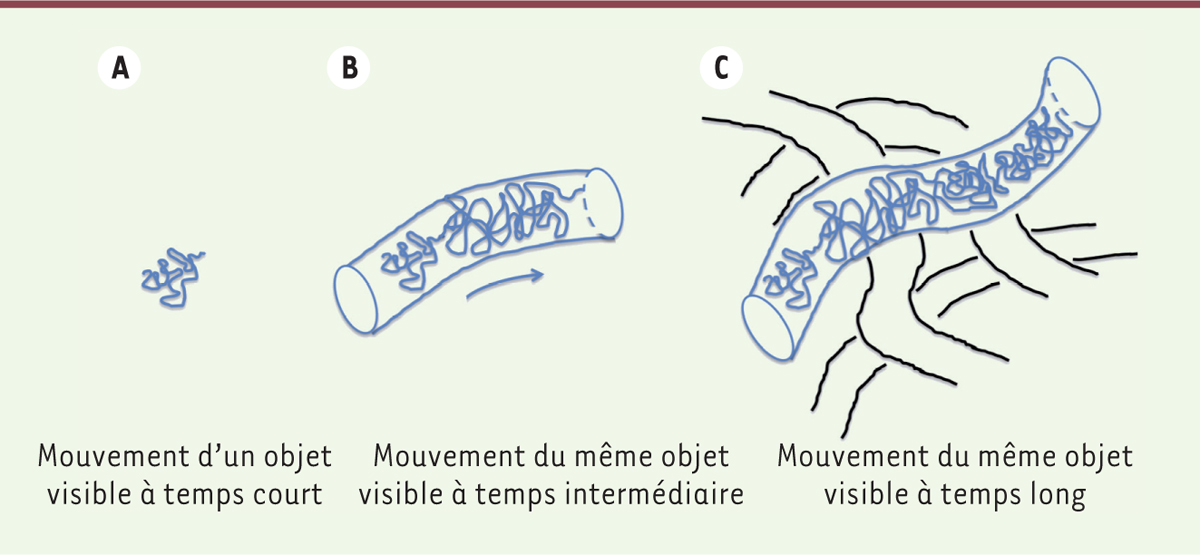
« Suivi » multi-échelles de la chromatine La dynamique de la chromatine peut être mesurée selon différentes échelles de temps variant de quelques secondes à quelques minutes [11]. Lorsque l’on étudie la diffusion d’un objet, l’échelle de temps à laquelle les mesures sont collectées révèle la nature de la diffusion uniquement à cette échelle spatio-temporelle précise. Jusqu’à présent, aucun consensus n’avait été atteint sur la nature exacte du mouvement de la chromatine dans les cellules, sans doute parce que chaque étude se restreignait à une échelle de temps particulière en décrivant un processus de diffusion potentiellement différent à une autre échelle temporelle. De la même façon que la chromatine présente une organisation multi-échelle, on peut imaginer que la chromatine diffuse différemment selon l’échelle spatio-temporelle examinée. Dans ce cas, le mouvement de la chromatine serait décrit par un mouvement complexe résultant de la superposition de différents régimes de diffusion. Par exemple, un locus peut évoluer avec un coefficient de diffusion anormale Amicro dans une région de l’espace qui diffuse elle-même avec un coefficient de diffusion Amacro. L’ADN d’une cellule forme des domaines à plusieurs échelles imbriqués les uns dans les autres, et il est donc concevable qu’un grand domaine, lui-même mobile, contienne un sous-domaine qui en plus de la mobilité qui lui est imposée par le grand domaine, diffuse au sein de ce dernier et ainsi se déplace à plusieurs échelles temporelles et spatiales. Un mouvement complexe peut également résulter de l’alternance d’une diffusion libre et d’une diffusion dirigée, par exemple si l’objet étudié s’accroche par intermittence à un filament d’actine ou de microtubules. La Figure 1, inspirée du modèle de reptation développé par Pierre-Gilles de Gennes (1932-2007)6 [12], illustre un mouvement complexe qui présente différents régimes de diffusion selon l’échelle temporelle examinée.
 | Figure 1. Modèle de reptation appliqué à la chromatine. Illustration tirée du modèle de reptation de P.-G. de Gennes. À différentes échelles temporelles (A, B, C) correspondent différents régimes de diffusion. |
Jusqu’à présent, la dynamique de la chromatine a été étudiée principalement à des échelles de temps relativement grandes : par exemple, la position d’un site lacO-LacI est mesurée toutes les 10 secondes. Cet intervalle de temps correspond au temps nécessaire au microscope pour mesurer la position d’un spot en 3 dimensions ; la position du spot est alors moyennée pendant le temps de l’acquisition, mais le mouvement détaillé de la chromatine pendant cet intervalle de temps reste inconnu. Avec l’arrivée de microscopes plus performants, il est maintenant possible de mesurer la position d’un spot à une vitesse 1 000 fois plus importante (toutes les millisecondes). Afin de mettre en évidence l’existence de différents régimes de diffusion, nous avons mesuré la dynamique de la chromatine à 4 échelles de temps différentes, avant et après induction de dommages de l’ADN (une mesure toutes les 10 secondes [3], toutes les secondes, toutes les 100 ms et même toutes les 10 ms [10]). |
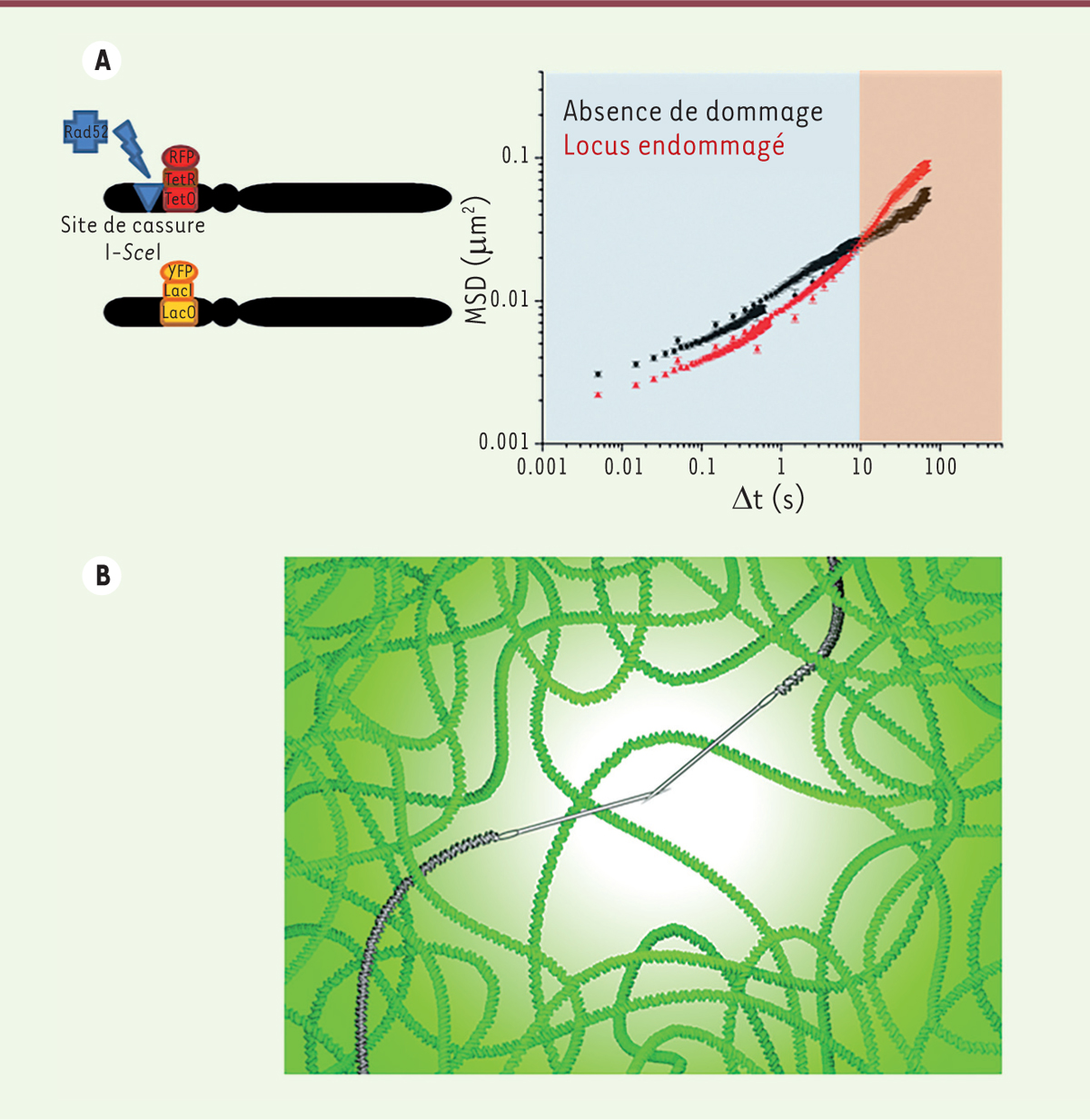
Le suivi multi-échelle révèle le mouvement complexe de la chromatine en réponse aux dommages de l’ADN En suivant le mouvement de la chromatine à différentes échelles temporelles, nous avons montré que la dynamique de la chromatine est plus complexe que nous ne l’anticipions [10]. À la suite de dommages de l’ADN, la chromatine est plus mobile lorsqu’elle est observée à des intervalles de temps de l’ordre de la seconde. Cependant, de manière surprenante, elle est moins mobile lorsqu’elle est examinée à des intervalles de temps très courts (une mesure au maximum toutes les 100 millisecondes). Cela se traduit par un croisement des courbes de déplacements carrés moyens (mean square displacement ou MSD) traduisant le mode de diffusion d’un locus (Figure 2A). En d’autres termes, l’augmentation de mobilité décrite précédemment dans de nombreuses études [3, 5] se révèle n’être en fait que la partie apparente, visible à des temps longs, des modifications affectant la chromatine après des dommages de l’ADN. De manière intéressante, l’augmentation de mobilité à des temps longs ainsi que la réduction de mobilité à des temps courts sont toutes deux observées à la fois au site de cassure et dans l’ensemble du génome. Les changements de mobilité de la chromatine observés aux différentes échelles de temps sont donc à la fois locaux et globaux [10]. La protéine Rad51, qui joue un rôle clé dans le processus de recombinaison homologue, est essentielle pour les changements de mobilité observés à toutes les échelles de temps.
 | Figure 2. Analyse de la mobilité de la chromatine à différentes échelles de temps. A. À gauche : schéma de la souche utilisée pour décrire la dynamique de la chromatine au locus où la cassure est provoquée. Des opérateurs lacO (ou tetO) sont introduits au locus d’intérêt, auxquels vont se lier leurs partenaires de liaison LacI (ou TetR) marqués par des fluorophores. RFP (red fluorescent protein) et YFP (yellow fluorescent protein) sont des protéines naturellement fluorescentes, respectivement rouge et jaune. Les protéines impliquées dans la recombinaison homologue, dont Rad51, sont recrutées au site de cassure de l’ADN, le site de reconnaissance de l’endonucléase I-SceI. À droite : carrés moyens des déplacements (MSD) représentés sur une échelle log-log, en l’absence de cassure (courbe noire) et en présence d’une cassure unique au site de cassure de l’endonucléase I-Scel (courbe rouge). B. Modèle de la pelote de laine et de l’aiguille proposé pour illustrer la dynamique multi-échelle de la chromatine en réponse aux dommages de l’ADN. |
|
Dynamique multi-échelle et lien avec la rigidité de la chromatine Afin d’interpréter nos résultats expérimentaux, nous avons comparé les profils de MSD obtenus (traduisant la mobilité du locus observé) (Figure 2A) à des simulations numériques de la dynamique de polymères [13]. Il a été montré que lorsqu’un polymère devient plus rigide (dans sa globalité) sa mobilité est réduite à petite échelle de temps alors qu’elle augmente à grande échelle de temps [13]. Ce résultat, comparé à nos observations expérimentales, suggère donc que la chromatine devient globalement plus rigide en réponse aux dommages de l’ADN. Les changements de profils de MSD étant plus importants au niveau du locus endommagé que dans le reste du génome, nous avons proposé que l’augmentation de rigidité devait être plus importante à ce niveau, même si elle affecte également l’ensemble du génome dans une moindre mesure. Nos résultats sont concordants avec une étude récemment réalisée avec des méthodes différentes sur des cellules de levure haploïdes [14]. Nous avons proposé un modèle dans lequel la chromatine peut être comparée à une pelote de laine : en réponse aux dommages de l’ADN, les extrémités endommagées sont recouvertes de protéines de recombinaison homologue, symbolisées dans ce modèle par une aiguille (Rad51 étant connu pour rigidifier l’ADN in vitro). En réponse aux dommages, la chromatine deviendrait globalement plus rigide, cet effet étant plus important au niveau de la cassure. À intervalles de temps courts, l’aiguille est peu mobile car très rigide ; au contraire, à intervalles de temps longs, l’aiguille pourrait traverser les mailles de laine plus facilement et deviendrait plus mobile qu’avant la cassure de l’ADN. De façon générale, une dynamique multi-échelle est-t-elle un mécanisme favorisant la cinétique de recherche de cibles ? Dans de futurs travaux, il serait intéressant d’examiner si d’autres mécanismes biologiques font intervenir de tels mouvements résultant de la superposition de différents types de diffusion. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données pubiées dans cet article. |
Footnotes |
1. Heun P, Laroche T, Shimada K, et al. Chromosome dynamics in the yeast interphase nucleus . Science. 2001; ; 294 : :2181.–2186. 2. Sung P. Introduction to the thematic minireview series: DNA double-strand break repair and pathway choice . J Biol Chem. 2018 ; doi: 10.1074/jbc.TM118.003212. 3. Miné-Hattab J, , Rothstein R. Increased chromosome mobility facilitates homology search during recombination . Nat Cell Biol. 2012; ; 14 : :510.–517. 4. Miné-Hattab J, , Rothstein R. Réparation de l‘ADN. Comment trouver le bon partenaire ? . Med Sci (Paris). 2012; ; 28 : :714.–716. 5. Dion V, Kalck V, Horigome C, et al. Increased mobility of double-strand breaks requires Mec1, Rad9 and the homologous recombination machinery . Nat Cell Biol. 2012; ; 14 : :502.–509. 6. Miné-Hattab J, , Rothstein R. DNA in motion during double-strand break repair . Trends Cell Biol. 2013; ; 23 : :529.–536. 7. Aten JA, Stap J, Krawczyk PM, et al. Dynamics of DNA double-strand breaks revealed by clustering of damaged chromosome domains . Science. 2004; ; 303 : :92.–95. 8. Chiolo I, Minoda A, Colmenares SU, et al. Double-strand breaks in heterochromatin move outside of a dynamic HP1a domain to complete recombinational repair . Cell. 2011; ; 144 : :732.–744. 9. Krawczyk PM, Borovski T, Stap J, et al. Chromatin mobility is increased at sites of DNA double-strand breaks . J Cell Sci. 2012; ; 125 : :2127.–2133. 10. Miné-Hattab J, Recamier V, Izeddin I, et al. Multi-scale tracking reveals scale-dependent chromatin dynamics after DNA damage . Mol Biol Cell. 2018 : doi: 10.1091/mbc.E17-05-0317. 11. Hajjoul H, Mathon J, Ranchon H, et al. High-throughput chromatin motion tracking in living yeast reveals the flexibility of the fiber throughout the genome . Genome Res. 2013; ; 23 : :1829.–1838. 12. De Gennes PG,
Kinetics of diffusioncontrolled processes in dense polymer systems. II.Effects of entanglements . J Chem Phys. 1982; ; 76 : :3322.–3326. 13. Faller R, Müller-Plathe F,
Chain stiffness intensifies the reptation characteristics of polymer dynamics in the melt . Chemphyschem. 2008; ; 2 : :180.–184. 14. Herbert S, Brion A, Arbona JM, et al. Chromatin stiffening underlies enhanced locus mobility after DNA damage in budding yeast . EMBO J. 2018; ; 36 : :2595.–2608. |