| |
| Med Sci (Paris). 34(5): 485–490. doi: 10.1051/medsci/20183405024.Chroniques génomiques - Gènes et longévité : nouvelles données, nouvelles controverses Bertrand Jordan1* 1UMR 7268 ADÉS, Aix-Marseille, Université/EFS/ CNRS ; CoReBio PACA,case 901, Parc scientifique de Luminy, 13288Marseille Cedex 09, France MeSH keywords: Certificats de naissance, Bases de données factuelles, Certificats de décès, Études d'associations génétiques, Génétique des populations, Humains, Longévité, Pedigree, Terminologie comme sujet, Transcriptome, tendances, génétique |

De nombreuses familles comportent parmi leurs membres une personne passionnée par la généalogie, qui consacre ses loisirs à établir le mieux possible l’arbre généalogique familial en remontant aux ascendants les plus anciens et en découvrant l’existence de lointains cousins. Depuis quelques décennies, l’informatique est venue au secours de ces recherches, et il existe aujourd’hui des bases de données très étendues et ouvertes à toute personne intéressée, qui répertorient les résultats de ces travaux et facilitent grandement leur extension. L’un des nombreux sites offrant ce service aux généalogistes, Geni.com (https://www.geni.com/), indique qu’il catalogue 180 millions de profils, dont 120 millions sont reliés entre eux, formant ce que l’entreprise appelle le « Grand Arbre ». Pour chaque individu, la base de données répertorie nom, prénom, dates et lieux de naissance, de décès et éventuellement de mariage, et indique sa relation avec d’autres personnes présentes dans la base. Bien que ces informations soient assez sommaires, elles peuvent être soumises à une analyse informatique susceptible d’apporter des conclusions intéressantes : c’est ce que rapporte un récent article paru dans la revue Science [
1]. Je m’intéresserai ici au résultat le plus médiatisé de ce travail, l’affirmation que l’héritabilité de la longévité humaine est plus faible que la valeur généralement admise jusqu’ici : 0,10 à 0,15 au lieu de 0,25 à 0,30. |
Un immense arbre généalogique Les auteurs sont en fait partis des 86 millions de profils disponibles dans la base de données de Geni lors du début de leur étude. Après différentes étapes de vérification, ils parviennent à définir un arbre généalogique validé qui comporte pas moins de 13 millions d’individus couvrant 11 générations, depuis 1400 jusqu’à nos jours (il s’agit pour l’essentiel de personnes d’origine européenne). La longévité de chaque individu est donnée par ses dates de naissance et de décès, et la longévité moyenne à chaque époque pour cet échantillon correspond aux valeurs historiques connues. Il est alors possible d’évaluer la corrélation entre la longévité des parents et celle des enfants tout au long de la période couverte : si la part génétique de la longévité est importante, on s’attend à une forte corrélation. La
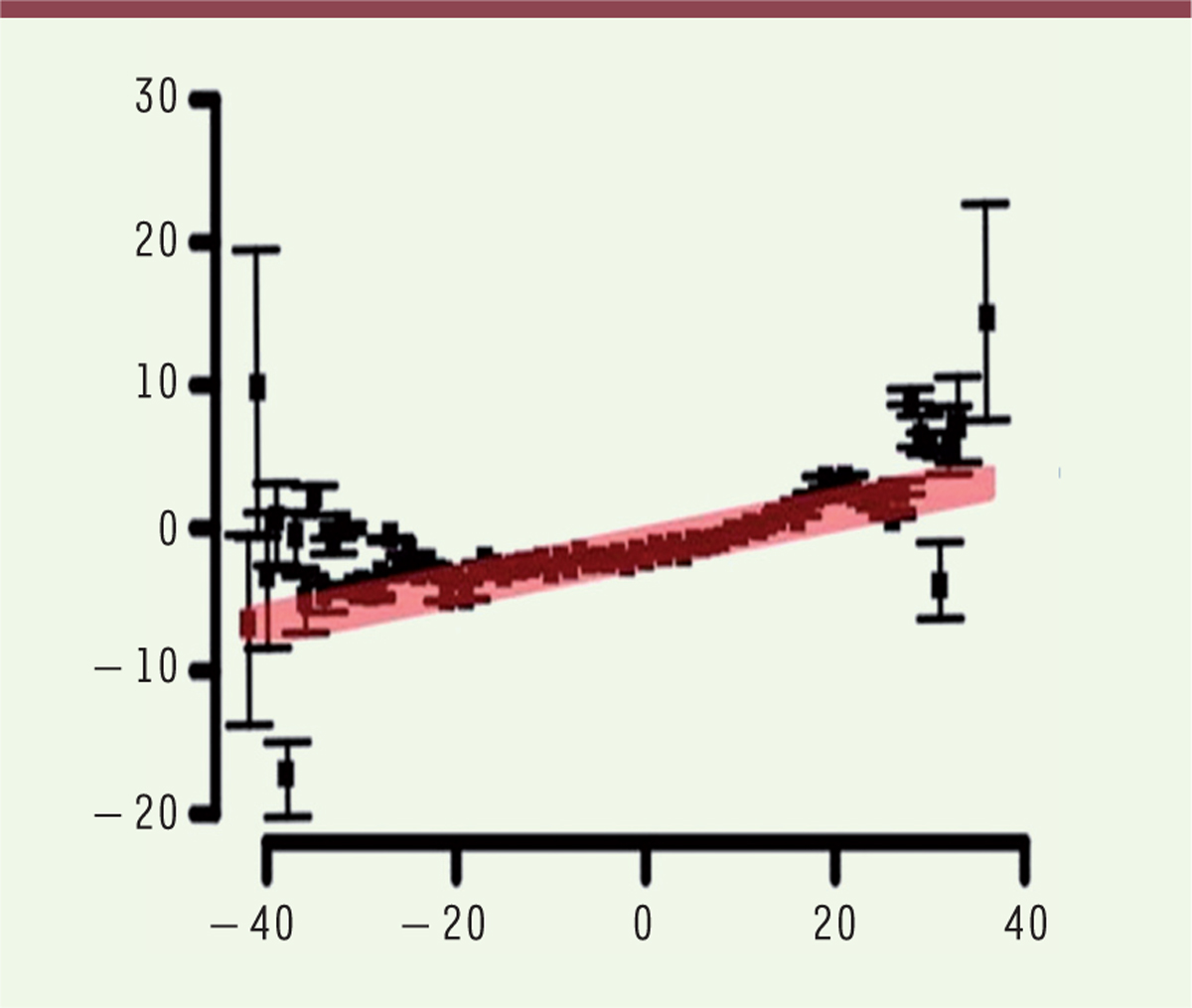
Figure 1
montre qu’il y a bien une corrélation, mais celle-ci est faible, le calcul montre qu’elle indique une héritabilité d’environ 12 %.
 | Figure 1.
Corrélation entre la longévité des parents (ordonnée) et celle de leurs enfants (en abscisse), montrant une corrélation modeste entre ces deux grandeurs (ligne rouge). La longévité est définie ici comme le nombre d’années de vie en plus ou en moins de la valeur moyenne à la même époque. Les barres d’erreur aux extrémités de la distribution sont importantes en raison du faible nombre d’individus vivant beaucoup plus (ou beaucoup moins) d’années que la moyenne (extrait partiel modifié de la figure 3A de [ 1]). |
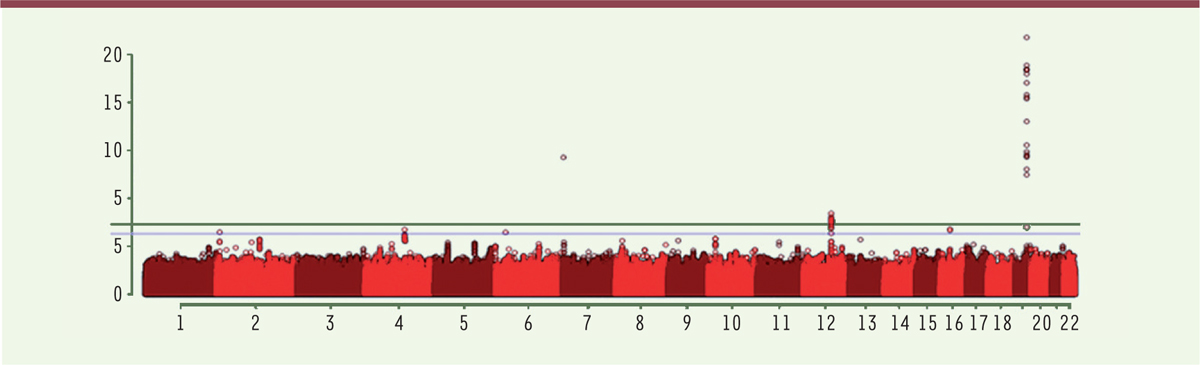
 | Figure 2.
Manhattan plot de l’analyse GWAS pour la longévité extrême. En abscisse, les différents chromosomes, de 1 à 22 (couleurs alternées). En ordonnée, significativité des signaux observés, la ligne inférieure correspond à p = 5 x 10 -7, la supérieure à p = 5 x 10 -8 (limite de significativité « génome entier ») (extrait partiel remanié de la figure 2A de [
4]). |
La valeur trouvée est donc nettement plus faible que celle généralement admise [
2] ; notons, de plus, que cette héritabilité n’est pas nécessairement due uniquement aux gènes : une similitude entre parents et enfants au niveau du mode de vie pourrait induire une certaine corrélation entre leurs espérances de vie respectives. Mais, comme nous allons le voir, la définition même de la longévité est sujette à caution. |
La nécessité d’une définition précise Les déterminants génétiques de la longévité ont fait l’objet de nombreuses études, notamment chez l’homme et la souris (pour une synthèse récente voir [2]) ainsi que chez de nombreuses autres espèces. Comme déjà indiqué, les évaluations de l’héritabilité de la longévité dans notre espèce tournent autour de 0,25 ; des études GWAS (genome-wide association study) récentes ont confirmé l’implication du locus APOE (apolipoprotéine E), l’allèle APOE e2 diminuant le risque de maladies cardiovasculaire et d’Alzheimer. L’autre locus confirmé par plusieurs études est le gène FOXO3 (Forkhead box O3). D’autres gènes ou locus ont été repérés dans diverses études mais ne sont pas encore confirmés [2]. On doit à deux équipes de Boston (États-Unis) qui travaillent depuis longtemps sur ce thème, un intéressant article sur les différentes acceptions du terme de longévité [
3]. Ils montrent en particulier, grâce à l’étude d’un échantillon provenant de la New England Centenarian Study, que les corrélations relatives entre apparentés augmentent avec l’âge ; en d’autres termes, que les effets génétiques sont plus visibles si l’on se limite aux individus les plus âgés. Par exemple, pour des individus nés en 1900, si un membre de la fratrie a vécu jusqu’à 90 ans, la probabilité qu’un autre membre atteigne au moins cet âge est presque doublée (par rapport à la moyenne de la cohorte). S’il a vécu jusqu’à 100 ans, la probabilité pour que frère ou sœur atteigne le même âge est multipliée par neuf ; pour 105 ans, elle est multipliée par 35 [3] ! Bien sûr ces probabilités sont faibles dans l’absolu, mais l’effet est très net, comme si les influences génétiques étaient révélées par le grand âge. En tous cas, cet exemple montre qu’effectivement une définition précise de la longévité est indispensable, ne serait-ce que pour pouvoir comparer les résultats de différentes études. Un article plus récent met en pratique ces recommandations [4]. Les mêmes auteurs, rejoints par d’autres équipes notamment italiennes, ont effectué une étude de type GWAS dans laquelle les « cas » (extrême longévité) étaient des personnes nées en 1900 et ayant vécu au moins jusqu’à 96 ans pour les hommes, 100 ans pour les femmes : ceci correspond aux âges auxquels il reste moins de 1 % de survivants de la cohorte. Ayant réussi, grâce à un consortium de quatre équipes, à rassembler 2 070 participants correspondant à ces critères, ils ont déterminé six millions de polymorphismes (SNP, single nucleotide polymorphism) chez chacun d’eux ainsi que chez 6 259 témoins. La
Figure 2
montre le Manhattan plot
1 obtenu, sur lequel on discerne clairement trois locus significatifs situés sur les chromosomes 7, 12 et 19. Le fort signal observé sur le chromosome 12 correspond au gène APOE déjà mentionné, les deux autres locus détectés sont nouveaux. En revanche, le gène FOXO3 n’est pas retrouvé dans cette étude. Cinq autres associations sont repérables mais n’atteignent pas la limite nécessaire pour être pleinement significative. Les nouveaux locus repérés correspondent, pour le chromosome 7, à un variant « synonyme »2, dans la séquence codante du gène USP42 (ubiquitin specific peptidase 42), qui est deux à trois fois plus fréquent chez les centenaires3, que chez les témoins, mais dont la fonction est inconnue, et, pour le chromosome 12, un variant dans un intron du gène TMTC2 (transmembrane and tetratricopeptide repeat containing 2), variant majoritaire chez tous, mais un peu plus fréquent chez les centenaires. Ce gène est plus faiblement exprimé dans le cerveau (dans l’hippocampe) chez les personnes homozygotes pour le variant ; par ailleurs, certains SNP situés dans ce gène ont été associés au risque de glaucome. Une étude plus approfondie reprenant l’ensemble des données amène les auteurs à suggérer que l’effet du gène APOE correspond à l’élimination relativement précoce de porteurs de l’allèle e4, tandis que les effets de USP42 et TMTC2 correspondraient réellement à un effet sur la longévité extrême. En tout état de cause, et comme l’indiquent les auteurs, ces résultats restent décevants dans la mesure où les allèles trouvés ne sont présents que chez une toute petite partie des centenaires étudiés4 et ne peuvent donc rendre compte que d’une faible composante de l’héritabilité de la longévité – malgré l’ampleur de l’étude et la taille de l’échantillon de centenaires rassemblé à grande peine. |
Difficultés scientifiques et engouement médiatique La recherche des composants génétiques de la longévité s’avère donc bien difficile [2], même lorsqu’on arrive à rassembler de grandes cohortes de « supercentenaires » et que l’on prend soin de définir le plus précisément possible la grandeur mesurée [4]. Ce n’est pas très étonnant puisque l’on peut a priori penser qu’il s’agit d’un caractère très multigénique : rappelons que pour la schizophrénie, affection fortement héritable, il a fallu attendre des analyses GWAS portant sur des dizaines de milliers d’individus pour voir apparaître des signaux significatifs [
5]. De plus, l’héritabilité de la longévité, que l’on savait déjà peu importante (0,25), ne serait en fait que de 0,10 à 0,15 d’après l’analyse rapportée au début de cette chronique [1]. Pourtant l’idée que les gènes jouent un grand rôle dans notre espérance de vie est très répandue et est sous-jacente dans le business model de plusieurs entreprises récentes comme Human Longevity, Inc (HLI, San Diego, États-Unis), affirmant qu’elle combine « séquençage d’ADN, analyse experte et apprentissage informatique »5 pour révolutionner la médecine, ou Calico (California Life Company, San Francisco, États-Unis), émanation de Google et dont la mission est la recherche sur la longévité et les thérapies associées. Il semble peu probable que, malgré leurs moyens, ces entreprises parviennent rapidement à des résultats significatifs sur le plan purement génétique. Dans la mesure où leurs travaux ne se limitent pas à l’ADN, mais s’intéressent à la protéomique et à la métabolomique, et compte tenu de la qualité des scientifiques impliqués (par exemple David Botstein pour Calico), il reste tout à fait possible qu’ils débouchent sur des avancées significatives. Cependant, même si la « part génétique » de la longévité est minoritaire, il reste intéressant de définir des gènes et des allèles influant sur cette caractéristique. Compte tenu des difficultés, il ne faut pas s’attendre à des révélations rapides, mais l’accroissement de la proportion de personnes très âgées dans la population devrait faciliter le recrutement, et la baisse – qui se poursuit – du coût du séquençage de génomes va faciliter des études plus détaillées et plus résolutives. L’espoir, à terme, est que les gènes qui pourront alors être identifiés fourniront des indications précieuses pour comprendre les mécanismes du vieillissement et peut être pour arriver à le ralentir. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Kaplanis
J
,
Gordon
A
,
Shor
T
, et al.
Quantitative analysis of population-scale family trees with millions of relatives . Science.
2018; ; 360 : :171.–175. 2.
Hook
M
,
Roy
S
,
Williams
EG
, et al.
Genetic cartography of longevity in humans and mice: Current landscape and horizons . Biochim Biophys Acta.
2018
Feb 2. pii: S0925–4439(18)30037–1. doi: 10.1016/j.bbadis.2018.01.026.
3.
Sebastiani
P
,
Nussbaum
L
,
Andersen
SL
, et al.
Increasing sibling relative risk of survival to older and older ages and the importance of precise definitions of aging, life span, and longevity . J Gerontol A Biol Sci Med Sci.
2016; ; 71 : :340.–346. 4.
Sebastiani
P
,
Gurinovich
A
,
Bae
H
, et al.
Four genome-wide association studies identify new extreme longevity variants . J Gerontol A Biol Sci Med Sci.
2017; ; 72 : :1453.–1464. 5.
Schizophrenia working group of the psychiatric genomics consortium . Biological insights from 108 schizophrenia-associated genetic loci . Nature.
2014; ; 511 : :421.–427. |