| |
| Med Sci (Paris). 34(2): 179–182. doi: 10.1051/medsci/20183402017.Bases alternatives et organismes synthétiques Chroniques Génomiques Bertrand Jordan1* 1UMR 7268 ADÉS, Aix-Marseille, Université/EFS/CNRS; CoReBio PACA, case 901, Parc scientifique de Luminy, 13288Marseille Cedex 09, France MeSH keywords: Acides aminés, Appariement de bases, ADN, Évolution moléculaire dirigée, Évolution moléculaire, Régulation de l'expression des gènes au cours du développement, Code génétique, Humains, Ingénierie des protéines, Biologie synthétique, composition chimique, génétique, synthèse chimique, méthodes, tendances |
La biologie de synthèse continue à faire parler d’elle. J’avais évoqué la synthèse d’un génome bactérien [1](➔) ainsi que, plus récemment, les travaux sur la levure et le projet – à mon avis chimérique – de la construction d’un génome humain par synthèse d’ADN [2] (➔). (➔)Voir la Chronique génomique de B. Jordan, m/s n° 6-7 juin-juillet 2016, page 651 (➔)Voir la Chronique génomique de B. Jordan, m/s n° 10, octobre 2016, page 898 Les travaux sur la levure avancent et près de la moitié de ce génome de 12 mégabases a déjà été assemblé1; mais la nouvelle qui fait l’objet de cette chronique, concerne une approche différente, celle de l’élaboration de nouvelles bases nucléotidiques et de leur incorporation dans l’ADN pour diriger in vivo l’incorporation de nouveaux acides aminés dans des protéines [3]. Il s’agit là d’une avancée importante du point de vue conceptuel puisqu’elle étend le vocabulaire de l’hérédité, jusqu’ici limité aux quatre bases classiques T, A, G, C, et modifie ainsi les mécanismes les plus fondamentaux de la vie. Ce procédé peut également avoir des retombées pratiques notables en étendant considérablement les possibilités de modification de protéines. |
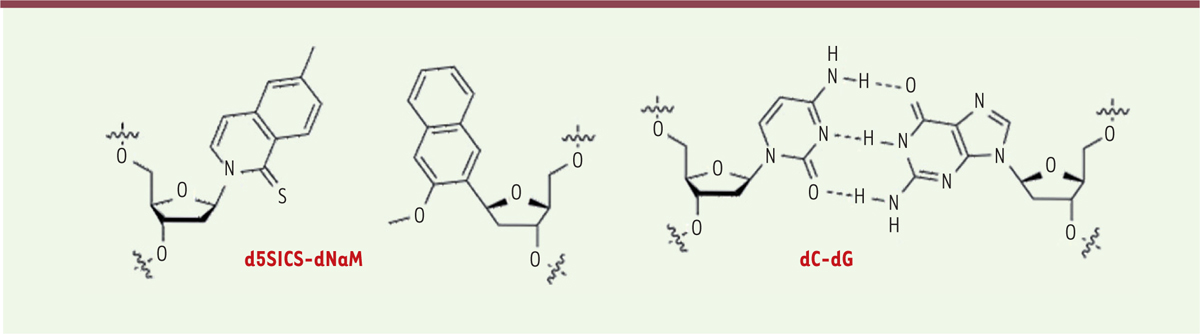
Bases alternatives, les débuts Plusieurs équipes se sont intéressées, dès la fin des années 1980, à la synthèse de paires de bases « alternatives » susceptibles d’être incorporées dans l’ADN [4]. Il s’est avéré que l’appariement de ces bases au sein de la double hélice ne devait pas nécessairement se faire par liaisons hydrogène (comme pour les paires classiques A-T et C-G), mais pouvait reposer sur leur compatibilité stérique et sur des interactions hydrophobes. La réplication et la transcription de segments d’ADN contenant ces entités avaient été démontrées in vitro, et un pas important fut franchi en 2014, avec la démonstration qu’une paire de bases alternatives, d5SICS-dNaM (Figure 1), pouvait être incorporée dans l’ADN d’un plasmide et que ce dernier pouvait être répliqué dans un hôte bactérien [5]. La bactérie employée était bien particulière puisque, pour permettre la duplication du plasmide, elle devait fournir à l’ADN polymérase les nucléosides triphosphates2, alternatifs : les auteurs tournèrent la dif-ficulté en ajoutant au génome bactérien le gène d’un transporteur de triphosphates (isolé d’une diatomée3,). Il suffisait dès lors d’ajouter les précurseurs des deux nucléotides alternatifs au milieu de culture pour permettre leur utilisation par la bactérie. À partir de là, les ADN polymérases bactériennes s’avérèrent capables de synthétiser l’ADN contenant la paire alternative. Celle-ci reste relativement stable au fil de la croissance bactérienne et des réplications successives du plasmide puisqu’après 15 h de culture, sa rétention est supérieure à 99 % [5]. Même si la stabilité à long terme n’était pas assurée, en raison sans doute de l’intervention des mécanismes d’excision et de réparation, on avait bien là le premier exemple d’un organisme incorporant dans son ADN deux bases alternatives créées par l’homme et bien distinctes des quatre qui sont le fondement de tous les êtres vivants et témoignent de leur origine commune. C’était donc un résultat important, mais encore incomplet dans la mesure où cette paire alternative ne jouait aucun rôle dans le fonctionnement de la bactérie puisqu’elle n’était ni transcrite ni traduite. C’est ce qu’apporte, trois ans plus tard, l’article qui fait l’objet de cette chronique [3].  | Figure 1.La paire de bases alternatives employée par Malyshev et al. [5], à gauche, comparée à une paire classique dC-dG. La liaison est assurée par les interactions hydrophobes. Les entités représentées sont en fait des désoxyribonucléosides (base liée à un désoxyribose). |
|
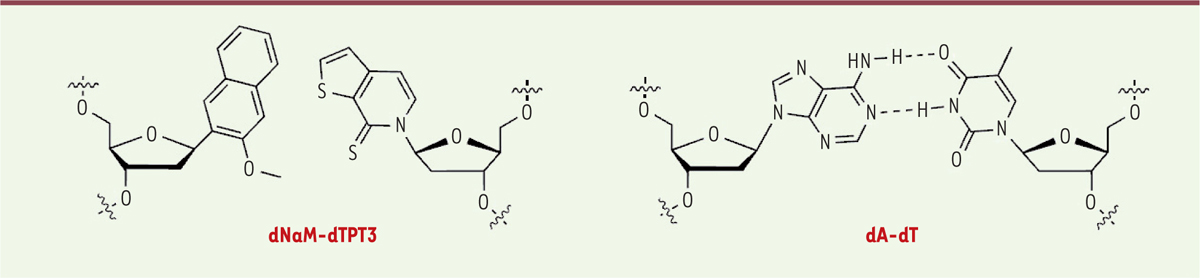
De la paire de bases à la protéine L’étape suivante est l’œuvre de la même équipe, et elle représente un progrès considérable puisqu’elle démontre la transcription et la traduction, in vivo, d’un ADN contenant une paire alternative, aboutissant à la synthèse d’une protéine contenant un acide aminé lui aussi « alternatif ». Cela suppose l’existence d’un ARN de transfert (ARNt) comportant une base alternative dans son anticodon, le chargement de cet ARN par l’acide aminé alternatif grâce à une aminoacyl ARNt synthé- tase spécifique, et une incorporation efficace de cet acide aminé dans la protéine… un ensemble d’étapes délicates, mais que les auteurs ont accomplies avec succès et présentent dans un article assez dense [3]. La protéine choisie est une version superfolder de la GFP (green fluorescent protein), dite sfGFP, qui présente une structure secondaire très solide et compacte, et peut du coup tolérer l’insertion d’un acide aminé quelconque (y compris « alternatif ») en position 151 (normalement une sérine) sans perdre ses propriétés de fluorescence. Le couple de bases alternatives est cette fois dNaM-dTPT3 (Figure 2), désigné par la suite par X-Y. 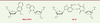 | Figure 2.La paire de bases alternatives (nucléosides, en fait) utilisée dans l’article de Zhang et al. [3], comparée à une paire dA-dT. |
Dans un premier temps, on a introduit dans la bactérie construite lors des travaux précédents [5], le gène sfGFP comportant le codon 151 AXC (remplaçant un codon sérine AGC) ainsi qu’un gène d’ARNt-sérine comportant l’anticodon complémentaire GYT. Après croissance en présence des précurseurs nécessaires et induction (les gènes étant portés par un vecteur inductible), il y a production de la protéine sfGFP si et seulement si les bases alternatives X et Y sont présentes dans le gène et dans l’ARNt. Cette sfGFP a incorporé la sérine en position 151 : il y a donc bien eu transcription et traduction de la base alternative sans perte de fidélité. La deuxième étape consiste naturellement à utiliser les bases alternatives pour introduire un acide aminé lui aussi alternatif. Il faut pour cela fournir l’acide aminé alternatif (ici la N6-[(2-pro- pynyloxy)carbonyl]-L-lysine, en abrégé PrK, pour Propyl-lysine) qui est ajouté dans le milieu de culture, un ARNt qui puisse être chargé avec cet acide aminé (ici un ARNt spécifique pour la pyrrolysine4 provenant de l’archée Methanosarcina barkeri, ou plutôt son gène, modifié de manière à ce que l’anticodon soit GYT), et surtout l’aminoacyl ARNt synthétase capable de charger cet ARNt avec l’acide aminé alternatif PrK : cette enzyme baptisée PylRS (pyrrolysyl-tRNA synthetase) provient de la même archée et est également introduite dans la bactérie grâce à un plasmide portant son gène. Dans ces conditions, la production de fluorescence après transformation des bactéries atteste que la protéine sfGFP est bien synthétisée lorsque le gène correspondant contient le codon AXC, l’ARNt porte l’anticodon GYT et que l’aminoacyl ARNt synthétase PylRS est produite dans la cellule. L’intensité de la fluorescence (60 à 70 % du témoin non alternatif) indique que transcription et traduction se font avec une bonne efficacité; une analyse chimique assez sophistiquée (et que je ne décrirai pas) montre que l’acide aminé PrK est bien présent à la position 151 de la protéine sfGFP. Une analyse quantitative après purification confirme que la production de sfGFP est de l’ordre de 50 % de celle du contrôle (sans bases alternatives) et que sa pureté (évaluée par spectrométrie de masse) est supérieure à 95 %. En somme, on a bel et bien inséré dans la protéine un acide aminé alternatif (PrK) grâce à la présence d’un codon alternatif AXC dans le gène, lu par un anticodon GYT dans l’ARN de transfert. Des résultats équivalents sont obtenus en utilisant un gène sfGFP contenant le codon GXC en position 151 associé à un ARNt dont l’anticodon est GYC. L’expérience a également été répétée avec un autre acide aminé alternatif et un autre couple ARNt/aminoacyl ARNt synthétase provenant d’une autre souche d’archée. |
Une avancée fondamentale et des conséquences pratiques On ne peut qu’être impressionné par la sophistication des techniques mises en œuvre dans ces travaux : cela témoigne de la maîtrise désormais acquise dans la manipulation de systèmes bactériens. On peut aujourd’hui jongler avec les différents éléments (ADN, ARN, transporteurs et organelles) pour construire des bactéries à la demande en assemblant des « pièces détachées » provenant d’organismes très divers. Le point le plus critique, si l’on veut incorporer un acide aminé alternatif, est sans doute de disposer de l’aminoacyl ARNt synthétase correspondante : dans l’exemple décrit ci-dessus, on joue sur le fait que l’acide aminé alternatif ressemble à un acide aminé atypique présent chez une archée, ce qui permet d’utiliser l’enzyme provenant de cet organisme. Quoi qu’il en soit, la démonstration est ainsi faite que l’on peut créer de toutes pièces de nouvelles lettres à ajouter à l’alphabet classique (T, A, G et C) et surtout les faire fonctionner in vivo, y compris jusqu’à diriger l’insertion de nouveaux acides aminés dans des protéines. Cette avancée est importante tant du point de vue théorique que pratique [6]. |
On a débattu par le passé pour savoir si le code génétique est arbitraire, ou si, au contraire, il existe une relation structurale entre codons et acides aminés, limitant la gamme des codons possibles pour un acide aminé donné. Plus généralement, on s’est interrogé sur le caractère aléatoire de l’évolution, en se demandant ce qui se passerait si l’on pouvait reprendre le développement de la vie depuis le début : retrouverait-on les mêmes molécules, les mêmes étapes ? Stephen Jay Gould a développé de manière très convaincante l’idée que le hasard a joué un grand rôle dans ces étapes initiales et qu’en cas de « replay », le déroulement de l’évolution et même la structure de la vie pourraient être très différents [7]. Dans cette optique, notre code génétique apparu il y a plus de trois milliards d’années, n’est universel que parce que toutes les formes de vie actuelles descendent du même organisme primitif souvent baptisé LUCA (last universal common ancestor). Le fait que l’on puisse ajouter de nouvelles lettres à ce code et aboutir à un système fonctionnel va tout à fait dans ce sens. |
Le fait de disposer maintenant de six lettres (dont les deux nouvelles X et Y) dans l’alphabet de l’ADN augmente considérablement la gamme des entités protéiques dont on peut envisager la synthèse. On pourrait, par exemple, (ce n’est qu’une des nombreuses possibilités) modifier la spécificité d’une cytokine comme l’interleukine 2 (souvent utilisée en cancérologie) grâce à l’insertion d’un acide aminé alternatif en un point précis de sa chaîne peptidique. Floyd Romesberg, l’auteur senior de l’article rapporté ici [3], a d’ailleurs fondé une entreprise appelée Synthorx dont l’objectif est de « créer de nouvelles protéines pour de nouvelles thérapies » (create novel proteins for new therapeutics). D’une manière générale, cette possibilité d’extension du code génétique (et du répertoire des acides aminés) vient s’ajouter aux facilités offertes par les techniques de modification du génome (CRISPR-Cas9 et dérivés) et aux performances accrues des systèmes de synthèse d’ADN pour faire entrevoir une nouvelle ingénierie du vivant [8]. Celle-ci aura à coup sûr un impact considérable en biotechnologie, ouvrant la voie à la fabrication de molécules et d’organismes « sur mesure » répondant à des besoins très variés. Elle aura aussi des retombées dans le domaine clinique : on en a déjà un aperçu avec les thérapies anticancéreuses employant des lymphocytes T largement modifiés [9] (➔). Ce n’est là que le début d’une révolution dont les implications médicales mais aussi éthiques vont être considérables. (➔)Voir la Chronique génomique de B. Jordan, m/s n° 11, novembre 2017, page 1003 |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article. médecine/sciences (m/s) est une revue internationale mensuelle francophone de diffusion des connaissances scientifiques, médicales et en santé. Les articles qui y sont publiés s’adressent à des chercheurs scientifiques et cliniciens, des professeurs, des étudiants et des médecins. Ouverte à ce large public, la revue se veut compréhensible par tous, quelle que soit leur discipline. Elle rapporte les avancées les plus récentes de la recherche en biomédecine. Les articles sont référencés dans PubMed depuis 2003, à l’exception des Brèves. En écrivant dans m/s, les auteurs font partager aux lecteurs, dans leur langue, leur intérêt et même leur enthousiasme pour le sujet traité, en y apportant leur touche d’humour et de culture, surtout dans la partie Forum, où la plus grande liberté d’expression est autorisée. m/s s’adressant à un lectorat très varié, exige des auteurs de développer leurs sujets, en allant jusqu’au bout des connaissances scientifiques, quelle qu’en soit la difficulté. Notamment pour les Synthèses, états de l’art sur un sujet donné, dans un style clair et intelligible pour les non-spécialistes. |
Footnotes |
1. Jordan B. Synthétique, vous avez dit « Synthétique » ? Med Sci. (Paris). 2016;; 32 : :651.-3. 2. Jordan B. HGP-write : après la lecture, l’écriture ? Med Sci (Paris). 2016;; 32 : :898.-901. 3. Zhang Y, Ptacin JL, Fischer EC, et al. A semi-synthetic organism that stores and retrieves increased genetic information. Nature. 2017;; 551 : :644.-7. 4. Benner SA, Karalkar NB, Hoshika S, et al. Alternative Watson-Crick synthetic genetic systems. Cold Spring Harb Perspect Biol. 2016;; 8 : a023770.. 5. Malyshev DA, Dhami K, Lavergne T, et al. A semi-synthetic organism with an expanded genetic alphabet. Nature. 2014;; 509 : :385.-8. 6. Callaway E. Alien DNA makes proteins in living cells for the first time. Nature. 2017;; 551 : :550.-1. 7. Gould SJ. The structure of evolutionary theory. Harvard : Harvard University Press. , 2002 : :1434. p. 8. Chari R, Church GM. Beyond editing to writing large genomes. Nat Rev Genet. 2017;; 18 : :749.-60. 9. Jordan, B. Immunothérapie « CAR-T » : une autorisation qui fait date. Med Sci (Paris). 2017;; 33 : :1003.-6. |