Le phénotype d’un organisme multicellulaire est généralement considéré comme le produit de l’expression de son génome et de son interaction avec l’environnement. En révélant l’extraordinaire étendue de la diversité bactérienne et archéenne qui vit sur et à l’intérieur des organismes multicellulaires, les récents progrès technologiques du séquençage de l’ADN ont remis en cause cette vision simpliste. Il est de plus en plus évident que ces communautés de microorganismes, appelées microbiote1, influencent profondément les phénotypes de l’hôte qui les portent et vice-versa : hôtes et microbiotes seraient hautement interdépendants [1, 2]. L’intestin des animaux représente des conditions de vie propices à la croissance des microorganismes qu’il héberge : on y retrouve en effet la nourriture en abondance (les aliments en cours de digestion) et une très faible teneur en oxygène (un poison pour ces microorganismes qui sont pour la plupart strictement anaérobies). Ces communautés permettent en retour de compenser les déficits enzymatiques de l’hôte en digérant une partie de la nourriture, ce qui permet une meilleure extraction de l’énergie contenue dans les molécules alimentaires complexes [3] (→).
(→) Voir la Nouvelle de A. El Kaoutari et al., m/s n° 3, mars 2014, page 259
De nombreuses maladies, notamment auto-immunes, sont associées à des déséquilibres de l’écosystème microbien intestinal et des solutions thérapeutiques fondées sur la restauration de la biodiversité intestinale sont développées [2] (→).
(→) Voir aussi le numéro thématique Le microbiote : cet inconnu qui réside en nous, m/s n° 11, novembre 2016
La définition classique d’un « organisme biologique » comme entité indépendante a ainsi été remise en cause [4]. Selon la nouvelle vision, un organisme est un ensemble d’entités interdépendantes fonctionnant en réseaux, permettant ainsi d’intégrer à la définition d’un organisme multicellulaire, l’ensemble des microbiotes qui sont en constante interaction fonctionnelle avec lui. Cependant, contrairement au génome qui est transmis verticalement et quasi fidèlement de la mère à l’enfant, l’organisme doit acquérir de novo, à chaque génération, l’ensemble des bactéries formant son microbiote, à partir de son environnement qui, lui-même, pourra influencer la composition du microbiote tout au long de sa vie.
La forte interdépendance fonctionnelle supposée entre l’organisme hôte et son microbiote intestinal est souvent interprétée comme le résultat d’une co-évolution2, entre les deux acteurs depuis des millions d’années. Pourtant, les mécanismes qui régissent l’évolution et la diversification3 des microbiotes intestinaux de mammifères restent encore très mal connus et sujets à controverses [5]. Comment cet ensemble d’organismes (microbiote intestinal et hôte) a-t-il évolué au cours du temps ? Est-ce véritablement le résultat d’une longue et intime co-évolution ? Si c’est le cas, quels sont les mécanismes principaux qui régissent cette co-évolution et la diversification des microbiotes intestinaux de mammifères ? Afin de répondre à ces questions, il est d’abord primordial de décrire la diversité des microbiotes intestinaux de mammifères et les facteurs qui la modulent. De nombreux travaux ont été entrepris en écologie « classique » depuis des siècles (par exemple, par le naturaliste suédois Carl von Linné, ou Alfred Russel Wallace, anthropologue et biologiste britannique, ou le naturaliste anglais Charles Darwin) afin de décrire et de comprendre les caractéristiques de la distribution géographique des animaux et des plantes sur la planète. Il est, par exemple, très bien établi que la richesse des animaux varie de façon systématique avec la latitude ou l’altitude. Dans le cas des microbiotes intestinaux, ces facteurs sont beaucoup plus discutés. Ainsi, la question de savoir qui, du régime alimentaire ou de la proximité phylogénétique des espèces de mammifères, influence majoritairement la composition du microbiote et la distribution des bactéries intestinales est grandement débattue.
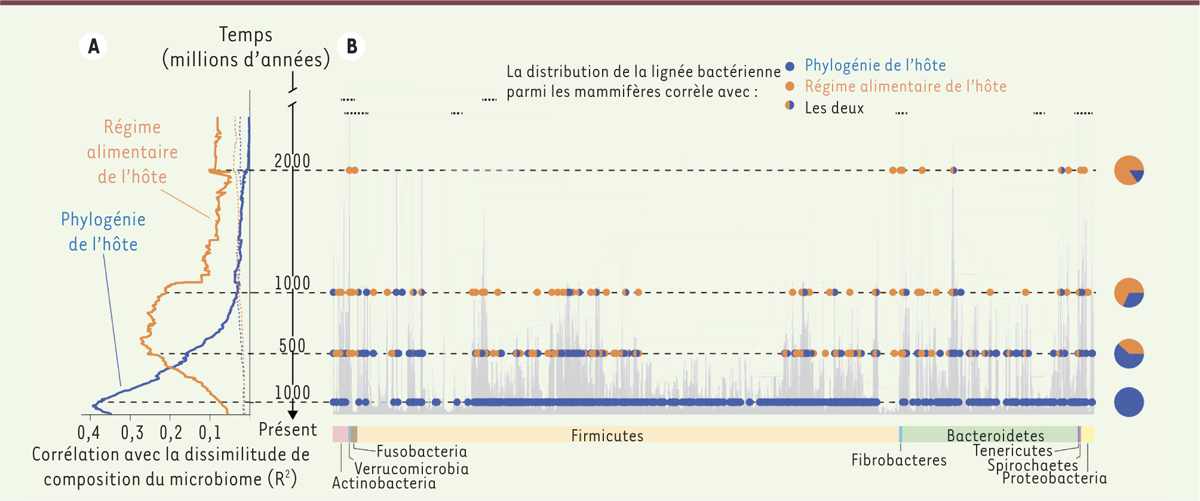
Dans notre travail récent [6], nous montrons que le régime alimentaire et la phylogénie de l’hôte influencent tous deux très fortement la composition microbienne de l’intestin, mais à des niveaux taxonomiques bactériens très différents (Figure 1A et B). Les espèces de mammifères qui se sont adaptées de manière convergente4 à un même régime alimentaire, par exemple l’herbivorie (comme le kangourou et la biche), ont acquis dans leur intestin des bactéries identiques appartenant à de grandes catégories taxonomiques. L’intestin des herbivores est en effet enrichi en bactéries de la famille des Ruminococcaceae et Lachnospiraceae qui produisent des enzymes nécessaires à la dégradation de sucres complexes de plantes (comme la cellulose). Ces enzymes n’étant pas codées par le génome de l’hôte, ces bactéries jouent donc un rôle fondamental dans l’assimilation, par l’hôte, de ces molécules énergétiques. Les carnivores, de leur côté, ont un microbiote intestinal enrichi en certaines familles de bactéries (telles Lactobacillus) impliquées dans la dégradation d’acides aminés, en forte concentration dans leur régime alimentaire.
| Figure 1.
La phylogénie et le régime alimentaire de l’hôte déterminent la composition des microbiotes intestinaux de mammifères à différentes échelles taxonomiques bactériennes. A. Ce graphique montre l’intensité de la corrélation entre la dissemblance de composition du microbiote intestinal entre espèces de mammifères et leur distance phylogénétique (en bleu) ou leur différence de régime alimentaire (en orange). L’axe vertical indique le temps de divergence entre les lignées bactériennes peuplant les microbiotes de mammifères. À chaque temps t, les différents groupes bactériens sont définis, leur distribution à travers les hôtes calculée, et ainsi la composition des microbiotes déterminée. Les corrélations sont calculées à l’échelle des compositions globales des microbiotes. La phylogénie de l’hôte est fortement associée aux compositions du microbiote lorsque celles-ci sont déterminées en définissant les lignées bactériennes à un niveau taxonomique récent. En revanche, le régime alimentaire influence la composition du microbiote à plus large (c’est-à-dire ancienne) échelle taxonomique bactérienne. B. La phylogénie calibrée des bactéries intestinales observées chez l’ensemble des mammifères est représentée en gris. Les corrélations sont ici montrées à l’échelle des lignées bactériennes individuelles. Chaque point bleu ou orange indique que la distribution de la lignée bactérienne à travers les mammifères corrèle significativement avec leur distance phylogénétique ou leur distance de régime alimentaire, respectivement. Les camemberts sur la droite résument la proportion de lignées individuelles dont la distribution corrèle avec la phylogénie de l’hôte ou le régime alimentaire. |
Bien qu’ils aient en commun de grandes familles bactériennes, des espèces ayant le même régime alimentaire n’abritent pas les mêmes bactéries. À un niveau taxonomique plus précis, la répartition des bactéries entre intestins de mammifères n’est plus expliquée par des différences de régimes alimentaires, mais semble refléter la position phylogénétique des espèces [6] (Figure 1A et B). Deux espèces proches dans l’arbre phylogénétique des mammifères vont ainsi avoir tendance à partager davantage d’espèces ou de souches bactériennes, à régime alimentaire constant. Par exemple, le kangourou et les ruminants sont éloignés phylogénétiquement, mais tous deux hébergent des espèces de bactéries de la famille Ruminococcaceae. Cependant, les ruminants partagent entre eux des espèces bactériennes proches qui sont totalement différentes de celles présentes chez le kangourou. Cette distribution particulière des bactéries intestinales reflétant la phylogénie de l’hôte, a parfois été appelée « phylosymbiose » [7].
Dès lors, comment interpréter le fait que des espèces de mammifères proches vont avoir tendance à partager des groupes taxonomiques bactériens qui sont apparus récemment (espèces ou souches bactériennes) ? Cette observation a généré de nombreuses controverses ces dernières années [4, 5, 8, 9]. Pour certains, il s’agit d’une véritable co-diversification5,, voire d’une co-évolution, entre les microbes et leurs hôtes, résultat d’une interaction mutuelle et durable (sur des centaines de milliers voire des millions d’années) qui a façonné l’évolution des deux partenaires symbiotiques par le biais de la sélection naturelle [4, 8]. D’autres ont très vivement critiqué cette interprétation [5], proposant un processus de filtre écologique n’impliquant pas de co-diversification et de co-évolution, mais produisant un signal de phylosymbiose. Des hôtes proches phylogénétiquement ont ainsi tendance à posséder des intestins aux caractéristiques biochimiques similaires (comme le pH). Ces conditions intestinales, définissant les niches écologiques bactériennes, influencent donc la nature des bactéries acquises susceptibles de se développer. Les intestins d’espèces de mammifères proches peuvent être colonisés par des espèces, ou souches, bactériennes similaires qui occuperont les mêmes niches écologiques.
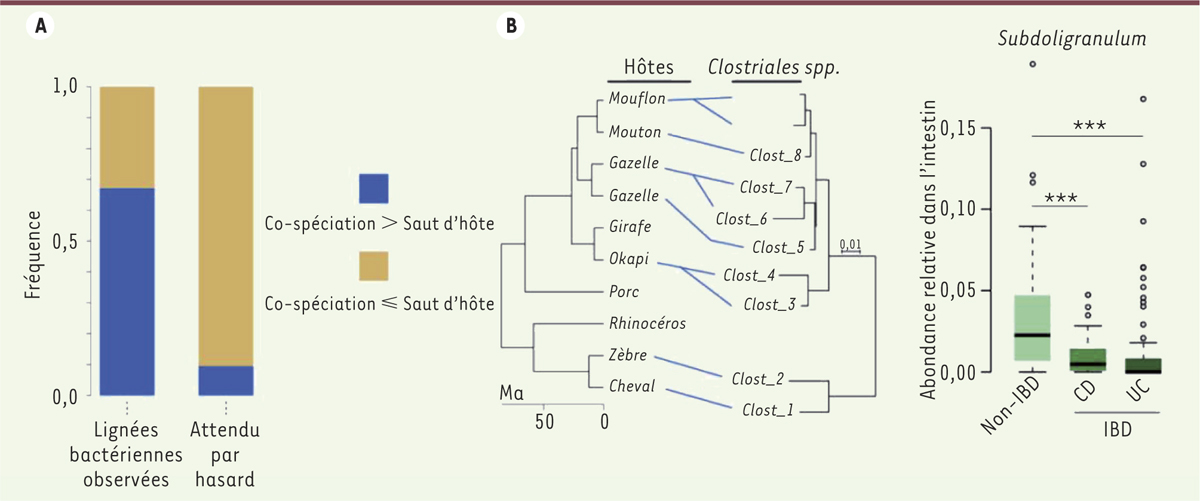
Nous avons tenté de distinguer ces deux hypothèses [6]. Une trace possible de la co-évolution entre deux partenaires est la co-spéciation6, : lorsqu’une des deux espèces impliquées dans une relation symbiotique fonctionnelle subit un événement de spéciation, l’autre partenaire peut également subir, au même moment, un événement de spéciation. Les phylogénies de l’hôte (ici les mammifères) et du ou des symbiontes (ici les bactéries intestinales) sont alors congruentes : pour une espèce bactérienne donnée, la lignée présente chez l’homme sera plus proche phylogénétiquement de celle présente chez le chimpanzé, que de celle présente chez le gorille. Au contraire, il n’est pas attendu que le processus de filtre écologique, expliqué plus haut, entraîne des co-spéciations entre mammifères et bactéries. Nous avons donc tenté de quantifier l’étendue de ces co-spéciations et avons trouvé que le phénomène de co-spéciation entre mammifères et bactéries intestinales se produisait bien plus qu’attendu par hasard (Figure 2A et 2B). La relation entre mammifères et bactéries intestinales peut donc être très intime, et cela sur des millions d’années d’évolution au point que lorsque les mammifères subissent un évènement de spéciation, certaines bactéries intestinales font de même. Qu’implique cette persistance relationnelle ? Un des processus sous-jacents à la co-spéciation peut être la co-évolution (hypothèse de co-évolution). Cependant, une forte transmission verticale des bactéries intestinales de la mère à l’enfant, couplée à un processus de spéciation allopatrique7 des hôtes, pourrait produire des évènements de co-spéciation sans impliquer nécessairement de co-évolution (hypothèse neutre). Nous avons tenté de distinguer ces deux hypothèses. Dans le cas d’une co-évolution véritable, les partenaires de la relation devraient être interdépendants fonctionnellement, signifiant que le succès reproducteur (souvent appelé « fitness ») d’un des partenaires dépend de la présence de son partenaire fonctionnel. Dans le cas de l’hypothèse neutre, l’absence d’un des partenaires a peu d’effet sur la santé ou la « fitness » de l’autre. Nous avons donc étudié si des lignées bactériennes en forte co-spéciation avec les mammifères, tout en étant présentes chez l’homme, étaient associées à la santé humaine. L’abondance de certaines bactéries est systématiquement réduite chez des patients atteints de la maladie de Crohn ou de rectocolite hémorragique, deux maladies inflammatoires chroniques de l’intestin (MICI) [10]. Nous avons effectivement observé que de telles bactéries, appartenant aux genres Subdoligranulum, Oscillobacter, Butyricoccus, Sporobacter et Anaerotruncus, avaient de forts taux de co-spéciation avec l’homme (Figure 2C). En contribuant à maintenir un niveau d’inflammation bas dans l’intestin humain, ces bactéries sont des candidats idéaux pour la recherche de marqueurs génétiques bactériens et humains qui seraient en co-évolution et qui auraient une importance fonctionnelle dans la physiologie de l’hôte. Elles représentent également des candidats idéaux pour la recherche thérapeutique dans le but de mettre au point des probiotiques efficaces, capables de coloniser de manière pérenne les intestins d’individus atteints de ces maladies intestinales et de restaurer l’équilibre physiologique de l’écosystème intestinal [11] (→).
(→) Voir la Nouvelle de S. Normand et al., m/s n° 6-7, juin-juillet 2013, page 586
| Figure 2.
Co-spéciation entre bactéries intestinales et mammifères, et lien avec les maladies inflammatoires intestinales chez l’humain. A. Transmission de bactéries intestinales d’un hôte à l’autre, soit via un héritage vertical (co-spéciation avec l’hôte), soit via des sauts d’un hôte à l’autre. La couleur bleue représente les bactéries se transmettant avec un taux de co-spéciation supérieur au taux de saut d’hôtes, la couleur orangée représentant le cas inverse. La proportion de bactéries ayant une tendance à se transmettre majoritairement par des événements de co-spéciation est supérieure à celle attendue par hasard, calculée à l’aide d’une hypothèse nulle. B. Exemple d’une bactérie intestinale appartenant à la famille des Clostridiales se transmettant par co-spéciation avec des espèces de mammifères (Ma). Chaque trait bleu indique la présence de la bactérie dans l’intestin de l’hôte. La topologie de la phylogénie bactérienne est parfaitement congruente avec celle de la phylogénie des hôtes, indice suggérant que les événements de spéciation bactérienne ont été congruents avec ceux des hôtes. C. Abondance relative d’une lignée bactérienne appartenant au genre Subdoligranulum qui est en co-spéciation chez les primates, incluant l’homme, et qui est associée avec deux maladies inflammatoires de l’intestin (inflammatory bowel disease, IBD) : la maladie de Crohn (CD) et la rectocolite hémorragique (UC, aussi appelée colite ulcéreuse). Chez les patients atteints d’IBD, il est observé une diminution significative de l’abondance de cette bactérie Subdoligranulum. |
L’association entre bactéries qui co-spécifient ou co-évoluent avec l’hôte et la santé humaine devrait pouvoir être étendue à d’autres phénotypes immunologiques ou physiologiques. Certaines espèces de Bacteroides ou de Clostridium favorisent en effet la différenciation de lymphocytes T en lymphocytes T régulateurs anti-inflammatoires. Des espèces des genres Bifidobacterium, Lactobacillus, et Faecalibacterium jouent un rôle protecteur contre l’inflammation de la muqueuse en inhibant la production de cytokines pro-inflammatoires ou en stimulant celle d’autres cytokines anti-inflammatoires comme l’interleukine 10. À quel degré ces bactéries sont en co-spéciation et/ou co-évolution avec l’homme reste inconnu.
Notre étude a donc permis de mettre en évidence qu’incorporer une approche évolutive dans l’analyse des compositions des microbiomes intestinaux et de leurs constituants eux-mêmes peut permettre de révéler les éléments qui ont un fort impact sur la santé de l’hôte. Si le microbiote intestinal inclut des éléments qui influencent la santé de l’hôte, la sélection naturelle a dû, et doit encore, agir sur ces éléments (ou sur le couple microbiote/hôte) et y laisser des traces dans les compositions actuelles des microbiotes ou dans les génomes bactériens. Révéler ces signatures évolutives revêt donc un enjeu majeur pour le traitement futur des maladies liées à un déséquilibre de la flore intestinale.