Les marques épigénétiques constituent le véritable support de la mémoire cellulaire parce qu’elles permettent à une cellule d’exprimer un nombre bien défini de gènes qui vont déterminer le type cellulaire et son identité [1, 2]. En effet, pour qu’une cellule du foie soit toujours une cellule hépatique et qu’une cellule du rein soit toujours une cellule rénale, certains gènes doivent demeurer « muets » ou « silencieux » alors que d’autres doivent être constamment exprimés. La capacité à rendre muet un gène est assurée, entre autres, par les cytosines méthylées (une des quatre bases de l’ADN portant un groupement méthyle) positionnées à des endroits stratégiques du génome. La présence de cytosines méthylées empêche l’expression d’un gène et, in fine, la traduction de la protéine correspondante. La méthylation de la cytosine s’effectue sur les 2 brins de l’ADN lorsqu’elle est située à côté d’une guanine. Cette symétrie permet ainsi la transmission semi-conservative de la méthylation de l’ADN (Figure 1) selon un modèle qui fait appel à des acteurs bien particuliers (voir ci-après).
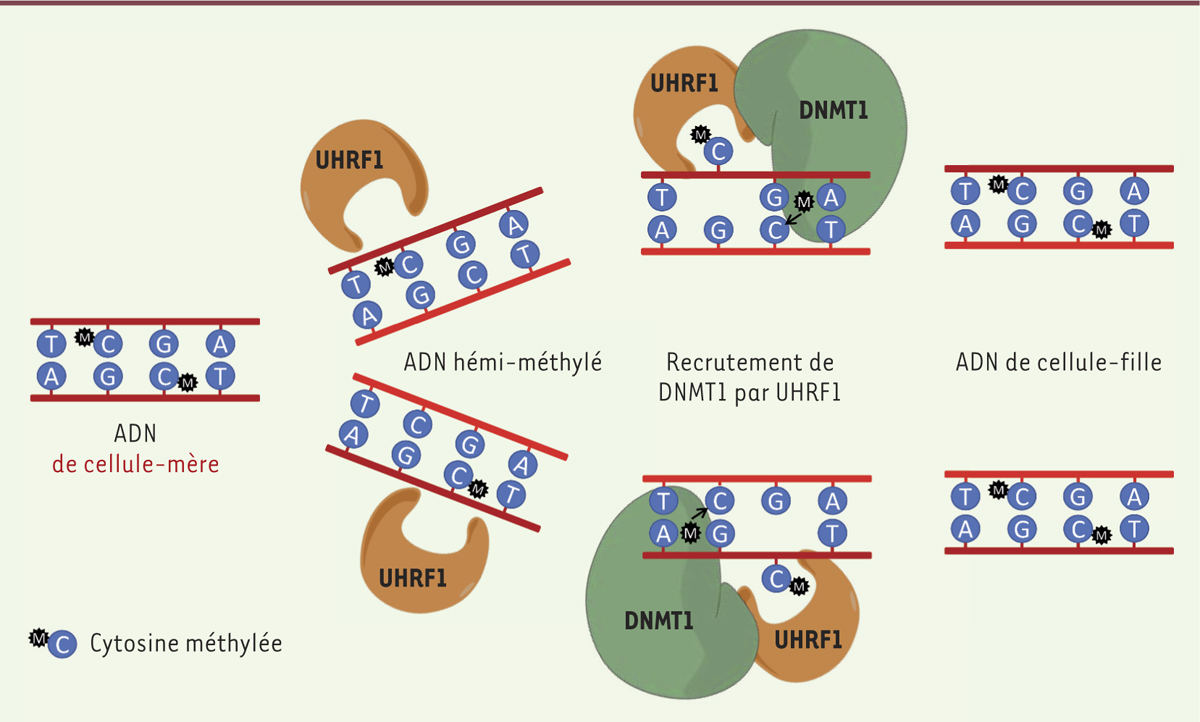
| Figure 1.
Représentation schématique de la coopération entre UHRF1 et DNMT1 pour la réplication de la méthylation de l’ADN. Dans la cellule-mère, l’ADN est méthylé sur ses deux brins au niveau de sites CpG. Lors de la duplication de l’ADN, il y a synthèse d’un nouveau brin dont les cytosines ne sont pas méthylées. L’ADN généré est donc un ADN hémi-méthylé (un seul des 2 brins est méthylé). Les sites CpG hémi-méthylés sont reconnus par la protéine UHRF1, grâce à son domaine SRA (set and ring associated). Cette reconnaissance est suivie d’un basculement de la cytosine méthylée hors de la double hélice d’ADN, afin de permettre à UHRF1 de recruter la protéine DNMT1 dans le but de lui indiquer quelle cytosine est à méthyler sur l’autre brin. DNMT1 : ADN méthyltransférase 1 ; UHRF1 : ubiquitin-like containing PHD and RING finger domains 1. |
Le défi, pour une cellule qui prolifère, est de recopier fidèlement les profils de méthylation à l’emplacement exact sur le génome, et de les transmettre à la descendance afin de préserver l’identité cellulaire. Une altération de cette fidélité signifierait, pour la cellule, une perte des capacités à assurer ses fonctions et, dans certains cas, l’apparition d’anomalies pouvant conduire à des cancers ou des maladies neuro-dégénératives de type maladie d’Alzheimer [3].
La cytosine méthylée n’existe pas, comme telle, à l’état naturel et c’est donc une cytosine nue qui est incorporée lors de la duplication de l’ADN, provoquant ainsi l’apparition d’un ADN hémi-méthylé, où un seul des deux brins de l’ADN est méthylé. L’une des premières étapes de la duplication du profil de méthylation implique, par conséquent, la reconnaissance de l’ADN hémi-méthylé. La méthylation de l’autre brin de l’ADN est assurée par la protéine DNMT1 (ADN méthyltransférase 1) [4] (→).
(→) Voir la Nouvelle de M. Weber, m/s n° 5, mai 2011, page 483
Pour aider cette dernière à identifier les cytosines à méthyler, il lui faut un guide, en l’occurrence la protéine UHRF1 (ubiquitin-like, containing PHD and RING finger domains 1) dont le domaine SRA (set and ring associated) reconnaît l’ADN hémi-méthylé [5]. Cette reconnaissance est suivie d’un basculement de la cytosine méthylée, hors de la double hélice de l’ADN, afin de permettre à l’UHRF1 de marquer une pause, probablement pour « dialoguer » avec l’enzyme DNMT1 afin de lui indiquer quelle cytosine est à méthyler sur l’autre brin (Figure 1).
La manière dont s’opère le dialogue de ce ménage à trois, entre l’UHRF1, la DNMT1 et l’ADN hémi-méthylé, reste un mystère. Sa compréhension ouvrirait des perspectives intéressantes pour corriger les erreurs ou inhiber le processus d’hyperméthylation. Prenons l’exemple du cancer, caractérisé notamment par la mise en « silence » de gènes bénéfiques comme les gènes suppresseurs de tumeurs. Les promoteurs de ces gènes sont fréquemment hyperméthylés car de nombreuses cytosines sont méthylées en amont du site d’initiation de la transcription. Lorsqu’ils sont déméthylés ou lorsque les profils de méthylation ne sont pas dupliqués, ces gènes peuvent à nouveau s’exprimer, avec pour conséquence l’activation de l’apoptose, un processus conduisant à la mort de la cellule. Ainsi, cibler la transmission des profils de méthylation serait un moyen ingénieux d’inciter la cellule cancéreuse à se « suicider » en lui permettant de ré-exprimer les gènes suppresseurs de tumeurs.
Dans ce contexte, l’objectif de notre travail publié dans la revue J Am Chem Soc, a été de suivre et de caractériser la dynamique du basculement des cytosines méthylées sous l’effet du domaine SRA de la protéine UHRF1 qui reconnaît l’ADN hémi-méthylé [6]. Pour atteindre cet objectif, nous avons utilisé deux analogues fluorescents de nucléosides très innovants, la 2-thiényl-3-hydroxychromone (3HCnt) capable de substituer toutes les nucléobases de l’ADN [7] et la thiénoguanosine (thG) qui remplace parfaitement la guanine [8, 9]. En incorporant la 3HCnt ou la thG à des positions choisies d’un ADN double brin hémi-méthylé ou non méthylé, nous avons montré par spectroscopie de fluorescence qu’il était possible de suivre de manière sensible et fidèle le basculement des cytosines méthylées, induit par le domaine SRA de la protéine UHRF1 (Figure 2). En effet, le domaine SRA induit une forte augmentation de la fluorescence de ces deux sondes, indiquant la très grande sensibilité de ces analogues fluorescents de nucléosides aux changements structuraux mineurs accompagnant le basculement des bases [10]. Cette forte augmentation ne se produit que pour les séquences hémi-méthylées et non pour les séquences non méthylées correspondantes. En outre, cette forte augmentation de fluorescence est abolie quand le domaine SRA est remplacé par une protéine mutante incapable d’induire un basculement de base [10]. Par ailleurs, nous avons vérifié que (1) les analogues thG et 3HCnt n’affectent pas ou peu la stabilité du double brin d’ADN dans lequel ils sont insérés, et (2) qu’aux positions où ces sondes sont sensibles au basculement de base induit par SRA, ce domaine conserve une affinité préférentielle pour les séquences hémi-méthylées par rapport aux séquences non méthylées. Ces sondes ont permis également de déterminer par une technique de « stopped-flow 1, » que la liaison du domaine SRA avec l’ADN est rapide et donc compatible avec l’activité de lecture de l’UHRF1 pour trouver les sites à méthyler sur le brin d’ADN néo-synthétisé [6, 11]. En revanche, l’interaction de SRA avec la cytosine méthylée et le basculement de cette dernière dans la poche de liaison constituent un événement lent, d’une durée compatible avec le recrutement de la DNMT1 par l’UHRF1 à l’emplacement adéquat. À nouveau, cette cinétique lente disparaît quand le domaine SRA est remplacé par un motif mutant incapable d’induire un basculement de base. Nos résultats sont, par conséquent, en parfaite cohérence avec l’image de l’UHRF1 servant de guide à la méthyl-transférase DNMT1 pour identifier les cytosines à méthyler.
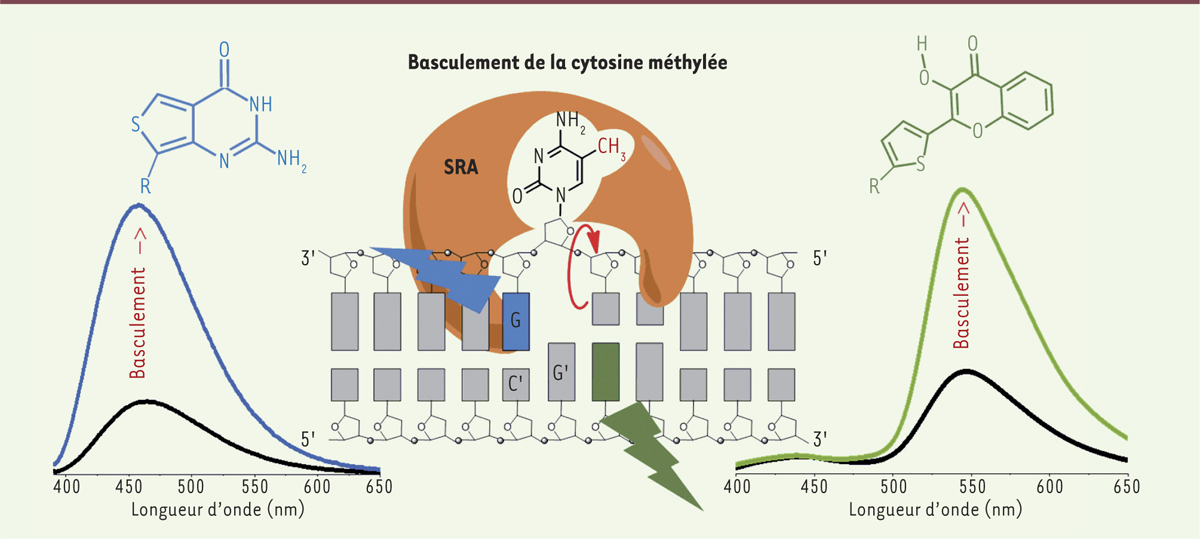
| Figure 2.
Suivi par fluorescence du basculement de la cytosine méthylée par le domaine SRA (set and ring associated) de la protéine UHRF1. L’ADN est marqué par un analogue nucléosidique fluorescent : la thiénoguanosine (thG, représentée en bleu) ou la 2-thiényl-3-hydroxychromone (3HCnt, représentée en vert). Au niveau du site mCpG, le domaine SRA fait basculer la cytosine méthylée, ce qui va stabiliser la liaison de ce domaine à l’ADN hémi-méthylé et permettre le recrutement de la protéine DNMT1. Le basculement de la cytosine méthylée à l’extérieur de l’hélice d’ADN se traduit par une forte augmentation de la fluorescence de la thG ou de la 3HCnt (figure adaptée de l’article de Kilin et al. [6]). DNMT1 : ADN méthyltransférase 1 ; UHRF1 : ubiquitin-like containing PHD and RING finger domains 1. |
Cette étude donne un nouvel éclairage sur le fonctionnement du tandem UHRF1/DNMT1 et illustre la complexité du mécanisme de transmission d’un caractère épigénétique. L’étape suivante est de comprendre comment UHRF1 dialogue avec la DNMT1 pour lui indiquer quelle est la cytosine à cibler afin de boucler le processus de duplication des profils de méthylation. La technologie mise en œuvre devrait également permettre d’envisager le développement de tests de criblage pour identifier des inhibiteurs du basculement de la cytosine méthylée.