| |
| Med Sci (Paris). 33(6–7): 603–606. doi: 10.1051/medsci/20173306015.Contrôle de la virulence de Salmonella enterica par la machinerie de biogenèse des centres Fe-S Alexis Carreaux,1*a Ségolène de Champs de Saint-Leger,1*b Yousra Kouidri,1*c and Marie-Pierre Golinelli-Cohen2d 1M1 Biologie Santé, Université Paris-Saclay, 91405Orsay, France 2Institut de chimie des substances naturelles, CNRS UPR2301, Université Paris-Sud, Université Paris-Saclay, 91190Gif-sur-Yvette, France |

Équipe pédagogique
Karim Benihoud (professeur, université Paris-Sud)
Sophie Dupré (maître de conférences, université Paris-Sud)
Olivier Guittet (maître de conférences, université Paris-Sud)
Boris Julien (maître de conférences, université Paris-Sud)
Hervé Le Stunff (professeur, université Paris-Sud)
Philippe Robin (maître de conférences, université Paris-Sud)
karim.benihoud@u-psud.fr
Série coordonnée par Laure Coulombel. La salmonellose, maladie infectieuse intestinale pouvant évoluer vers une septicémie ou une méningite, est causée par les entérobactéries de type Salmonella dont S. enterica serovar Typhimurium. Ces bactéries représentent encore aujourd’hui la principale cause d’intoxication alimentaire. Le cycle infectieux de S. enterica serovar Typhimurium débute par l’invasion des cellules épithéliales intestinales. Celle-ci nécessite l’activation de Spi1 (Salmonella pathogenicity island 1), un système de sécrétion de type III (TTSS ou type three secretion system) (voir Encadré) très finement régulé en réponse à des variations de signaux environnementaux [1, 2]. Chez les bactéries comme dans tous les organismes, les protéines Fe-S sont impliquées dans de nombreuses voies essentielles, et l’assemblage de ces centres métalliques fait intervenir deux voies de biogenèse, ISC (iron-sulfur cluster) et SUF (sulfur assimilation). Cette dernière est activée uniquement en condition de stress [3] (→).
(→) Pour une revue sur les centres Fe-S, voir la Synthèse de B. Py et F. Barras, m/s n° 12, décembre 2014, page 1110
Les centres Fe-S sont typiquement sensibles au stress oxydatif ainsi qu’à une carence en fer, conditions retrouvées notamment lors de l’infection de l’hôte par un organisme pathogène comme S. enterica. Or, depuis quelques années, beaucoup d’arguments plaident en faveur d’une implication des voies ISC et SUF dans la virulence de nombreuses bactéries, dont S. enterica [4]. Ainsi, récemment, Vergnes et al., une équipe française, ont clairement démontré un nouveau mécanisme moléculaire impliquant la protéine iron-sulfur cluster regulator (IscR), senseur de centre Fe-S [5]. |
Le système de sécrétion de type III (TTSS) Spi1 Ce système fonctionne comme une aiguille moléculaire qui permet d’injecter des protéines effectrices dans la cellule cible infectée. HilA, HilC, HilD et RtsA sont les principaux régulateurs du système. HilA est un membre de la famille des régulateurs transcriptionnels OmpR/ToxR ; ces molécules activent l’expression des gènes structuraux et des effecteurs de Spi1, qui semble être directement dépendante du niveau d’expression de HilA. Trois protéines homologues, RtsA, HilC and HilD, qui appartiennent à la famille des régulateurs transcriptionnels AraC/XylS, se lient au locus en amont de hilA et en stimulent l’expression. Selon le modèle actuellement admis, HilD est le principal responsable du contrôle de l’expression du système car son expression va activer la transcription de HilC et RtsA, qui s’autorégulent et se régulent aussi entre eux. Ces trois activateurs induisent alors l’expression de hilA et, par conséquent, de l’ensemble de Spi1.
|
|
La machinerie ISC est un régulateur majeur du facteur de virulence Spi1 chez S. enterica Dans un premier temps, les auteurs ont évalué la contribution respective des machineries ISC et SUF dans la prolifération de S. enterica dans différents contextes cellulaires en introduisant une mutation dans l’une ou l’autre des deux voies de biogenèse des centres Fe-S de cette bactérie. Ils ont alors observé que seule la voie ISC est requise pour l’infection in vitro de la lignée cellulaire épithéliale modèle HeLa, ou in vivo de la souris. Or, l’expression du facteur de virulence Spi1 est régulée positivement par la protéine HilA. De plus, HilD (voir encadré 1), qui fait elle-même partie du TTSS Spi1, régule positivement sa propre expression ainsi que celle d’HilA et donc de Spi1 lui-même [2]. En utilisant le gène gfp (codant la green fluorescent protein) placé sous le contrôle du promoteur d’hilD (PhilD) dans les mêmes souches que celles précédemment utilisées, il apparaît que la machinerie ISC est nécessaire à l’activation de l’expression d’hilD et donc de Spi1. Ainsi, la machinerie ISC est un régulateur majeur de la synthèse du facteur de virulence Spi1. |
IscR réprime l’expression d’HilD et de ses cibles indépendamment de la présence de son centre Fe-S IscR est une protéine Fe-S de la machinerie ISC qui agit dans la cellule comme un senseur de centre Fe-S et permet la régulation concertée de l’expression d’ISC et SUF. En situation de carence en fer ou en présence d’espèces réactives de l’oxygène (ERO), IscR perd son centre Fe-S (forme apo) et active l’opéron suf. Inversement, en présence de fer, IscR possède son centre Fe-S (forme holo) et réprime l’expression d’ISC et donc sa propre expression [6]. L’analyse bioinformatique de PhilD montre qu’il possède deux sites de liaison putatifs pour IscR, dont un se superpose partiellement au site de fixation de HilD. La fixation de deux dimères d’IscR sur PhilD a été démontrée par des expériences de retard sur gel et par interférométrie à l’aide de la protéine purifiée. La spécificité de la fixation d’IscR sur le promoteur de hilD (PhilD) a ensuite été confirmée par mutagenèse du promoteur. De nouveau, en utilisant gfp comme gène rapporteur, les auteurs montrent qu’IscR agit comme un régulateur négatif de l’expression d’hilD et, par conséquent, des gènes régulés par HilD dont Spi1. De manière intéressante, la surexpression de la forme holo d’IscR (contenant un centre Fe-S) ou d’une forme mutante incapable d’assembler un centre Fe-S (apo-IscR) conduit aux mêmes observations. Ces observations montrent que IscR réprime l’expression d’hilD et des gènes de Spi1 indépendamment de la présence de son centre Fe-S. |
IscR contrôle la régulation, dépendante du fer, des gènes du TTSS Spi1 Des études précédentes avaient montré que l’expression de l’opéron Spi1 était réprimée lors d’une carence en fer. Or, lorsque la bactérie est mutée pour IscR (absence d’expression de la protéine) et carencée en fer, l’expression de TTSS Spi1 est moins efficacement régulée. IscR semble donc être un intermédiaire dans la régulation de l’expression de Spi1 par le fer. Pour finir, les auteurs ont montré que l’invasion par S. enterica mutée pour IscR était augmentée dans le cas d’infection de cellules épithéliales in vitro, mais aussi lors d’une infection de la souris par la bactérie. Cependant, dans ce dernier cas, la virulence des bactéries mutées était diminuée par rapport à celle des bactéries sauvages, ce qui pourrait s’expliquer par l’implication d’IscR dans la régulation d’autres gènes liés à la pathogénicité de la bactérie. Donc, alors que la fixation de IscR sur Phild – gène qui contrôle la régulation de Spi1 - ne requiert pas le centre Fe-S, ce dernier intervient néanmoins dans l’invasion bactérienne. Comment ? |
Les protéines Fe-S, nouveaux régulateurs majeurs de la virulence bactérienne Cette étude démontre très clairement le mécanisme moléculaire de la régulation de l’expression du facteur de virulence Spi1 de S. enterica par IscR, protéine Fe-S qui agit comme régulateur transcriptionnel des voies de biogenèse des centres Fe-S. Les auteurs démontrent qu’il y a compétition entre IscR et HilD pour la fixation au promoteur du gène hilD, dont IscR régule négativement l’expression, indépendamment de la présence d’un centre Fe-S dans IscR. C’est la concentration de IscR elle-même, et donc sa régulation, qui sont alors déterminantes pour le contrôle de la virulence. Or, en présence de fer et en l’absence de stress, IscR possède un centre Fe-S et régule négativement la voie ISC. La concentration de IscR diminue donc, et celui-ci n’entre plus en compétition avec HilD. Celle-ci peut alors se lier à son propre promoteur et réguler positivement son expression et, par conséquent, celles de HilA et de Spi1. Inversement, en absence de fer et/ou en présence de stress, IscR perd son centre Fe-S et ne régule plus la voie ISC. Sa concentration restant élevée, IscR se lie au promoteur de hilD et le régule négativement (Figure 1). Ainsi, Salmonella utilise IscR pour moduler son infectiosité en fonction de la disponibilité en fer et en oxygène : elle favorise l’invasion des tissus à faible teneur en oxygène et forte concentration en fer comme l’intestin.
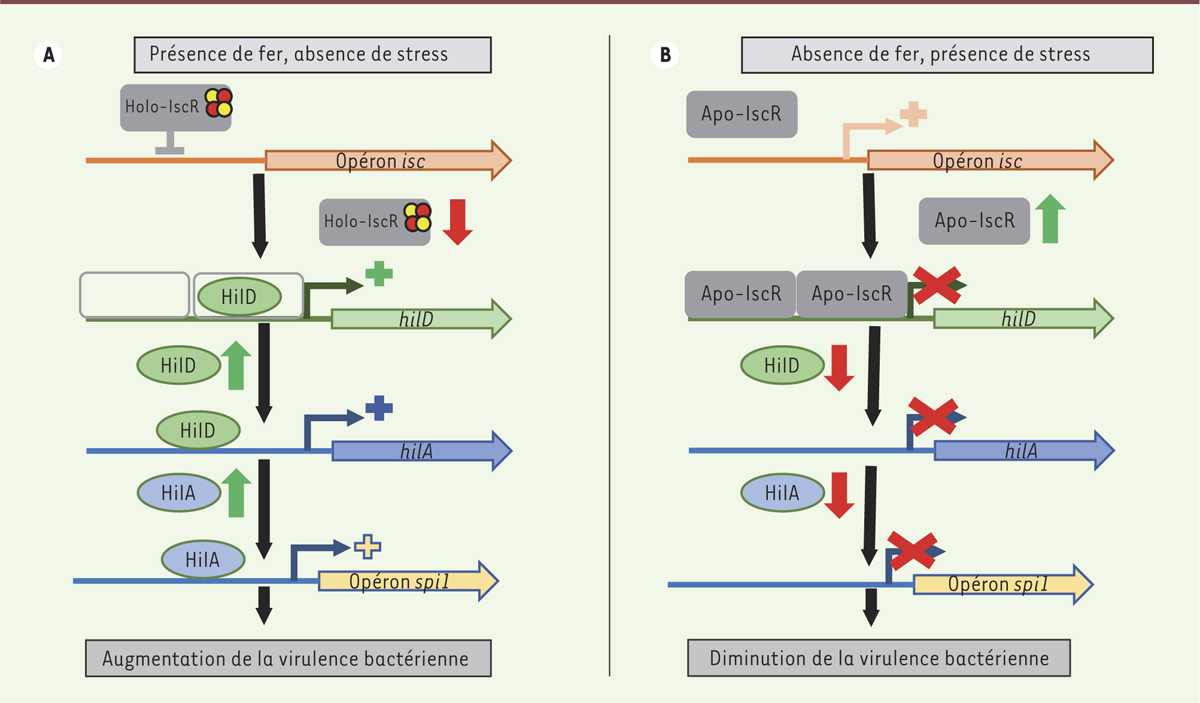
 | Figure 1.
Mécanisme moléculaire de la régulation de la virulence bactérienne de S. enterica par IscR, un senseur de Fe-S. A. En présence de fer et absence de stress, IscR possède un centre Fe-S (holo-IscR). Il réprime alors l’expression de l’opéron isc, ce qui diminue la concentration en IscR. Ainsi, HilD peut se fixer sur son propre promoteur et en réguler positivement l’expression. La concentration en HilD augmente, ce qui va réguler positivement l’expression de HilA et donc de Spi1. Il en résulte une hausse de la virulence bactérienne. B. Inversement, en absence de fer ou en présence d’un stress oxydatif, IscR perd son centre Fe-S (apo-IscR). Cette forme ne réprime pas l’opéron isc, la concentration en IscR augmente alors. Apo-IscR se fixe sur le promoteur d’hilD et en réprime l’expression. Par conséquent, la quantité en HilD diminue et les synthèses d’HilA et donc de Spi1 ne sont plus activées. La virulence bactérienne diminue alors. |
Récemment, il a été montré qu’un autre régulateur majeur de l’homéostasie du fer chez la bactérie, la protéine Fur (ferric uptake regulator), est aussi capable de se lier au promoteur d’hilD en présence de fer (forme Fur-Fe2+) et de réguler positivement son expression [7]. Ainsi, chez S. enterica, les systèmes Fur et IscR régulent l’expression du facteur de virulence Spi1 en fonction de la disponibilité en fer. De manière très intéressante, IscR est aussi impliqué dans la régulation de la virulence d’une autre bactérie pathogène, Yersinia, mais, dans ce cas, IscR la régule de manière positive [8]. Ainsi, ces deux pathogènes utilisent IscR de manière opposée pour réguler leur virulence. Pour finir, chez Staphylococcus aureus, c’est le système à deux composants AirSR qui contrôle finement la virulence. Or, la kinase AirS est une protéine Fe-S dont l’activité (qui se traduit in fine dans la virulence de la bactérie) est contrôlée par l’état d’oxydation de son centre Fe-S [9]. On voit ainsi que différentes bactéries pathogènes utilisent diverses stratégies faisant intervenir des protéines Fe-S et/ou les voies de biogenèse de ces agrégats métalliques afin de rapidement coordonner la production de facteurs de virulence en réponse à des variations de la concentration en fer ou en oxygène chez l’hôte. Les protéines Fe-S apparaissent ainsi comme de nouvelles cibles potentielles pour le développement de molécules antibactériennes comme récemment démontré pour S. aureus [10]. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Que F, Wu S, Huang R. Salmonella pathogenicity island 1(SPI-1) at work . Curr Microbiol. 2013; ; 66 : :582.–587. 2.
Ellermeier JR, Slauch JM. Adaptation to the host environment: regulation of the SPI1 type III secretion system in Salmonella enterica serovar Typhimurium . Curr Opin Microbiol. 2007; ; 10 : :24.–29. 3.
Py B, Barras F. Du fer et du soufre dans les protéines. Comment la cellule construit-elle les cofacteurs fer-soufre essentiels à son fonctionnement ? Med Sci (Paris). 2014;; 30 : :1110.–1122. 4.
Velayudhan J, Karlinsey JE, Frawley ER, et al. Distinct roles of the Salmonella enterica serovar Typhimurium CyaY and YggX proteins in the biosynthesis and repair of iron-sulfur clusters . Infect Immun. 2014; ; 82 : :1390.–1401. 5.
Vergnes A, Viala JP, Ouadah-Tsabet R, et al. The iron-sulfur cluster sensor IscR is a negative regulator of Spi1 type III secretion system in Salmonella enterica . Cell Microbiol. 2016;; 19 : doi: 10.1111/cmi.12680. 6.
Santos JA, Pereira PJ, Macedo-Ribeiro S. What a difference a cluster makes: The multifaceted roles of IscR in gene regulation and DNA recognition . Biochim Biophys Acta. 2015; ; 1854 : :1101.–1112. 7.
Teixido L, Carrasco B, Alonso JC, et al. Fur activates the expression of Salmonella enterica pathogenicity island 1 by directly interacting with the hilD operator in vivo and in vitro . PLoS One. 2011; ; 6 : :e19711.. 8.
Miller HK, Kwuan L, Schwiesow L, et al. IscR is essential for yersinia pseudotuberculosis type III secretion and virulence . PLoS Pathog. 2014; ; 10 : :e1004194.. 9.
Golinelli-Cohen MP, Bouton C. Fe-S proteins acting as redox switch: new key actor of cellular adaptative responses . Curr Chem Biol. 2017 (sous presse).. 10.
Choby JE, Mike LA, Mashruwala AA, et al. A small-molecule inhibitor of iron-sulfur cluster assembly uncovers a link between virulence regulation and metabolism in staphylococcus aureus . Cell Chem Biol. 2016; ; 23 : :1351.–1361. |