Les systèmes vivants sont régis par un jeu d’événements biologiques dynamiques finement orchestrés de l’échelle moléculaire à celle de l’organisme entier. Comprendre les mécanismes complexes contrôlant les cellules et les organismes nécessite des méthodes d’imagerie efficaces pour observer et quantifier les biomolécules en temps réel avec une grande résolution spatiotemporelle. L’imagerie biologique a bénéficié ces dernières décennies d’interactions synergiques entre l’instrumentation physique et l’élaboration de sondes moléculaires permettant de visualiser les biomolécules en action. La microscopie de fluorescence est de nos jours la méthode de choix pour réaliser des images des systèmes vivants. Permettant d’observer les constituants cellulaires et tissulaires avec une très grande sensibilité et une très haute résolution spatiale et temporelle, cette technique a été indispensable à notre compréhension actuelle des processus biologiques.
La découverte de la protéine fluorescente verte GFP (green fluorescent protein) dans la méduse Aequorea victoria, pour laquelle les chercheurs Osamu Shimomura, Martin Chalfie et Roger Y. Tsien ont reçu le prix Nobel de chimie en 2008, a révolutionné l’imagerie biomoléculaire [1] (→). Pour la première fois, il était possible d’étudier la dynamique des protéines dans des systèmes vivants simplement en fusionnant génétiquement la séquence codant la protéine GFP à celle d’une protéine d’intérêt. La découverte, puis l’ingénierie de nouvelles protéines autofluorescentes, ont facilité le développement de l’imagerie multi-couleur, la conception de biosenseurs, et ont contribué à l’essor de nouvelles techniques de microscopie comme la microscopie de super-résolution [2,3].
(→) Voir la Nouvelle de H. Pasquier, m/s n° 11, novembre 2008, page 985
Un nombre grandissant d’études a cependant révélé que les protéines auto-fluorescentes n’étaient pas toujours des sondes optimales. Leur taille et leur tendance naturelle à oligomériser conduisent parfois à des protéines de fusion non fonctionnelles. De plus, la maturation de leur chromophore nécessite la présence de dioxygène empêchant leur utilisation en conditions anaérobiques. Ce processus de maturation est en outre un mécanisme relativement lent (pouvant durer de quelques dizaines de minutes à quelques heures selon les protéines), limitant la résolution temporelle de certaines expériences [4]. Enfin, certaines protéines fluorescentes possèdent des propriétés photophysiques/photochimiques inattendues qui compliquent l’interprétation quantitative des expériences [4].
L’importance des protéines fluorescentes pour l’imagerie biologique a motivé les chimistes et les biologistes à développer des stratégies alternatives pour rendre fluorescentes les protéines. Une approche qui a été explorée consiste à utiliser des chromophores fluorogéniques (appelés également fluorogènes) qui sont non-fluorescents s’ils sont libres en solution, mais le deviennent lorsqu’ils interagissent avec une cible donnée [5]. Des étiquettes protéiques, capables d’interagir avec ces fluorogènes et d’activer leur fluorescence, ont été développées afin de pouvoir génétiquement marquer des protéines cellulaires d’intérêt. Ces fluorogènes ne sont fluorescents que lorsqu’ils interagissent avec leur étiquette protéique : leur excès est donc quasiment invisible, assurant un excellent contraste lors de l’imagerie. En utilisant ce principe générique, des protéines fluorescentes - telles que la FbFP (flavin-based fluorescent protein) [6], le iLOV (improved light, oxygen, or voltage domain) [7], le miniSOG (mini singlet oxygen generator) [8], la UnaG (une protéine isolée de l’anguille japonaise) [9] - complexant des fluorogènes naturels tels que les flavines ou la bilirubine ont récemment été proposées comme alternatives à la protéine GFP. Ces protéines sont plus petites que la GFP et, à la différence de cette dernière, présentent une fluorescence indépendante de la présence de dioxygène. Des protéines fluorescentes dans le rouge lointain ou le proche infrarouge à base de biliverdine - par exemple, la IFP1.4 (infrared fluorescent protein 1.4), la iRFP (near infrared fluorescent protein) ou la smURFP (small ultrared fluorescent protein) - ont ouvert de nouvelles possibilités pour l’imagerie in vivo. En effet, l’utilisation d’excitations infrarouges permet d’effectuer une imagerie plus en profondeur dans les tissus. D’autres développements intéressants incluent l’utilisation de systèmes de marquage (tels que SNAP-tag [10], PYP-tag [11], CRABPII [12] et FAP [13])1 composés d’une étiquette protéique interagissant de manière covalente ou non-covalente avec un fluorogène synthétique exogène. À la différence des protéines fluorescentes exploitant des fluorogènes naturels endogènes, la capacité d’additionner un composé exogène ouvre de nouvelles opportunités pour des applications « à la demande », quand la fluorescence est souhaitée seulement à un temps et/ou une densité donnés.
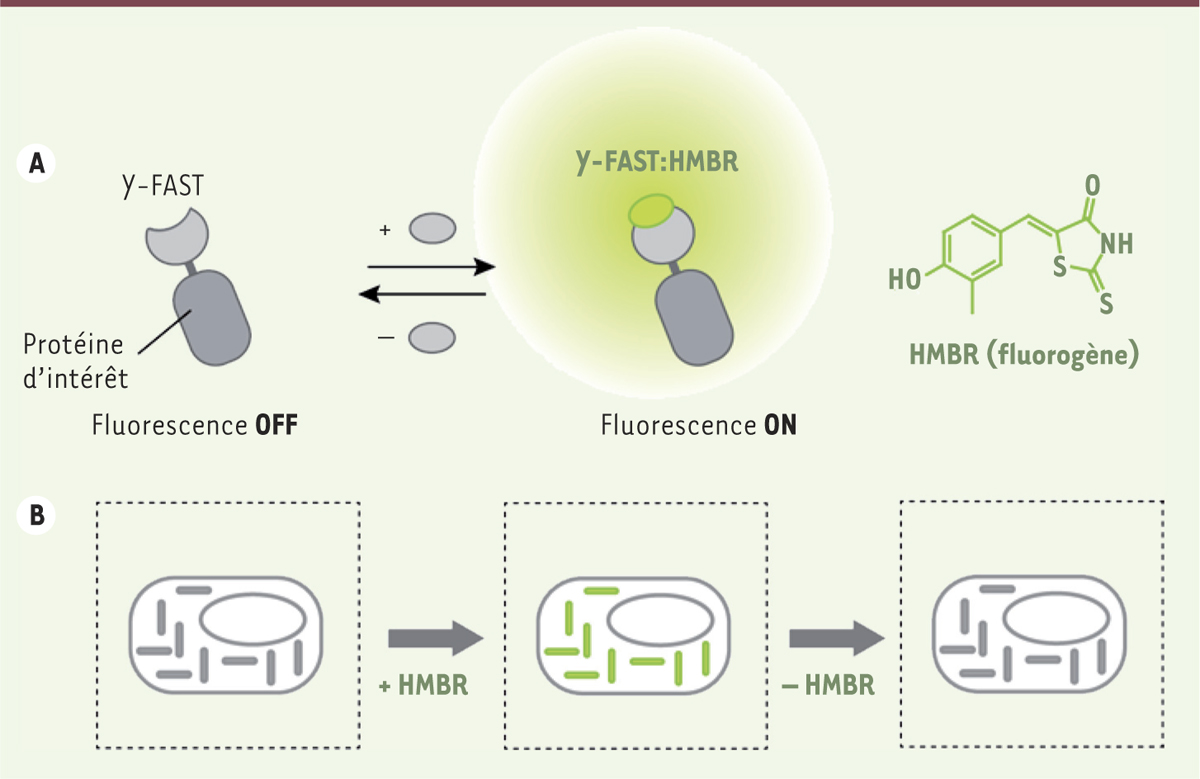
Nous avons récemment étendu ce concept en développant une protéine fluorescente hybride composée d’une partie protéique, appelée Y-FAST (yellow fluorescence-activating and absorption-shifting tag), et d’un fluorogène synthétique (4-hydroxy-3-méthylbenzylidène-rhodanine, HMBR) [14]. Y-FAST est un variant de la protéine photo-activable jaune PYP, photorécepteur naturel monomérique de 14 kDa trouvé chez Halorhodospira halophila, conçu par évolution dirigée2 pour complexer le fluorogène HMBR de manière réversible et activer sa fluorescence. Le fluorogène synthétique HMBR est non-fluorescent en solution. Il émet une fluorescence jaune (λem = 540 nm) sous excitation en lumière bleue lorsqu’il est complexé à la protéine Y-FAST.
Y-FAST est comparable aux protéines autofluorescentes classiques en termes de brillance et de photostabilité et permet, comme ces dernières, de marquer des protéines dans différentes organelles et dans une grande variété de systèmes. Son application est possible dans les cellules de mammifères (par exemple les cellules des lignées HeLa3 ou HEK [human embryonic kidney] et les neurones), les microorganismes et les organismes multicellulaires (tels que les embryons de poisson zèbre). Y-FAST se distingue des protéines autofluorescentes par (1) sa petite taille (deux fois plus faible que celle de la GFP) qui permet de minimiser la perturbation fonctionnelle introduite lors de la fusion à une protéine d’intérêt, et (2) son caractère monomérique, qui permet d’éviter des phénomènes d’oligomérisation dans les environnements cellulaires denses et compacts (par exemple les membranes) ou dans les fusions multi-modulaires (telles que les biosenseurs).
À la différence des protéines autofluorescentes qui ne sont fluorescentes qu’après la maturation post-traductionnelle de leur chromophore, l’apparition de la fluorescence d’Y-FAST (en présence de fluorogène) est quasi instantanée après son repliement et ne nécessite aucun autre additif. Cette propriété permet de suivre la synthèse de protéines en temps quasi réel et ouvre de nouvelles opportunités, telles que (1) la visualisation de processus rapides impliquant l’activation précoce de promoteurs, (2) le marquage de protéines à courte durée de vie, ou (3) le suivi d’événements de traduction ou de repliement de protéines en temps quasi réel. Comme de plus, le processus d’activation de la fluorescence ne nécessite pas la présence de dioxygène, la protéine Y-FAST permet, à la différence des protéines autofluorescentes, le marquage fluorescent de protéines en conditions anaérobiques.
La sonde fluorescente Y-FAST se distingue des autres méthodes de marquage car l’interaction avec son fluorogène est non-covalente et réversible (Figure 1). Cette caractéristique permet ainsi d’allumer ou d’éteindre la fluorescence de la protéine Y-FAST « à la demande » par ajout ou retrait (par exemple par lavage) du fluorogène synthétique. Grâce à cette possibilité de marquage réversible, il est possible d’envisager : (1) un marquage fluorescent transitoire pour libérer ensuite des canaux d’observation, (2) la régénération des fluorogènes après leur photodégradation prolongeant ainsi la durée du signal fluorescent, (3) le contrôle du niveau de fluorescence : la concentration de fluorogène ajouté permet d’ajuster l’intensité de fluorescence aux conditions expérimentales.
| Figure 1.
Y-FAST : une sonde fluorescente hybride pour l’imagerie « à la demande » des protéines intracellulaires. (A) Y-FAST (yellow fluorescence-activating and absorption-shifting tag) est une protéine fluorescente hybride composée d’une étiquette protéique et d’un fluorogène synthétique (4-hydroxy-3-méthylbenzylidène-rhodanine ; HMBR). Le fluorogène fluoresce (ON) lorsqu’il interagit avec l’étiquette protéique, assurant ainsi un fort contraste. (B) L’interaction entre l’étiquette protéique et le fluorogène est non-covalente et réversible, permettant ainsi de contrôler à souhait l’intensité de fluorescence par ajout (ON) ou retrait du fluorogène (OFF). |
Enfin, la méthode utilisée pour le développement d’Y-FAST est générique et devrait permettre de concevoir des systèmes possédant des propriétés chromatiques et physico-chimiques variées. La possibilité de modifier les propriétés du système par ingénierie à la fois de la structure du fluorogène et de la séquence de l’étiquette protéique ouvre la voie à la génération de marquages sélectifs (tels celui de protéines exposées à la membrane extracellulaire). Il est également possible d’envisager la création d’une collection de marqueurs fluorescents de couleurs variées, couvrant l’ensemble du spectre visible.
En conclusion, dans les prochaines années, va probablement se développer une large collection de systèmes hybrides possédant des caractéristiques de plus en plus innovantes. Ces systèmes seront à la fois des sondes moléculaires pour la localisation et la quantification de biomolécules, ou des biosenseurs capables de fournir des informations sur l’état physiologique des cellules et des organismes. Combinés avec les microscopies optiques les plus avancées, ces technologies pourraient avoir un impact important sur notre connaissance et notre compréhension de l’organisation spatiotemporelle des cellules et des organismes vivants.