| |
| Med Sci (Paris). 33(6–7): 573–576. doi: 10.1051/medsci/20173306005.Mise en évidence de la physiologie rythmique du foie par protéomique nucléaire Daniel Mauvoisin1,2 and Frédéric Gachon1,3* 1Diabetes and circadian rhythms department, Nestlé institute of health sciences, CH-1015Lausanne, Suisse 2Institute of bioengineering, School of life sciences, École polytechnique fédérale de Lausanne, CH-1015Lausanne, Suisse 3School of life sciences, École polytechnique fédérale de Lausanne, CH-1015Lausanne, Suisse |
Les balbutiements de la protéomique circadienne Le rôle de l’horloge circadienne dans la régulation des rythmes d’expression des gènes a été intensivement étudié au niveau transcriptionnel [1] (→).
(→) Voir la Synthèse de H. Duez et al., m/s n° 8-9, août-septembre 2013, page 772
Cependant, jusqu’à récemment, nous n’avions que très peu d’informations concernant la régulation rythmique au niveau traductionnel et son impact sur l’accumulation des protéines. Si de nombreux travaux suggéraient que des mécanismes post-transcriptionnels contribuaient à la génération de rythmes circadiens au niveau protéique [2], ce n’est que récemment qu’une régulation de la traduction a été démontrée [3, 4]. Des études pionnières, utilisant l’électrophorèse en deux dimensions, avaient permis de quantifier quelques dizaines de protéines rythmiques au niveau hépatique, dont une grande partie était codée par des ARN messagers (ARNm) non rythmiques, soulignant l’implication de mécanismes post-transcriptionnels dans leur régulation [5]. |
La protéomique circadienne à grande échelle Afin d’améliorer la résolution de ces études protéomiques et pour découvrir de nouvelles fonctions biologiques régulées par l’horloge biologique ou les rythmes alimentaires, nous avons utilisé une technique de spectrométrie de masse quantitative à haute résolution incluant un marquage in vivo par des isotopes non radioactifs (SILAC, stable isotope labeling with amino acids in cell culture) [6] et évalué le profil protéique hépatique journalier chez la souris. Nous avons ainsi quantifié pas moins de 5 000 protéines parmi lesquelles 6 % présentaient un profil d’accumulation rythmique. En comparant les rythmes d’accumulation des ARNm correspondants à ce protéome rythmique, nous avons mis en évidence que seule la moitié de celui-ci était effectivement codée par des ARNm rythmiques. L’analyse ontologique de ce premier groupe de protéines révèle que celui-ci est impliqué dans les fonctions du foie, comme le métabolisme lipidique (en phase diurne) ou comme celui du glucose et des xénobiotiques (en phase nocturne). L’autre moitié du protéome rythmique codé par des ARNm non rythmiques est fortement enrichie en protéines qui sont sécrétées. Ce groupe inclut, notamment, l’albumine et les serpines1, qui présentent un rythme d’accumulation synchrone dans le plasma. Le rythme de ce groupe de protéines persiste chez les souris déficientes pour les gènes régulant l’horloge circadienne et nourries uniquement en phase nocturne. Il disparait pendant le jeûne. Cela suggère que des signaux d’entraînement reliés à l’alimentation influencent le rythme d’expression de ces protéines et plus généralement des facteurs circulant dans le plasma [7]. |
L’avènement de la protéomique circadienne à l’échelle subcellulaire L’analyse d’extraits protéiques provenant de cellules entières reste toujours difficile malgré les progrès technologiques récents. La quantification des protéines faiblement exprimées, comme les facteurs de transcription, reste complexe. Pour contourner cet inconvénient, nous avons effectué une analyse de protéomique et de phosphoprotéomique2 quantitative sur des noyaux provenant d’hépatocytes purifiés. Une approche similaire a été récemment utilisée pour les mitochondries [8]. Nous avons combiné l’approche in vivo SILAC précédemment utilisée [7] et l’enrichissement de noyaux, pour quantifier plus de 4 000 protéines nucléaires. Le taux de détection des protéines nucléaires du foie est alors sans précédent, puisque 70 % du protéome nucléaire connu selon UNIPROT3 est représenté dans nos données, ainsi que 90 % des composants des compartiments nucléaires connus. Une analyse de rythmicité très sélective (présentant un taux inférieur à 5 % de faux positifs – dont l’expression est faussement attribué au rythme) de ce protéome nucléaire révèle qu’environ 10 % des protéines présentent une accumulation rythmique. Cependant, ce taux de protéines rythmiques atteint 45 % dès lors que nous utilisons les mêmes critères de sélection que ceux de l’étude précédente [7], évoquant une régulation diurne importante au niveau nucléaire. Ces protéines rythmiques présentent des maximums d’accumulation en fin de journée et de nuit. En comparant les profils d’accumulation des ARNm correspondant à ce protéome nucléaire rythmique, on s’aperçoit que seulement le quart de ces protéines est codé par des ARNm rythmiques, suggérant une importante régulation post-transcriptionnelle, voire post-traductionnelle [9]. |
Les complexes protéiques présentent des accumulations nucléaires rythmiques et mettent en évidence la coordination temporelle de processus cellulaires fondamentaux La plupart des protéines nucléaires détectées appartiennent à des complexes protéiques. Beaucoup de sous-unités de complexes connus, rythmiques ou non, présentent en effet des rythmes d’accumulation synchrones dans le noyau. Il est donc probable que la régulation diurne du transport nucléaire de ces complexes soit un élément important de la régulation du protéome nucléaire rythmique. La rythmicité de ces complexes a été décomposée en valeurs singulières. Le résultat de cette analyse montre que les complexes exprimés le jour sont plutôt impliqués dans la réparation de l’ADN et la régulation transcriptionnelle, tandis que les complexes exprimés de nuit sont impliqués dans la régulation de l’organisation du cytosquelette, du transport protéique et de la protéolyse. Ainsi, en accord avec des travaux précédents portant notamment sur la synthèse rythmique des protéines et des ARN ribosomaux (ARNr) [3, 4], nous avons montré que la majorité des protéines impliquées dans les différentes étapes de la biogenèse des ribosomes s’accumulait de façon séquentielle. Celles-ci représentent environ 20 % du protéome nucléaire rythmique. Ces résultats constituent un exemple supplémentaire du rôle fondamental de l’horloge biologique dans la régulation temporelle de processus biologiques énergivores, même au niveau nucléaire [9]. |
Phosphoprotéome rythmique et activités des protéine-kinases associées Afin d’obtenir plus d’informations sur la dynamique du protéome diurne, nous avons également analysé sa régulation au niveau post-traductionnel. Nous avons ainsi évalué le phosphoprotéome rythmique et analysé les activités kinases associées. Nous avons quantifié près de 1 500 sites phosphorylés, dont 10 % présentaient un profil rythmique. Deux tiers de ces sites étaient situés sur des protéines nucléaires non rythmiques et montraient une répartition d’accumulation en fin de journée et de nuit. Ces données nous ont permis de déduire la rythmicité de 39 motifs consensus de kinases, dont une grande partie est impliquée dans la régulation du cycle cellulaire [9]. |
Orchestration rythmique de la transcription nucléaire Parmi les protéines et les motifs phosphorylés rythmiques nucléaires, nous avons identifié de nombreux facteurs de transcription. Nos données montrent, en effet, que 99 facteurs et 80 co-régulateurs de transcription nucléaires sont rythmiques. Parmi ceux-ci, 16 facteurs et 17 co-régulateurs de transcription conservent leur rythme chez des souris dépourvues d’horloge circadienne. Ils sont donc principalement régulés par des signaux d’entraînement indépendants de l’horloge moléculaire, tels que le rythme alimentaire [9], comme c’est le cas pour la protéine SREBP1 (sterol regulatory element binding protein 1) impliquée dans la régulation du métabolisme des lipides. |
La polyploïdie hépatique orchestrée de manière rythmique Les différentes analyses réalisées ont montré l’existence d’évènements qui sont relatifs au cycle cellulaire comme l’activité de nombreuses kinases et de facteurs de transcription, liés au cycle cellulaire. Cela est en accord avec des travaux précédents portant sur le lien entre le cycle cellulaire et l’horloge circadienne [10]. Ainsi, il semblerait que les cycles cellulaire et circadien soient alignés, bien que le foie soit un organe principalement quiescent. Le foie étant un tissu sujet à la polyploïdie [11], nous avons examiné si un lien pouvait éventuellement exister entre le cycle circadien et la polyploïdie. Nous avons donc analysé la quantité d’ADN présente dans les noyaux et observé une proportion anti-phasique des noyaux diploïdes, comparés aux noyaux tétraploïdes. Indépendamment, nous avons également évalué, par analyse de coupes histologiques, la proportion des différentes populations hépatocytaires. Nous avons constaté que la proportion des hépatocytes binucléés diploïdes (2 x 2N) est anti-phasique par rapport à celle des hépatocytes mononucléés tétraploïdes (1 x 4N). Cette polyploïdie rythmique pourrait donc être la conséquence d’un lien entre les cycles cellulaire et diurne [9]. Ces différents phénomènes sont résumés dans la
Figure 1
.
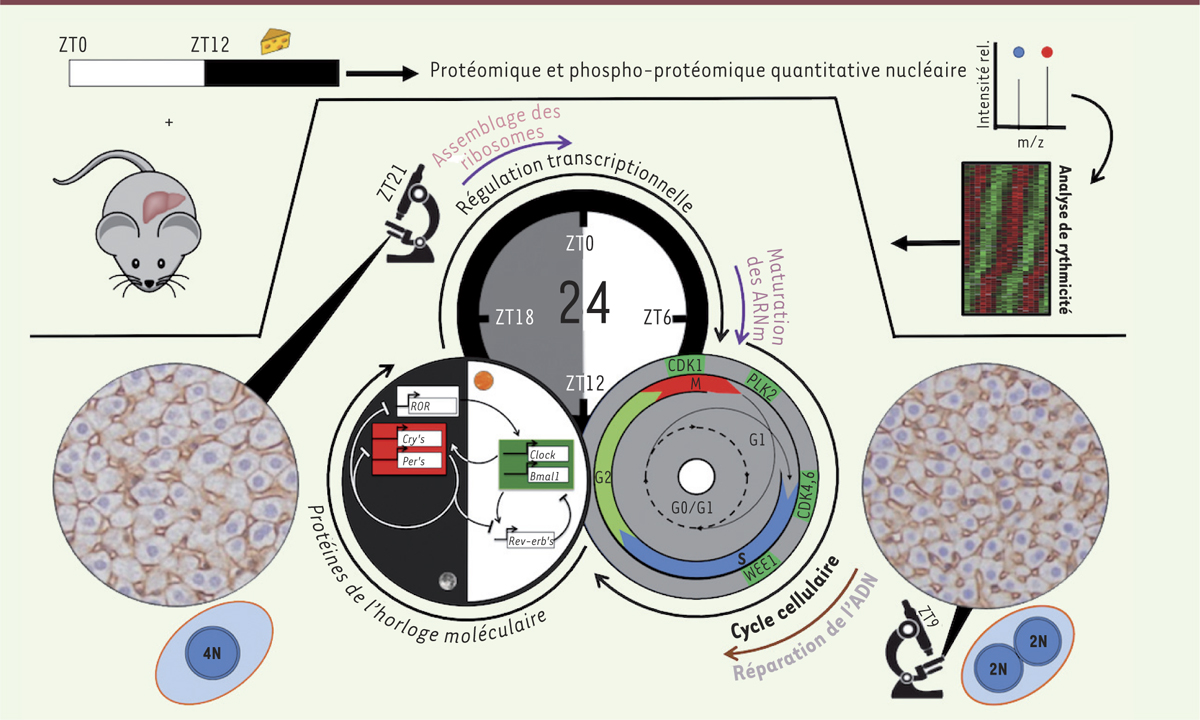
 | Figure 1.
Orchestration de la physiologie rythmique du foie mise en évidence par protéomique nucléaire quantitative. La combinaison de l’analyse quantitative et temporelle du protéome et du phosphoprotéome nucléaire in vivo a permis de mettre en évidence la rythmicité de nombreux processus cellulaires fondamentaux tels que la régulation transcriptionnelle, l’assemblage des ribosomes et la réparation de l’ADN. Les cycles cellulaire et diurne (imagés en noir et blanc) semblent alignés et la polyploïdie rythmique que nous observons serait une conséquence de ce lien. La quantification de l’intensité relative des différents isotopes est symbolisée en haut à droite (m/Z représente le rapport de la masse [m] sur la charge [Z]). À la suite de la quantification du protéome en fonction du temps, sa rythmicité est ensuite évaluée (symbolisée par la carte thermique). Les gènes codants les protéines de l’horloge moléculaire quantifiées dans [ 9] sont indiqués. Cry’s : c ryptochrome1 et cryptochrome2 ; Pers’s : period1 et period2 ; Clock : circadian locomotor output cycles kaput ; Bmal1 : brain and muscle aryl hydrocarbon receptor nuclear translocator-like protein-1 ; Rev-erbs : Rev-erb-α ou Nr1d1 : nuclear receptor subfamily 1, group D, member 1 ; Rev-erb-β ou Nr1d2 : nuclear receptor subfamily 1, group D, member 2) ; RORα/γ : RAR-related orphan receptor alpha/ gamma. Les protéines du contrôle du cycle cellulaire sont les kinases WEE1 et polo-kinase -2 (PLK2) et les kinases dépendantes des cyclines ou CDK1, 4, 6. ZT : zeitgeber time ou temps donné. |
|
La combinaison de l’analyse quantitative et temporelle du protéome et du phosphoprotéome nucléaires in vivo constitue une approche puissante pour découvrir de nouvelles fonctions biologiques rythmiquement orchestrées et qui sont non identifiables sur des extraits cellulaires totaux. Les données obtenues représentent une étape importante vers l’identification de nouvelles fonctions biologiques orchestrées par l’horloge circadienne et/ou les rythmes alimentaires. La purification d’organelles associée à la protéomique quantitative in vivo est une nouvelle voie prometteuse, non seulement pour la biologie circadienne, mais également pour l’étude de nombreuses fonctions dans divers modèles et systèmes dont les mécanismes moléculaires sont encore peu et mal décrits. |
Daniel Mauvoisin et Frédéric Gachon sont employés de Nestlé Institute of Health Sciences SA.
|
Footnotes |
1.
Duez H, Sebti Y, Staels B. Horloges circadiennes et métabolisme : intégration des signaux métaboliques et environnementaux . Med Sci (Paris). 2013; ; 29 : :772.–777. 2.
Doherty CJ, Kay SA. Circadian control of global gene expression patterns . Annu Rev Genet. 2010; ; 44 : :419.–444. 3.
Atger F, Gobet C, Marquis J, et al. Circadian and feeding rhythms differentially affect rhythmic mRNA transcription and translation in mouse liver . Proc Natl Acad Sci USA. 2015; ; 112 : :E6579.–E6E88. 4.
Jouffe C, Cretenet G, Symul L, et al. The circadian clock coordinates ribosome biogenesis . PLoS Biol. 2013; ; 11 : :e1001455.. 5.
Reddy AB, Karp NA, Maywood ES, et al. Circadian orchestration of the hepatic proteome . Curr Biol. 2006; ; 16 : :1107.–1115. 6.
Krüger M, Moser M, Ussar S, et al. SILAC Mouse for quantitative proteomics uncovers kindlin-3 as an essential factor for red blood cell function . Cell. 2008; ; 134 : :353.–364. 7.
Mauvoisin D, Wang J, Jouffe C, et al. Circadian clock-dependent and -independent rhythmic proteomes implement distinct diurnal functions in mouse liver . Proc Natl Acad Sci USA. 2014; ; 111 : :167.–172. 8.
Neufeld-Cohen A, Robles MS, Aviram R, et al. Circadian control of oscillations in mitochondrial rate-limiting enzymes and nutrient utilization by PERIOD proteins . Proc Natl Acad Sci USA. 2016; ; 113 : :E1673.–E1E82. 9.
Wang J, Mauvoisin D, Martin E, et al. Nuclear proteomics uncovers diurnal regulatory landscapes in mouse liver . Cell Metab. 2017; ; 25 : :102.–117. 10.
Matsuo T, Yamaguchi S, Mitsui S, et al. Control mechanism of the circadian clock for timing of cell division in vivo . Science. 2003; ; 302 : :255.–259. 11.
Gentric G, Desdouets C. Polyploidization in liver tissue . Am J Pathol. 2014; ; 184 : :322.–331. |