| |
| Med Sci (Paris). 33(5): 519–527. doi: 10.1051/medsci/20173305016.Bdellovibrio et organismes apparentés Des prédateurs bactériens hors du commun ! Édouard Jurkevitch1* and Stéphan Jacquet2** 1Faculté d’Agriculture, de l’Alimentation et de l’Environnement, Université Hébraïque de Jérusalem, Rehovot, Israël 2INRA, UMR CARRTEL, 75, avenue de Corzent, 74200Thonon-les-Bains, France |
Les bactéries constituent quantitativement le groupe dominant des organismes cellulaires. En leur sein, les bactéries prédatrices d’autres bactéries ont été très largement sous-estimées comparativement à l’action prédatrice des virus ou d’autres prédateurs ou parasites eucaryotes (flagellés, cillés, champignons). Cela est en partie dû à ce que ces bactéries ne constituent pas des populations majeures en terme d’abondance quand les bactériophages, des virus infectant les bactéries, eux, peuvent atteindre des densités dix fois supérieures aux populations bactériennes [1]. Les bactéries prédatrices obligatoires de bactéries (c’est-à-dire dépendantes de la prédation pour se reproduire) sont regroupées dans un groupe polyphylétique1 appelé Bdellovibrio and like organisms (BALO) qui inclue divers groupes monophylétiques : le genre Micavibrio et les familles Bdellovibrionaceae, Bacteriovoraceae et Peridibacteraceae [2]. Ces BALO ont pour cible des bactéries Gram négatif. La pression de prédation, pouvant conduire à la sélection de proies génétiquement résistantes, et l’existence de défenses phénotypiques et de nature transitoire, qui constituent un mécanisme d’amortissement, assurent, in fine, à la fois la survie du prédateur et la capacité de la proie à se reconstituer [3, 4]. La dynamique des relations de type proie-prédateur entre bactéries s’avère donc complexe. S’ajoute à cette complexité, la nature mal définie du type d’interaction possible entre organismes (synergique, antagoniste, etc.). Cependant, les nouvelles technologies de séquençage, de bio-informatique et de microscopie, mais aussi le développement d’outils de manipulation génétique, permettent, aujourd’hui, une étude détaillée des mécanismes cellulaires qui font d’une bactérie, un prédateur obligatoire. Elles offrent également une approche plus large de l’étude de leur impact écologique. Ces connaissances ouvrent de nouvelles voies vers leur application en agriculture, en aquaculture, dans la préservation de notre environnement et en médecine. Cette revue est l’occasion de synthétiser une partie des connaissances actuelles sur les BALO. |
Le monde méconnu des BALO On sait aujourd’hui que les BALO sont très largement distribués au sein de la biosphère et sont présents aussi bien dans les océans, les mers, les eaux côtières, les estuaires, les rivières, les lacs, que dans les bassins d’aquaculture, les stations d’épuration, l’eau d’irrigation, ou encore dans les sols, les rizières, la rhizosphère et même dans les fèces animales [2]. Les BALO sont capables de cibler et consommer la grande majorité des bactéries Gram négatif présentes dans les eaux naturelles et donc potentiellement celles des autres écosystèmes dans lesquels ils sont présents. Les BALO étant capables de se déplacer vers des zones où leurs proies sont concentrées peuvent ainsi répondre à une augmentation rapide des cibles qui leur servent de nourriture. Le rôle biologique de certaines communautés de BALO pourrait donc être comparable à celui des virus [5] mais il est encore aujourd’hui mal caractérisé. Ainsi, les BALO du genre Halobacteriovorax semblent contribuer significativement à la mortalité bactérienne marine. Ils auraient un rôle important dans le mécanisme de recyclage des nutriments, une fonction largement attribuée aux virus jusque récemment. Une étude réalisée en microcosmes [5] montre qu’il n’est pas nécessaire que les BALO soient très abondants pour jouer un rôle déterminant dans la mortalité bactérienne. Elle révèle que la concentration de BALO peut augmenter d’un facteur 1 000 en quelques heures, mettant en évidence une capacité de croissance et de division très importante face à l’émergence et au développement d’une proie, ce qui peut avoir lieu dans de nombreuses situations : par exemple, au cours ou à la suite d’une efflorescence cyanobactérienne ou algale (c’est-à-dire lors de la prolifération d’une population bactérienne autotrophe ou d’algues) au sein de dispositifs comme les micropatchs, de biofilms ou de neige marine2. Ils pourraient donc affecter de nombreux paramètres de la structure (liés à la mortalité, la diversité) et du fonctionnement (comme le recyclage et redistribution des nutriments) des écosystèmes et former la base du développement de nouvelles approches antibactériennes ouvrant la voie à de nombreux champs d’application (Tableau I).
Tableau I.
| Discipline/Domaine |
Applications |
Prédateurs |
Référence |
| Médecine |
Traitement des infections (pathogènes intestinal et périodontal, infection oculaire, colonisation pulmonaire, inflammation et cytotoxicité) |
Bdellovibrio bacteriovorus, Bdellovibrio spp. (plusieurs isolats),Bacteriovorax stolpii, Bacteriovorax FCE, Peredibacter starrii, Micavibrio aeruginosavorus
|
[33]
[34]
[37]
[38]
[40]
[41] |
| Bio-contrôle de la production de β-lactamase, bactéries à Gram négatif multirésistantes |
B. bacteriovorus,M. aeruginosavorus
|
|
[42] |
| Contrôle des biofilms |
B. bacteriovorus,M. aeruginosavorus
|
|
[
43]
[
35] |
|
| Traitement des eaux |
Membrane bio-fouling
|
B. bacteriovorus
|
[44] |
| Purification de l’eau |
BALO détectés par qPCR ciblant les Bdellovibrionacées, Bacteriovorax, Bacteriolyticum; BALO isolés des eaux d’épuration, capables de prédater sur la plupart des Gram negatifs
Production de protéines à partir d’eaux usées, élimination du phosphore ; hautes concentrations de Bdellovibrio spp. détectées |
|
[
10]
[32]
[45] |
|
| Industrie alimentaire |
Élimination des bactéries des surfaces alimentaires ou sur des équipements |
B. bacteriovorus, Bdellovibrio spp. (plusieurs isolats) |
[46] |
| Contrôle de la croissance des bactéries pathogènes |
B. bacteriovorus, Peredibacter spp. |
|
[
46] |
|
| Agriculture |
Bio-contrôle des pathogènes de plantes |
B. bacteriovorus (plusieurs souches), Bdellovibrio spp. (plusieurs souches), B. stolpii, P. starrii, souche N-1 non classée |
[47]
[48] |
|
| Pisciculture |
Bio-contrôle des pathogènes de poissons |
Bdellovibrio C1, Bdellovibrio spp. (plusieurs isolats),
B. bacteriovorus, Bacteriovorax spp. (plusieurs isolats) |
[49]
[31] |
|
| Animaux d’élevage |
Probiotiques
Infections intestinale et oculaire |
B. bacteriovorus (plusieurs souches) |
[50] |
|
| Source de bio-produits |
Extraction des polyhydroxyalcanoates |
Co-culture avec B. bacteriovorus pour lyser les souches productrices de polyhydroxyalkanoates |
[51] |
Champs d’applications potentielles ou avérées des prédateurs obligatoires Bdellovibrio and like organisms (BALO).
|
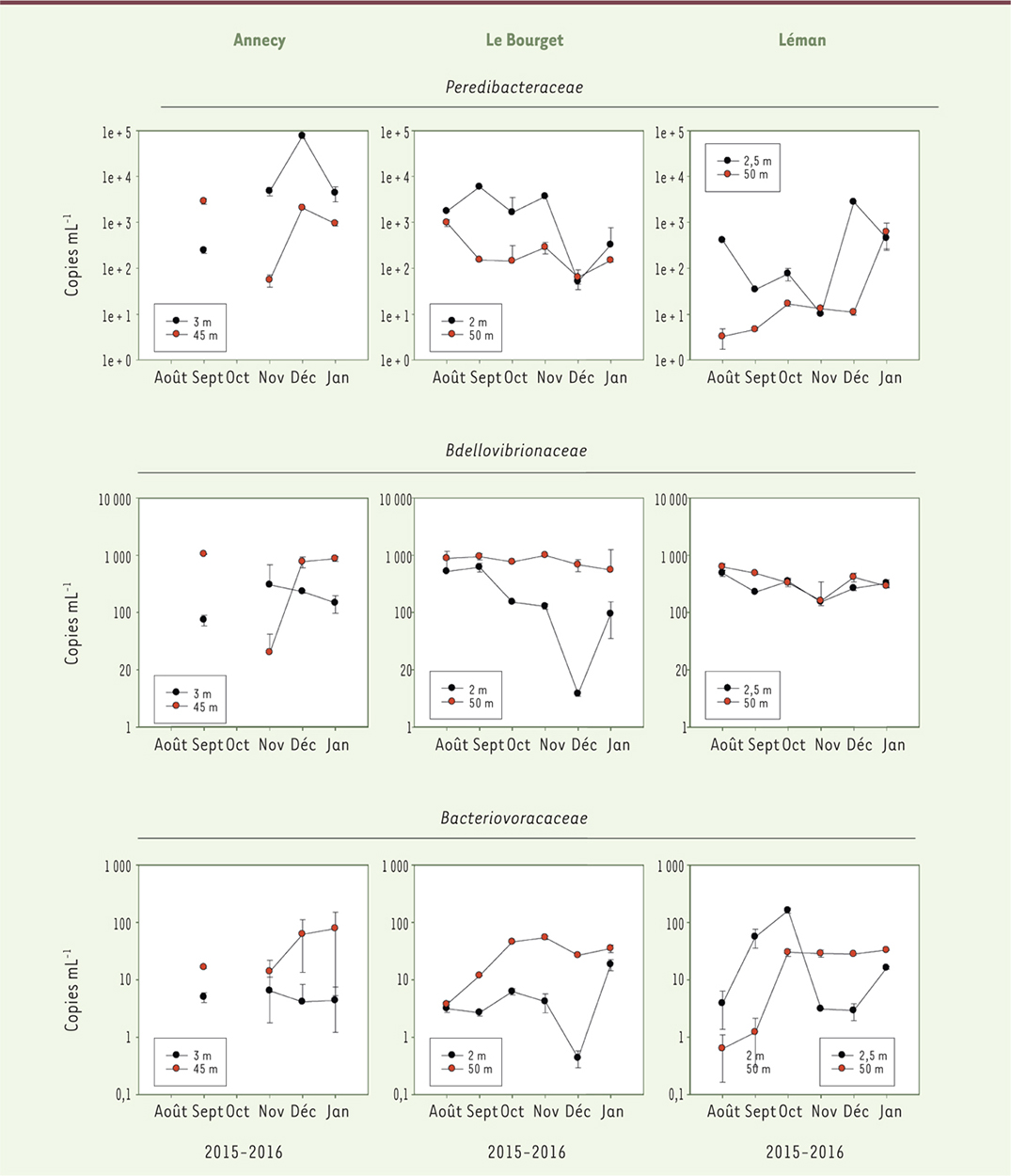
Le groupe des Bdellovibrionacées a longtemps été cantonné aux environnements d’eau douce ou du sol et celui des Bacteriovocacées, à des environnements salins ou hyper salins. Les Peredibacteracées ne coloniseraient aussi, a priori, que les eaux douces. Les Bdellovibrionacées semblent néanmoins capables de coloniser un grand nombre de milieux (des eaux douces aux eaux salées) et être assez ubiquistes [6, 7]. De la même manière, les Bacteriovocacées peuvent également être présents en eau douce. En fait la coexistence des différents groupes pourrait surtout être liée aux types de proies et à leurs caractéristiques (présentes/absentes, libres ou attachées, abondantes, qualité) dans les différents milieux, et au type de prédation lui-même (généraliste ou spécialisé). Les interactions proie-prédateur apparaissent très sensibles aux changements de l’environnement (température, concentration en nutriments) qui peuvent directement et indirectement déterminer la structuration et la composition de la communauté des BALO et de leurs proies (B. Paix et S. Jacquet, communication personnelle). L’étude la plus récente portant sur l’abondance, la distribution et la diversité des BALO en milieu naturel, est celle de Paix et Jacquet (Figure 1) dans laquelle il est observé que, dans les grands lacs péri-alpins, les Peredibacteracées sont peu diversifiées (majoritairement Peredibacter starii, une espèce d’eau douce) mais caractérisées par une abondance relativement élevée, en moyenne entre 100 à 10 000 fois supérieure à celle des Bdellovibrionacées et des Bacteriovoracacées, dont les souches décrites sont principalement isolées des milieux marins. L’abondance des BALO dans ces grands lacs semble donc fortement liée à leur très faible salinité. L’absence de sel dans le milieu favoriserait le développement de BALO adaptés comme Peredibacter starii et empêcherait l’abondance des bactéries telles que Bacteriovorax spp., plus adaptées aux milieux marins. L’abondance relative de ces prédateurs, par rapport à l’ensemble des bactéries hétérotrophes3, montre que les Peredibacteracées peuvent représenter jusqu’à 7,1 % de cette communauté, avec des concentrations dépassant 105 copies (du gène ARN 16S qui les caractérise) par mL. L’abondance des Peredibacteracées semble se distribuer de manière cohérente entre celle des prédateurs flagellés eucaryotes et celle des bactériophages (respectivement, 300 à 1 200 bactéries par mL dans le lac d’Annecy, et 4 x 107 à 108 particules virales par mL, dans le lac du Bourget) [8, 9]. Ces proportions indiquent que les Peredibacteracées pourraient jouer, de par leur abondance relative, un rôle non négligeable sur la dynamique biologique de leur proie et donc exercer un rôle important dans l’équilibre des grands lacs. L’étude de la dynamique des Peredibacteracées entre deux profondeurs distinctes montre qu’elles occupent une niche écologique majoritairement située en surface, contrairement aux Bdellovibrionacées et aux Bacteriovoracacées qui se situent principalement en profondeur. Les faibles concentrations de ces deux familles sont d’autant plus notables lorsqu’on les compare à celles mesurées dans les systèmes d’aquacultures (entre 103 et 106 cellules par mL) [10], laissant supposer qu’elles auraient un impact plus limité sur la communauté des bactéries hétérotrophes au sein des grands lacs. Rappelons cependant qu’une faible abondance de BALO n’est pas forcément corrélée à un faible impact fonctionnel sur la dynamique de leurs proies [5]. Les abondances bactériennes totales étant supérieures en moyenne à 106 cellules par mL [11], il ne fait aucun doute que les BALO ont trouvé dans les grands lacs une quantité et une diversité de proies suffisantes pour leur développement.
 | Figure 1.
Dynamique observée pendant quelques mois de l’année pour les trois grands lacs péri-alpins français et d’Europe occidentale (Annecy, Le Bourget, Léman) des différentes familles de BALO quantifiées par PCR ( polymerase chain reaction) quantitative à deux profondeurs distinguant les eaux de surface (entre 2 et 3 m) de celles plus en profondeur (entre 45 et 50 m) (d’après [ 8]) |
L’abondance et la diversité des populations de BALO dans les échantillons, initialement appréciées par des méthodes de culture in vitro, ont été largement sous-estimées. En effet, l’utilisation d’amorces spécifiques ciblant l’ADN codant l’ARNr 16S a révélé une quantité de deux ordres de magnitude supérieure à celle obtenue par comptage sur boîte de Pétri [11]. L’écologie moléculaire a ainsi permis d’évaluer la fraction de BALO dans un grand nombre d’environnements. Elle est en fait estimée, en moyenne, entre 0,2 et 0,5 % de la communauté totale des bactéries [2, 7]. Les approches moléculaires ont montré que l’identification d’isolats par culture ne permettait d’observer qu’un très faible pourcentage de la diversité des BALO [12]. Les BALO ne sont pas des prédateurs généralistes. Différentes souches ciblent en effet des proies différentes, ce qui a conduit à des erreurs dans l’estimation de la teneur en BALO dans l’environnement lors des approches d’isolement. La grande majorité des BALO et de leurs proies potentielles n’étant pas cultivables, il est donc difficile de les isoler. Il est d’ailleurs important de rappeler que les microprédateurs (et/ou parasites) incluent les phages, les BALO, et les protistes (représentés par les pico-, nano- et micro-flagellés et ciliés) sans oublier les champignons dont l’importance fonctionnelle est aujourd’hui reconnue, notamment dans les milieux aquatiques. Bien que les stratégies de ces prédateurs soient hiérarchisées (spécifiques, semi-généralistes ou versatiles et généralistes), il s’avère que celles-ci sont nettement plus complexes et comportent des interactions qui restent à peine connues. Ces interactions entre prédateurs pourraient ainsi jouer un rôle capital dans le fonctionnement des écosystèmes aquatiques et, par extrapolation, dans celui de tout autre écosystème intégrant de multiples microprédateurs et parasites [13]. L’étude de cette multi spécificité et donc de la diversité des interactions biologiques constituent aujourd’hui d’importants défis théoriques et expérimentaux. L’application de ces connaissances pourrait largement contribuer à améliorer la compréhension des flux de nutriments dans les environnements naturels mais aussi dans les systèmes créés par l’homme, par exemple pour la réduction de la biomasse dans le traitement des eaux usées. |
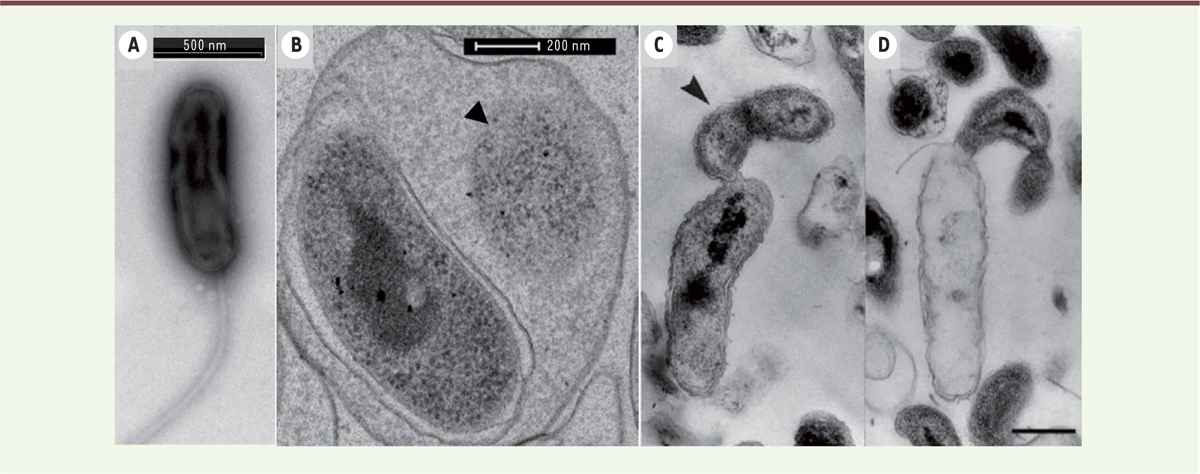
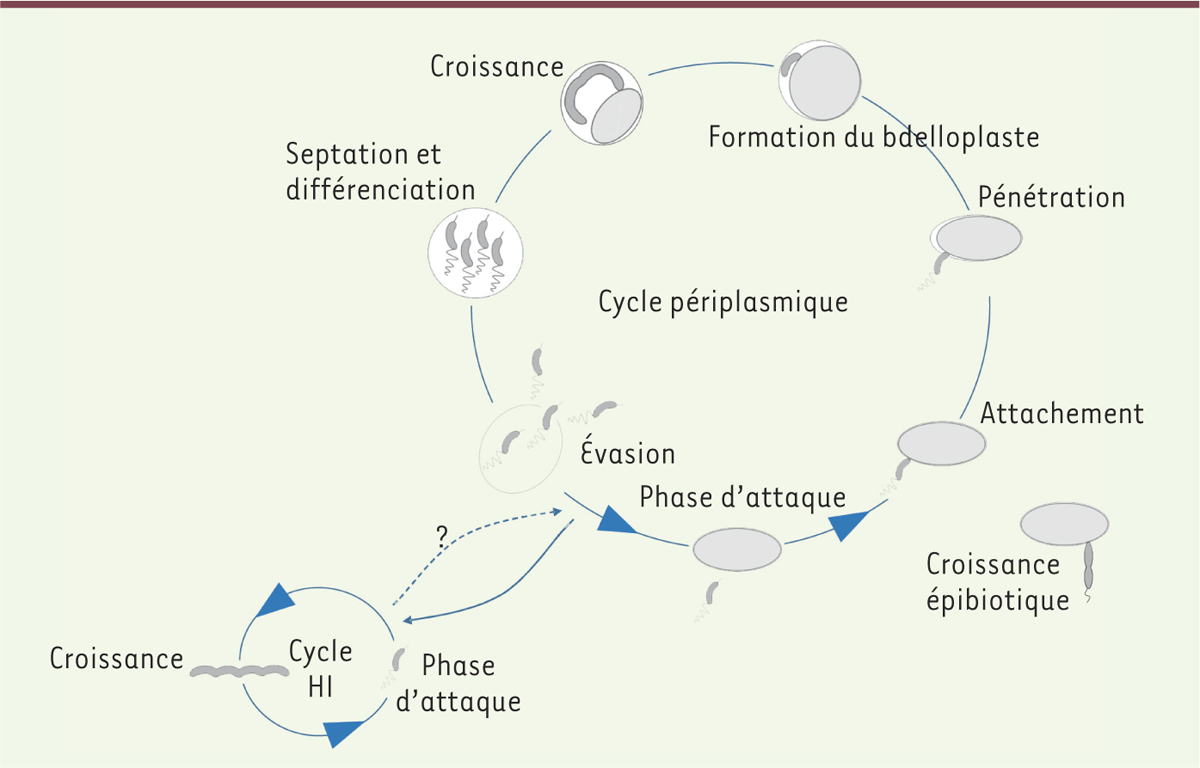
Le cycle cellulaire des Bdellovibrio et organismes apparentés Les BALO sont généralement considérés comme des organismes aérobies obligatoires mais l’action de prédation exercée par certaines espèces, comme Bdellovibrio bacteriovorus, peut être observée dans des conditions anoxiques, tant que l’apport en nitrates est possible [2]. Le cycle de vie des BALO est indissociable de leur cycle cellulaire. Il dépend de la présence de proies qui sont ciblées en phase d’attaque (PA). Durant cette phase, la bactérie prédatrice est motile et non-réplicative. La PA est suivie d’une phase de croissance et de reproduction (PC). Ces phases sont morphologiquement, spatialement, physiologiquement et transcriptionnellement très différentes. S’y ajoute une phase transitoire (PT) qui a été récemment décrite [14]. Une division supplémentaire de ces étapes dépend de la stratégie prédatrice adoptée. Elle est soit épibiotique soit périplasmique. Les prédateurs épibiotiques comme Bdellovibrio exovorus et Micavibrio aeruginosavorus s’ancrent à la paroi externe de leur cible. Ils digèrent le contenu de la cellule cible, croissent et se divisent de façon binaire (Figure 2 C et D). Les connaissances sur ce type de prédation sont plus limitées que celles sur la prédation périplasmique. Durant ce type de prédation, le périplasme4 de la cellule proie est envahi et sert d’habitat au développement et à la réplication du prédateur. Nous nous attacherons à décrire cette dernière plus en détail en utilisant B. bacteriovorus comme organisme modèle (Figure 2 A, B et Figure 3).
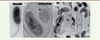 | Figure 2.
Observation en microscopie électronique à transmission de la phase d’attaque (A) et de la phase de croissance (B) du prédateur périplasmique Bdellovibrio bacteriovorus HD100 dans le périplasme d’une cellule d’Escherichia coli. On peut observer le cytoplasme réduit de la cellule proie (flèche en B) ; la croissance et la division binaire (flèche en C) du prédateur épibiotique B. exovorus fixé à sa proie (C). Proie vidée de son contenu cytoplasmique (D) (d’après [ 52]). |
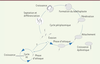 | Figure 3. Cycle cellulaire du prédateur périplasmique Bdellovibrio bacteriovorus, incluant les modes de vie du prédateur obligatoire et des mutants dérivés indépendants d’hôtes (host-independent, HI). |
La phase d’attaque Les cellules en phase d’attaque sont de petites dimensions (entre 0,7 et 1,5 x 0,5 mm). Elles adoptent souvent une forme de type vibrio, maintenue par MreB2, une protéine apparentée à l’actine. Le nucléoïde est compact et des granules denses aux électrons, ressemblant à des acidocalcisomes 5,, peuvent être présents dans le cytoplasme. Un flagelle unique, composé de six types de flagelline, ainsi que trois paires de protéines de motricité MotAB, confèrent une grande motilité qui permet au prédateur de rencontrer ses proies. Il n’est pas nécessaire à sa pénétration. La chimiotaxie influe positivement sur la détection des proies [ 2]. L’attachement de B. bacteriovorus à sa proie s’effectue par le pôle d’invasion, en contact avec la proie. Il est d’abord réversible mais, en quelques minutes, il devient irréversible. Le mécanisme de reconnaissance des proies par le prédateur reste obscur. Il pourrait reposer sur les oligosaccharides associés au lipopolysaccharide (LPS) exprimé par la proie pour B. bacteriovorus. B. bacteriovorus exprime également un lipide A (la base structurale du LPS) mais il est totalement dépourvu de groupes chargés négativement, en raison de la substitution des résidus phosphate (présents sur le LPS) par des résidus d’α-D-mannopyranose [15]. Le LPS de Bacteriovorax stolpii contient des sphingophosphonolipides qui sont uniques eux aussi. Ces altérations pourraient agir sur les interactions entre les membranes des prédateurs et celles des proies et ainsi avoir un rôle dans la prédation [16]. Cependant, la capacité d’un prédateur à cibler différentes proies suggèrent que ces interactions ne détermineraient pas la spécificité de la reconnaissance. Des pilus de type IVa6 présents au niveau du pôle d’invasion des cellules en PA, sont essentiels à la prédation bien que leur abolition ne compromette pas l’attachement du prédateur à sa proie [17]. Des analyses de transcriptomique effectuées sur B. bacteriovorus [18] ont révélé un profil d’expression génique hors du commun avec 15 % de gènes exprimés exclusivement durant la PA et 67 % de gènes exprimés seulement durant la PC. La majorité des gènes restants est exprimée au moins 5 fois plus dans l’une des deux phases [18]. Les fonctions exprimées en PA concernent la motilité, la chimiotaxie, l’adhérence et l’expression de protéines de la membrane externe. Ces résultats sont en accord avec ceux d’une étude protéomique menée par Dori-Bachash et al. sur B. bacteriovorus [19]. La phase transitoire Très récemment, l’existence entre PA et PC d’une phase discrète du cycle cellulaire [ 14] a été démontrée. Un système de croissance de souches sauvages de B. bacteriovorus (donc incapables de croissance axénique 7) sur des bactéries proies d’ Escherichia coli, vidées de leur contenu cytoplasmique, a été développé. Il a permis de montrer que les enveloppes, ainsi que le contenu cytoplasmique de la cellule, sont nécessaires mais que chacun séparément n’est pas suffisant pour permettre l’entrée en PC de cellules en PA. L’expression génique du prédateur en PA est successivement altérée de façon différentielle par chacune de ces deux composantes C’est pendant la phase transitoire (PT) que se construit le bdelloplaste, correspondant à la cellule proie pénétrée et modifiée par le prédateur, qui va lui fournir une niche protectrice et une base nutritionnelle pour sa croissance et sa multiplication. La PT apparaît comme un point de contrôle du cycle cellulaire où la décision de s’engager dans la croissance est liée à la détection du signal cytoplasmique. Celui-ci pourrait servir d’indicateur de la valeur nutritionnelle de la proie, afin d’empêcher l’entrée en PC en l’absence des ressources nécessaires à la complétion du cycle de reproduction [14]. Certains processus considérés jusqu’à présent comme faisant partie de la PC sont en fait caractéristiques de la PT. L’accès au périplasme dure entre 5 à 20 minutes et se réalise via une lyse locale de l’enveloppe de la cellule proie créant un pore par lequel le prédateur la pénètre [2]. Cette étape est sensible à la streptomycine, ce qui suggère la production d’enzymes pour la formation de ce pore. En parallèle, le peptidoglycane8 de la proie est déréticulé par des endo-carboxypeptidases spécifiques secrétées par le prédateur, empêchant ainsi la surinfection par un ou plusieurs autres prédateurs [20]. Le peptidoglycane est aussi déacétylé par deux N-acetylglucosamine déacetylases qui le rendent sensibles à la destruction enzymatique en fin de PC. B. bacteriovorus protège son propre peptidoglycane de l’action de ces enzymes par une protéine à motif ankyrine, permettant l’interaction entre protéines et souvent détecté chez les pathogènes intracellulaires des eucaryotes [21]. Phase de croissance et de réplication Dans le périplasme, la cellule prédatrice croît sous la forme de filaments (Figure 3) non segmentés. Elle contient alors de multiples nucléoïdes [ 2]. La division cellulaire et la répartition de ces nucléoïdes se produisent de façon synchronisée tout au long du filament, et ceci même si les proies sont infectées par deux prédateurs [ 2]. Il semblerait que la disparition du signal cytoplasmique de la proie enclenche ce processus [ 16]. Il serait intéressant de tester si le prédateur « se sent à l’étroit » dans le bdelloplaste. Les technologies de micro-lithographie et de micro-fluidique disponibles aujourd’hui pourraient le permettre [ 22]. Finalement, les cellules progénies en PA s’échappent du bdelloplaste par des pores créés dans l’enveloppe de la proie. Leur nombre est proportionnel à la taille de la proie [ 2]. Il varie de 5 à 7 en moyenne dans E. coli jusqu’à 30 dans Aquaspirillum serpens et plus dans des E. coli coenocitiques (multinucléés) de 30 à 100 µm de longueur. La durée du cycle de vie, de l’attachement du prédateur à la proie jusqu’à la libération de nouvelles cellules en PA, dure entre 2,5 et 4 h. Durant leur croissance, les BALO sont en mesure de synthétiser tous les nucléotides mais comme de nombreux symbiontes et pathogènes bactériens, ils ne peuvent pas synthétiser et dégrader certains acides aminés, ainsi que la riboflavine [23]. Pour des raisons encore inconnues, chez la plupart des BALO et chez des prédateurs bactériens non-obligatoires, la production des isoprénoïdes9 utilise la voie de biosynthèse du mévalonate et non la voie du méthylérythritol phosphate commune à la grande majorité des bactéries [24]. Cycle cellulaire hôte-indépendant Peu après la découverte de B. bacteriovorus, des variants saprophytes (hôte-indépendant - HI) capables de croître en l’absence de proie ont été isolés par étalement de suspensions concentrées de cellules de souches sauvages sur un milieu complet [ 25]. À ce jour, il a été possible de générer des mutants HI à partir de nombreuses souches de B. bacteriovorus et de Halobacteriovorax (auparavant Bacteriovorax). Quelques souches HI ont aussi été isolées de l’environnement. Par contre, il n’a pas été possible d’obtenir de souches HI à partir de BALO épibiotiques 10, [ 24]. Les souches HI conservent le cycle de vie dimorphique des BALO et présentent un potentiel prédateur amoindri par rapport à celui de la souche parentale. Elles le perdent complètement après transfert séquentiel en milieu complet. Deux types de souches HI ont été caractérisés : le type I, saprophyte, apparaît spontanément avec une fréquence de 10-6 à 10-7. Il forme de petites colonies qui ne se développent qu’en milieu riche contenant de l’extrait de proie. Il ne nécessite pas la présence de cellules ou d’enveloppes de proies. Il est donc indépendant du signal généré par l’interaction physique entre le prédateur et la paroi de la proie. Le type II dérive du type I avec une fréquence de 10-2 à 10-3. Axénique, il forme de robustes colonies qui croissent en milieu riche sans besoin d’apport d’extrait de proie. Le type I résulte de mutations dans le locus hit (host interaction, ou bd108) [26], un gène codant une protéine qui serait impliquée dans l’extension/rétraction des pilus de type IVa [27]. Les mutants HI de type II résultent de modifications dans rhlB (ATP-dependent RNA helicase) ou dans pcnB (poly[A] polymerase I), des gènes codant des composantes du dégradosome ARN11,. La perte de fonction de l’un des deux gènes permet la transition du type I au type II [26]. Les mutants de type II sont donc indépendants du facteur soluble qui contrôle l’entrée en PC (voir le chapitre phase transitoire). Signalisation cellulaire Les gènes hit et rhlB-pcnB sont donc respectivement impliqués dans la reconnaissance des signaux associés à l’enveloppe et au cytoplasme de la proie. Le premier signal est vraisemblablement lié au contact établi entre le pilus, situé au niveau du pôle d’invasion du prédateur, et la proie [ 28]. Les seconds dépendent de la fonction de dégradation des ARN [ 16]. Le pilus pourrait interagir avec le complexe protéique du pôle d’invasion constitué entre autres, de la protéine RomR ( required for motility response regulator) [ 28] qui interagit avec CdgA ( cyclic-di-GMP diguanylate cyclase), une protéine possédant un domaine de signalisation GGDEF dégénéré [ 29] dont une mutation ralentit la formation du bdelloplaste. CdgA fait partie du système de signalisation du messager secondaire di-GMP cyclique qui impacte fortement le phénotype de B. bacteriovorus et contrôle l’aiguillage entre prédation et croissance HI [ 29]. Le pôle d’invasion relierait donc la détection de la proie au système de signalisation du di-GMP cyclique. Ce système semble former un réseau étendu chez B. bacteriovorus qui pourrait affecter de nombreuses fonctions [ 30]. De plus, merRNA, un ARN très fortement exprimé dans la PA et rapidement dégradé pendant la TP et au début de la PC, coderait un riboswitch
12, du diGMP cyclique [ 18]. Le di-GMP cyclique pourrait donc constituer le lien entre le stade d’invasion de la proie et le début de la croissance cellulaire du prédateur. |
Les milieux naturels ont été encore très peu explorés pour évaluer l’abondance, la diversité et le rôle écologique des BALO. D’autres systèmes, comme les bassins d’aquaculture, ont permis de montrer que certaines espèces y étaient abondantes et susceptibles d’y jouer un contrôle biologique efficace contre certains pathogènes bactériens (du genre Aeromonas ou Vibrio). Les BALO peuvent donc constituer un type de thérapie microbienne contribuant in fine à l’augmentation des performances de croissance de certains animaux aquatiques, comme les poissons ou les crevettes [31]. Les BALO seraient également abondants dans des systèmes de retraitement des eaux usées [10, 32]. On pourrait ainsi les utiliser afin de réduire la biomasse et les concentrations de pathogènes dans des systèmes moins onéreux que les usines actuelles. Les applications médicales des BALO constituent un autre pôle d’intérêt croissant. L’efficacité des BALO dans la destruction des biofilms a été testée contre les pathogènes parodontaux [33, 34]. Ils peuvent également désagréger des biofilms mixtes constitués de proies Gram négatif et de bactéries à Gram positif non-proies, comme Staphylococcus aureus, réduisant ainsi leur virulence [35]. Les BALO ont été proposés comme antibiotiques vivants [36]. Un programme international, financé par l’agence américaine DARPA (Defense advanced research projects agency) a été lancé en mai 2015 afin d’explorer cette possibilité. Les premiers travaux en émanant mettent en évidence l’innocuité de ces prédateurs [37, 38] et leur efficacité in vivo dans des modèles animaux, incluant le rat [39, 40]. Enfin, l’écologie nous apprend que les prédateurs jouent un rôle primordial dans l’équilibre, la diversité et la résilience des écosystèmes. En fonction de ces connaissances, il a été récemment proposé que l’appauvrissement bactérien observé dans les dysbioses de l’écosystème microbien intestinal pourrait résulter de la perte des prédateurs de bactéries intestinales, plus particulièrement des protistes et des BALO [41]. Le
Tableau I
résume l’ensemble des applications potentielles ou avérées à ce jour. |
Conclusions et perspectives Les BALO jouent sans nul doute un rôle non négligeable dans le fonctionnement des écosystèmes naturels et artificiels mais ce rôle reste encore clairement à appréhender. L’utilisation du séquençage à haut débit alliée à des approches de PCR (polymerase chain reaction) ciblée et de visualisation directe par hybridation in situ pourrait (1) permettre un suivi fin et précis des BALO dans toute leur diversité, et l’identification de leurs proies dans l’environnement, et (2) révéler l’influence des différents paramètres du milieu sur les relations proie-prédateur et leur dynamique. Ce type d’étude serait pertinent dans des domaines aussi variés que l’écologie microbienne des milieux aquatiques et du sol, l’agriculture et la pisciculture, la qualité environnementale, mais aussi dans les domaines médicaux et pharmaceutiques. ‡ |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Fuhrman JA. Marine viruses and their biogeochemical and ecological effects . Nature. 1999; ; 399 : :541.–548. 2.
Rotem O, Pasternak Z, Jurkevitch E. Bdellovibrio and Like Organisms . In Rosenberg E, DeLong EF, Lory S, Stackebrandt E, Thompson F. eds The Prokaryotes: Deltaproteobacteria and Epsilonproteobacteria . Berlin, Heidelberg: : Springer Berlin Heidelberg; , 2014 : :3.–17. 3.
Gallet R, Tully T. M.K.E. Evans. Ecological conditions affect evolutionary trajectory in a predator-prey system . Evolution. 2009; ; 63 : :641.–651. 4.
Shemesh Y, Jurkevitch E. Plastic phenotypic resistance to predation by Bdellovibrio and like organisms in bacterial prey . Environ Microbiol. 2004; ; 6 : :8.–12. 5.
Williams HN, Lymperopoulou DS, Athar R, et al. Halobacteriovorax, an underestimated predator on bacteria: potential impact relative to viruses on bacterial mortality . ISME J. 2016; ; 10 : :491.–499. 6.
Dolinšek J, Lagkouvardos I, Wanek W, et al. Interactions of nitrifying bacteria and heterotrophs: Identification of a Micavibrio-Like putative predator of Nitrospira spp . Appl Environm Microbiol. 2013; ; 79 : :2027.–2037. 7.
Chen H, Young S, Berhane T-K, Williams HN. Predatory Bacteriovorax communities ordered by various prey species . PLoS ONE. 2012; ; 7 : :e34174.. 8.
Jacquet S, Domaizon I, Personnic S, et al. Estimates of protozoan- and viral-mediated mortality of bacterioplankton in Lake Bourget (France) . Freshwater Biol. 2005; ; 50 : :627.–645. 9.
Personnic S, Domaizon I, Dorigo U, et al. Seasonal and spatial variability of virio-, bacterio-, and picophytoplanktonic abundances in three peri-alpine lakes . Hydrobiologia. 2009; ; 627 : :99.–116. 10.
Kandel PP, Pasternak Z, van Rijn J, et al. Abundance, diversity and seasonal dynamics of predatory bacteria in aquaculture zero discharge systems . FEMS Microbiol Ecol. 2014; ; 89 : :149.–161. 11.
Zheng G, Wang C, Williams HN, Pineiro SA. Development and evaluation of a quantitative real-time PCR assay for the detection of saltwater Bacteriovorax . Environ Microbiol. 2008; ; 10 : :2515.–2526. 12.
Davidov Y, Friedjung A, Jurkevitch E. Structure analysis of a soil community of predatory bacteria using culture-dependent and culture-independent methods reveals a hitherto undetected diversity of Bdellovibrio-and-like organisms . Environ Microbiol. 2006; ; 8 : :1667.–1673. 13.
Miki T, Jacquet S. Complex interactions in the microbial world: underexplored key links between viruses, bacteria and protozoan grazers in aquatic environments . Aquatic Microbial Ecology. 2008; ; 51 : :195.–208. 14.
Rotem O, Pasternak Z, Shimoni E, et al. Cell-cycle progress in obligate predatory bacteria is dependent upon sequential sensing of prey recognition and prey quality cues . Proc Natl Acad Sci U S A. 2015; ; 112 : :E6028.–E6E37. 15.
Schwudke D, Linscheid M, Strauch E, et al. The obligate predatory Bdellovibrio bacteriovorus possesses a neutral Lipid A containing α-D-mannoses that replace phosphate residues . J Biol Chem. 2003; ; 278 : :27502.–27512. 16.
Kaneshiro ES, Hunt SM, Watanabe Y. Bacteriovorax stolpii proliferation and predation without sphingophosphonolipids . Biochem Biophys Res Commun. 2008; ; 367 : :21.–25. 17.
Evans KJ, Lambert C, Sockett RE. Predation by Bdellovibrio bacteriovorus HD100 requires type IV Pili . J Bacteriol. 2007; ; 189 : :4850.–4859. 18.
Karunker I, Rotem O, Dori-Bachash M, et al. A global transcriptional switch between the attack and growth forms of Bdellovibrio bacteriovorus . PLoS ONE. 2013; ; 8 : :e61850.. 19.
Dori-Bachash M, Dassa B, Pietrokovski S, Jurkevitch E. Proteome-based comparative analyses of growth stages reveal new cell cycle-dependent functions in the predatory bacterium Bdellovibrio bacteriovorus . Appl Environ Microbiol. 2008; ; 74 : :7152.–7162. 20.
Lerner TR, Lovering AL, Bui NK, et al. Specialized peptidoglycan hydrolases sculpt the intra-bacterial niche of predatory Bdellovibrio and increase population fitness . PLoS Pathog. 2012; ; 8 : :e1002524.. 21.
Lambert C, Cadby IT, Till R, et al. Ankyrin-mediated self-protection during cell invasion by the bacterial predator Bdellovibrio bacteriovorus . Nat Commun. 2015; ; 6 : :8884.. 22.
Hol FJ, Rotem O, Jurkevitch E, et al. Bacterial predator–prey dynamics in microscale patchy landscapes . Proc Biol Sci. 2016;; 283. pii: 20152154. 23.
Pasternak Z, Njagi M, Shani Y, et al. In and out: an analysis of epibiotic vs periplasmic bacterial predators . ISME J. 2014; ; 8 : :625.–635. 24.
Pasternak Z, Pietrokovski S, Rotem O, et al. By their genes ye shall know them: genomic signatures of predatory bacteria . ISME J. 2013; ; 7 : :756.–769. 25.
Shilo M, Bruff B. Lysis of Gram negative bacteria by host-independent ectoparasitic Bdellovibrio bacteriovorus isolates . J Gen Microbiol. 1965; ; 40 : :317.–328. 26.
Roschanski N, Klages S, Reinhardt R, et al. Identification of genes essential for prey-independent growth of Bdellovibrio bacteriovorus HD100 . J Bacteriol. 2011; ; 193 : :1745.–1756. 27.
Capeness MJ, Lambert C, Lovering AL, et al. Activity of Bdellovibrio hit locus proteins, Bd0108 and Bd0109, links type IVa pilus extrusion/retraction status to prey-independent growth signalling . PLoS ONE. 2013; ; 8 : :e79759.. 28.
Milner DS, Till R, Cadby I, et al. Ras GTPase-Like protein MglA, a controller of bacterial social-motility in myxobacteria, has evolved to control bacterial predation by Bdellovibrio . PLoS Genet. 2014; ; 10 : :e1004253.. 29.
Hobley L, Fung RKY, Lambert C, et al. Discrete cyclic di-GMP-dependent control of bacterial predation versus axenic growth in Bdellovibrio bacteriovorus . PLoS Pathog. 2012; ; 8 : :e1002493.. 30.
Rotem O, Nesper J, Borovok I, et al. An extended cyclic di-GMP network in the predatory bacterium Bdellovibrio bacteriovorus . J Bacteriol. 2016; ; 198 : :127.–137. 31.
Cao H, He S, Lu L, et al. Identification of a Proteus penneri isolate as the causal agent of red body disease of the cultured white shrimp Penaeus vannamei and its control with Bdellovibrio bacteriovorus . Antonie van Leeuwenhoek. 2014; ; 105 : :423.–430. 32.
Feng S, Tan CH, Cohen Y, Rice SA. Isolation of Bdellovibrio bacteriovorus from a tropical wastewater treatment plant and predation of mixed species biofilms assembled by the native community members . Environ Microbiol. 2016; ; 18 : :3923.–3931. 33.
Dashiff A, Kadouri DE. Predation of oral pathogens by Bdellovibrio bacteriovorus 109J . Mol Oral Microbiol. 2011; ; 26 : :19.–34. 34.
Van Essche M, Quirynen M, Sliepen I, et al. Killing of anaerobic pathogens by predatory bacteria . Mol Oral Microbiol. 2011; ; 26 : :52.–61. 35.
Monnappa AK, Dwidar M, Seo JK, et al. Bdellovibrio bacteriovorus Inhibits Staphylococcus aureus biofilm formation and invasion into human epithelial cells . Sci Rep. 2014; ; 4 : :3811.. 36.
Sockett ER, Lambert C. Bdellovibrio as therapeutic agents: a predatory renaissance? Nat Rev Microbiol. 2004; ; 2 : :669.–675. 37.
Gupta S, Tang C, Tran M, Kadouri DE. Effect of predatory bacteria on human cell lines . PLoS ONE. 2016; ; 11 : :e0161242.. 38.
Romanowski EG, Stella NA, Brothers KM, et al. Predatory bacteria are nontoxic to the rabbit ocular surface . Sci Rep. 2016; ; 6 : :30987.. 39.
Shatzkes K, Singleton E, Tang C, et al. Predatory bacteria attenuate Klebsiella pneumoniae burden in rat lungs . MBio. 2016;; 7. pii: :e01847.–16. 40.
Willis AR, Moore C, Mazon-Moya M, et al. Injections of predatory bacteria work alongside host immune cells to treat Shigella infection in zebrafish larvae . Curr Biol. 2016; ; 26 : :3343.–3351. 41.
Mosca A, Leclerc M, Hugot JP. Gut microbiota diversity and human diseases: should we reintroduce key predators in our ecosystem? Front Microbiol. 2016; ; 7 : :455.. 42.
Kadouri DE, To K, Shanks RM, Doi Y. Predatory bacteria: a potential ally against multidrug-resistant Gram-negative pathogens . PLoS One. 2013; ; 8 : :e63397.. 43.
Kadouri D, O’Toole GA. Susceptibility of biofilms to Bdellovibrio bacteriovorus attack . Appl Environ Microbiol. 2005; ; 71 : :4044.–4051. 44.
Kim E-H, Dwidar M, Mitchell RJ, Kwon Y-N. Assessing the effects of bacterial predation on membrane biofouling . Water Research. 2013; ; 47 : :6024.–6032. 45.
Zuo N, He J, Ma X, et al. Phosphorus removal performance and population structure of phosphorus-accumulating organisms in HA-A/A-MCO sludge reduction process . Bioengineered. 2016 : :1.–7. 46.
Framatico P.M. WRC. Ability of Bdellovibrio bacteriovorus 109J to lyse Gram-negative food-borne pathogenic and spoilage bacteria . J Food Protect. 1995; ; 58 : :160.–164. 47.
Uematsu T. Ecology of Bdellovibrio parasitic to rice bacterial leaf blight pathogen. , Xanthomonas oryzae. Rev Plant Protect Res. 1980; ; 13 : :12.–26. 48.
Jurkevitch E, Minz D, Ramati B. G B. Prey range characterization, ribotyping, and diversity of soil and rhizosphere Bdellovibrio spp. isolated on phytopathogenic bacteria . Appl Environ Microbiol. 2000; ; 66 : :2365.–2371. 49.
Li H, Chen C, Sun Q, et al. Bdellovibrio and Like Organisms enhanced growth and survival of Penaeus monodon and altered bacterial community structures in its rearing water . Appl Environ Microbiol. 2014; ; 80 : :6346.–6354. 50.
Atterbury RJ, Hobley L, Till R, et al. Effects of orally administered Bdellovibrio bacteriovorus on the well-being and Salmonella colonization of young chicks . Appl Environ Microbiol. 2011; ; 77 : :5794.–5803. 51.
Martínez V, Herencias C, Jurkevitch E, Prieto MA. Engineering a predatory bacterium as a proficient killer agent for intracellular bio-products recovery: The case of the polyhydroxyalkanoates . Sci Rep. 2016; ; 6 : :24381.. 52.
Koval SF, Hynes SH, Flannagan RS, et al. Bdellovibrio exovorus sp. nov., a novel predator of Caulobacter crescentus . Int J Syst Evol Microbiol. 2013; ; 63 : :146.–151. |