| |
| Med Sci (Paris). 33(5): 512–518. doi: 10.1051/medsci/20173305015.TopoVIL Un ciseau moléculaire essentiel à la reproduction Thomas Robert,1 Bernard de Massy,1* and Mathilde Grelon2** 1Institut de Génétique Humaine, UMR9002 CNRS-Université de Montpellier, 141, rue de la Cardonille, 34396Montpellier Cedex 05, France 2Institut Jean-Pierre Bourgin, INRA, AgroParisTech, CNRS, Université Paris-Saclay, RD10, 78026Versailles Cedex, France |
Vignette (Photo © Inserm – Manuel Mark et Norbert Ghyselinck). La méiose est une division cellulaire particulière, essentielle à la reproduction sexuée, car elle permet la formation, à partir d’une cellule diploïde, des cellules haploïdes à l’origine des gamètes. Elle comprend deux divisions chromosomiques successives qui sont précédées d’une seule phase de réplication de l’ADN. La première division, appelée méiose I ou division réductionnelle, permet la séparation des chromosomes homologues à destination des deux cellules filles. La seconde division (la méiose II ou division équationnelle) est similaire à une mitose car elle permet la séparation des chromatides sœurs. Lors de la méiose I, les chromosomes homologues sont associés afin d’être correctement ségrégés. Chez un grand nombre d’organismes, comme les mammifères ou les plantes, cet appariement dépend du processus de recombinaison homologue (RH) qui établit un lien physique entre les chromosomes homologues (Figure 1).
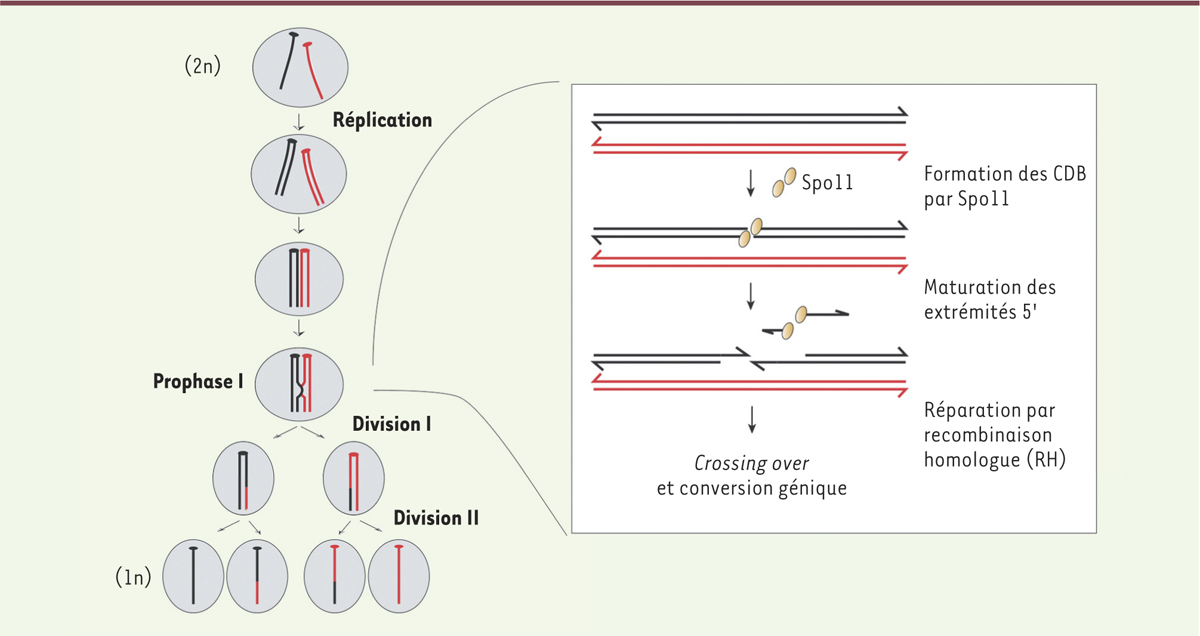
 | Figure 1.
Recombinaison homologue par formation et réparation de cassures double brin (CDB) lors de la méiose. La méiose assure la formation de cellules haploïdes (1n) à l’origine des gamètes, à partir de cellules diploïdes (2n), par une étape de réplication suivie de deux divisions successives. La recombinaison homologue est induite lors de la prophase de la première division de méiose par la formation de CDB catalysées par Spo11. Seule une copie de chaque chromosome parental avec les deux brins de l’ADN est représentée dans l’encadré. Les extrémités 3’ sont indiquées par des flèches. Spo11 forme un complexe covalent avec les extrémités 5’ de la CDB. Les extrémités subissent ensuite une maturation, ce qui conduit à la libération de Spo11 lié à des oligonucléotides courts et à la formation d’extrémités 3’ simple brin. La cassure est réparée par recombinaison homologue et conduit à des événements de conversion génique associés ou non à des crossing over.
|
La RH est un mécanisme de réparation des cassures de l’ADN qui permet de réparer une séquence cassée en recopiant l’information d’une séquence d’ADN homologue intacte. Elle conduit à des évènements de conversion génique associés ou non à des crossing-over (CO, en français, enjambements). En début de prophase de première division méiotique, des cassures double brin de l’ADN (CDB) sont produites, puis réparées en recopiant l’information sur le chromosome homologue. La méiose I a donc ceci de très particulier que, pour s’effectuer correctement, elle nécessite la formation de cassures de l’ADN qui représentent une menace si elles ne sont pas réparées correctement. La formation de ces cassures est programmée en début de prophase I et plusieurs protéines sont nécessaires à leur apparition. En particulier, il a été proposé, en 1997, que la protéine Spo11 catalyse leur formation (Figure 1) [1] (→). Spo11 est une protéine conservée, homologue de la sous-unité catalytique (appelée A) des topoisomérases TopoVI : la TopoVIA. Toutefois, en dépit de l’extrême importance de l’activité de Spo11, son mécanisme d’action reste mystérieux. En particulier, les TopoVI fonctionnent grâce à l’assemblage de deux sous-unités A et B, et la question de l’existence d’une sous-unité B, nécessaire à l’activité de Spo11, était restée ouverte depuis 1997. La récente identification de cette sous-unité B chez les plantes et les vertébrés permet, aujourd’hui, de progresser dans la vision que nous avons du mode d’action et d’évolution de Spo11 [2, 3].
(→) Voir la Synthèse de F. Baudrat et B. de Massy, m/s n° 2, février 2004, p. 213
L’objectif de cette synthèse est de présenter l’état des lieux des connaissances sur la protéine Spo11 et de faire un bilan des nouvelles données concernant l’identification de la sous-unité B nécessaire à la formation des CDB méiotiques. |
Spo11 ou l’identification d’une activité enzymatique méiotique grâce aux archées Un mutant de SPO11 a été initialement isolé chez la levure Saccharomyces cerevisiae en 1969 par un crible génétique qui avait pour but d’identifier des souches présentant un défaut de sporulation [4]. C’est ensuite, en 1985, qu’a été mis en évidence un défaut de recombinaison homologue à l’origine du défaut de sporulation chez ce mutant et, en 1990, qu’un défaut de formation des CDB méiotiques a été démontré [5, 6]. L’implication directe de Spo11 dans la formation de ces CDB repose sur deux études indépendantes réalisées, ensuite, en 1997. Dans la première, il a été montré que la protéine Spo11 était l’homologue de la sous-unité catalytique (A) de la topoisomérase TopoVI de l’archée1
Sulfolobus shibatae (TopoVIA). Les auteurs de cette étude ont, de plus, montré que la tyrosine catalytique de TopoVIA était conservée dans la séquence de Spo11 et ont démontré, par mutagenèse dirigée, que cette tyrosine était essentielle à la formation des CDB méiotiques chez la levure [7]. Les TopoVI sont des topoisomérases de type II. Elles sont composées de deux sous-unités A et de deux sous-unités B qui s’assemblent en un hétérotétramère (A2B2) (Figure 2A). Elles agissent sur la topologie de l’ADN en catalysant des cycles de coupure/ligature de la molécule. Les coupures sont des CDB qui conduisent à la constitution d’intermédiaires formés de liaisons covalentes réalisées entre les extrémités 5’ de l’ADN et la TopoVIA, via sa tyrosine catalytique. Cette liaison est transitoire et la CDB est ultérieurement ligaturée [8] (Figure 3A). Au vu de ces résultats, Bergerat et al. ont proposé que la protéine Spo11 catalyse la formation des CDB méiotiques selon un mécanisme similaire à celui utilisé par le dimère de TopoVIA. Parallèlement à cette première étude, une seconde, également réalisée chez la levure, a permis de détecter dans les cellules en méiose des intermédiaires covalents entre la protéine Spo11 et l’ADN [9]. Ainsi, les auteurs ont proposé que Spo11 catalysait les CDB méiotiques selon un processus proche de celui utilisé par les topoisomérases. Ces travaux ont donc permis de proposer non seulement que Spo11 porte l’activité enzymatique responsable de la formation des CDB méiotiques, mais aussi que son activité soit en partie similaire à celle des TopoVI.
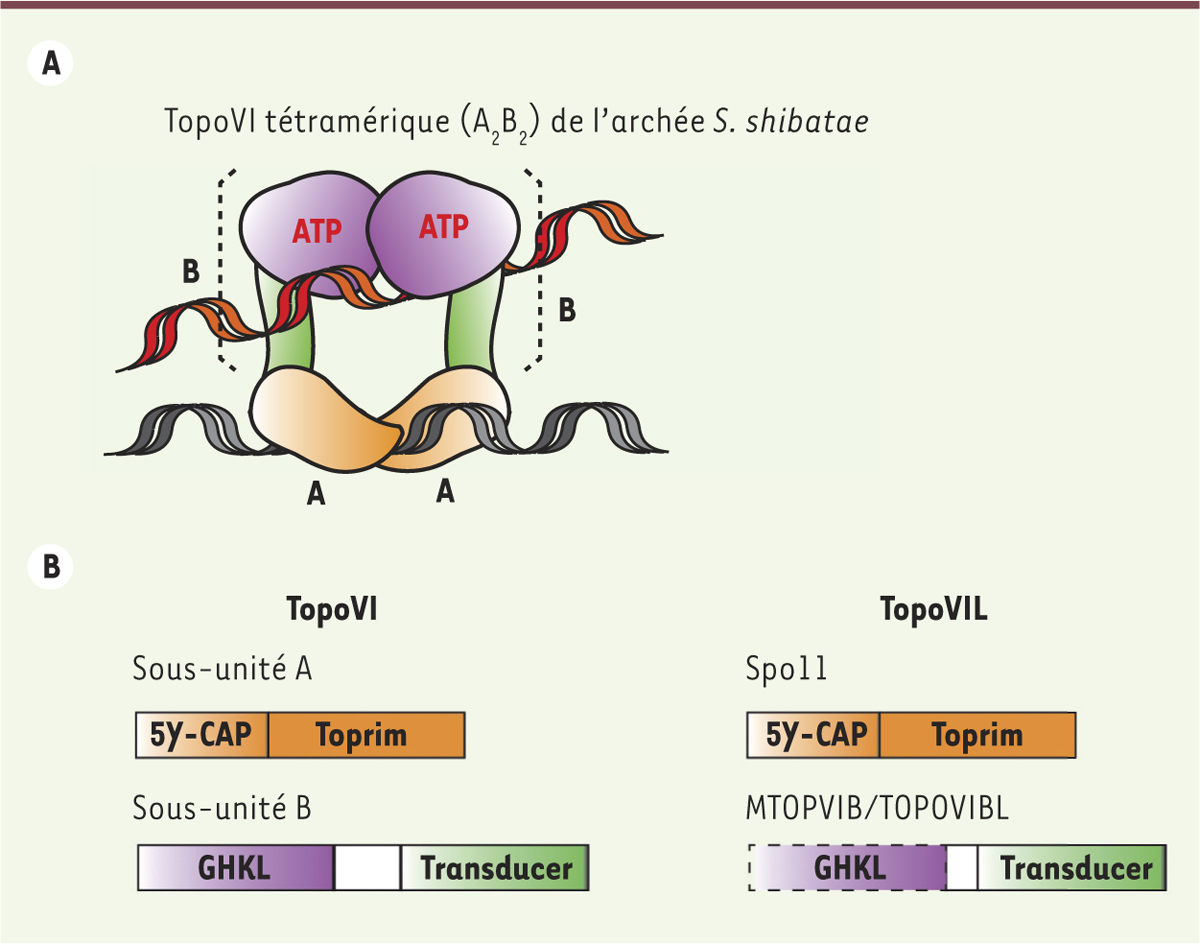
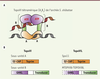 | Figure 2.
Spo11 et MTOPVIB/TOPOVIBL, orthologues des sous-unités A et B des TopoVI. A. La TopoVI est un hétérotétramère (A2B2) dans lequel les sous-unités A (en orange) se dimérisent et interagissent avec l’ADN. Les sous-unités B possèdent un domaine « transducer » (en vert) qui interagit avec la sous-unité A et un domaine commun à plusieurs ATPase/kinase, GHKL (en mauve), pouvant se dimériser après fixation d’ATP. Le schéma représente le complexe dit « fermé », engagé dans une réaction de coupure/ligature. B. Les deux domaines des TopoVIA (le 5Y-CAP qui inclut la tyrosine catalytique et le domaine Toprim) sont conservés chez Spo11. Le domaine GHKL des TopoVIB qui porte l’activité de fixation et d’hydrolyse de l’ATP avec les motifs définissant le « Bergerat Fold » est faiblement conservé dans la famille MTOPVIB/TOPOVIBL au niveau de la séquence primaire. Ce domaine est indiqué en pointillé pour les protéines MTOPVIB/TOPOVIBL car il n’est pas identifié chez certaines espèces (S. cerevisiae et D. melanogaster). Le domaine « transducer » qui inclut la région d’interaction avec la sous-unité A est conservé. |
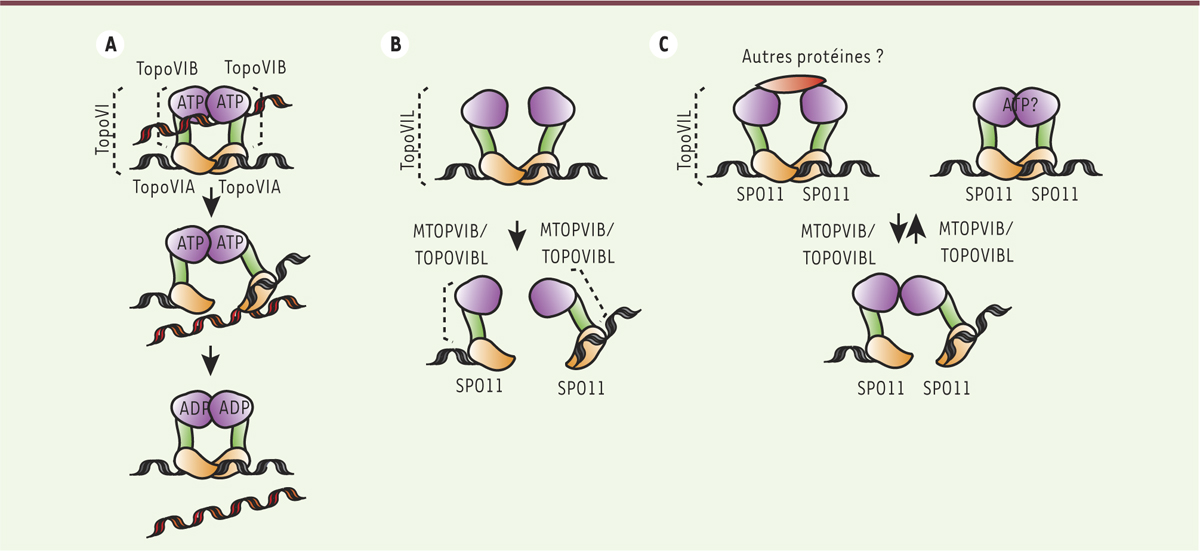
 | Figure 3.
Comparaison des modes d’action des complexes TopoVI et TopoVIL. A. Cycle de coupure/ligature de la TopoVI. Les TopoVI modifient la topologie de l’ADN en permettant le passage d’un duplex d’ADN (en rouge) à travers un deuxième duplex (en noir) transitoirement coupé. Les coupures générées sont des CDB qui conduisent à la formation d’intermédiaires covalents entre les extrémités de l’ADN (en noir) et la TopoVIA. Cette liaison est transitoire et la CDB est ultérieurement ligaturée. Ces cycles de coupure/ligature sont dépendants de la liaison et de l’hydrolyse de molécules d’ATP par la sous-unité B de l’enzyme. B. Dans ce modèle, TopoVIL possède une activité de formation des CDB qui ne nécessite pas la fixation de l’ATP et/ou d’interactions entre les sous-unités B. C. Ce modèle postule que soit l’ATP, soit d’autres protéines soient nécessaires à la formation des CDB, entre autres pour stabiliser le complexe. On peut dans ce contexte émettre l’hypothèse d’une réaction réversible. CDB : cassures double brin ; ATP : adénosine triphosphate ; ADP : adénosine diphosphate. |
À la suite de ces travaux, des homologues de la protéine Spo11 ont été identifiés chez de nombreuses espèces. Leur analyse révèle une forte conservation évolutive avec les protéines d’archées, en particulier au niveau des deux domaines principaux des TopoVIA, le domaine de fixation à l’ADN (5Y-CAP, pour catabolite gene activator protein) et le domaine d’interaction avec les cations bivalents (domaine Toprim [topoisomerase-primase], commun aux topoisomérases et aux primases) (Figure 2B) [10]. Lorsque cela a pu être testé, l’absence de Spo11 se caractérise par un défaut de CDB méiotiques accompagnée d’une diminution drastique de la recombinaison homologue ainsi que par des erreurs de ségrégation des chromosomes en méiose I et finalement par une très forte diminution de la fertilité [11]. En dépit de son rôle clé dans la reproduction, la caractérisation biochimique de l’activité de Spo11 n’a pas été réalisée, principalement du fait de son insolubilité, et les données concernant son mode d’action restent parcellaires [12]. À l’instar de ce qui est observé pour les TopoVI, les données obtenues suggèrent que Spo11 est active sous la forme d’un dimère qui catalyse la génèse de CDB via la formation de deux brèches simple brin dans la molécule d’ADN. Cependant, les nombreuses données biochimiques sur les topoisomérases ont démontré que les domaines Toprim et 5Y-CAP ne suffisent pas à promouvoir, seuls, l’activité de coupure de l’ADN et que la TopoVIA agit en interaction avec la sous-unité B, TopoVIB. TopoVIB possède, au niveau de son extrémité N-terminale, un domaine de liaison à l’ATP appelé « Bergerat fold
2,» [7], présent dans la famille de protéines ATP-ases dite GHKL (pour gyrase, HSP [heat shock protein] 90, histidine kinase et MutL3,), et en C-terminal un domaine appelé « transducer
4» impliqué dans l’interaction avec TopoVIA (Figure 2). À moins d’imaginer un mode d’action très différent entre TopoVIA et Spo11, l’hypothèse la plus probable était donc que l’activité de Spo11 implique une deuxième sous-unité (pour revue voir [13]). Un maillon manquant semblait donc envisageable et sa recherche a été entreprise dans plusieurs laboratoires. |
L’identification du partenaire manquant, MTOPVIB/TOPOVIBL : de l’importance de la phylogénie et des approches multi-organismes Au cours de ces 20 dernières années, des approches génétiques menées sur diverses espèces modèles - principalement S. cerevisiae, Schizosaccharomyces pombe, Arabidopsis thaliana, Mus musculus, Caenorhabditis elegans et Drosophila melanogaster - ont permis d’identifier une vingtaine de protéines indispensables à la formation des CDB méiotiques [11] (Tableau I). L’analyse de leurs séquences protéiques n’a apporté que peu d’information sur leurs rôles lors de l’initiation de la recombinaison. En effet, dans la grande majorité des cas, aucune similarité avec des protéines de fonction connue n’a pu être détectée et, à l’exception notable de Spo11 (voir ci-dessus), ces protéines « de cassures » apparaissent très rarement conservées d’une espèce à l’autre. Le développement des analyses bioinformatiques des génomes a permis de dépasser un certain nombre de ces blocages. D’une part, des analyses phylogénétiques poussées établissent des relations évolutives entre des protéines de cassure identifiées au sein d’espèces distantes ; d’autre part, l’amélioration des outils d’analyse et de prédiction des structures tridimensionnelles des protéines ont permis de révéler des pistes fonctionnelles jusqu’alors insoupçonnées.
Tableau I.
| S. cerevisiae |
S. pombe |
A. thaliana |
M. musculus |
C. elegans |
D. melanogaster |
| Spo11 |
Rec12 |
SPO11-1
SPO11-2 |
SPO11 |
SPO-11 |
Mei-W68 |
|
| Rec102 |
Rec6 |
MTOPVIB |
TOPOVIBL |
|
Mei-P22 |
|
| Rec104 |
|
|
|
|
|
|
| Ski8 |
Rec14 |
|
|
|
|
|
| Rec114 |
Rec7 |
|
|
|
|
|
| Mei4 |
Rec24 |
PRD2 |
MEI4 |
|
|
|
| Mer2 |
Rec15 |
|
|
|
|
|
| Mre11 |
|
|
|
MRE-11 |
|
|
| Rad50 |
|
|
|
|
|
|
| Xrs2 |
|
|
|
|
|
|
|
|
|
PRD1 |
MEI1 |
|
|
|
|
|
|
PRD3 |
|
|
|
|
|
|
|
DFO |
|
|
|
|
|
|
|
|
|
|
Trem |
|
|
|
Mde2 |
|
|
|
|
|
|
|
|
|
|
DSB-1 |
|
|
|
|
|
|
IHO1 |
|
|
|
| CDC7 |
Hsk1 |
|
|
|
|
|
| DBF4 |
|
|
|
|
|
Protéines indispensables à la formation des cassures double brin (CDB) méiotiques. Seules les protéines pour lesquelles un rôle majeur dans la formation des CDB méiotiques a été démontré sont mentionnées. Sur une même ligne sont indiquées les protéines présentant une certaine conservation de séquence, de structure et/ou de fonction d’une espèce à l’autre (d’après [ 2, 3, 11, 23]). |
Ainsi, une approche dite « phylo-génomique » menée en 2010, fondée sur des recherches de similarités de séquences et de structures secondaires, a établi un lien évolutif clair entre les protéines de cassures de S. cerevisiae, Mei4 (meiotic recombination protein 4), de S. pombe, Rec24 (meiotic recombination protein 24), et de A. thaliana, PRD2 (putative recombination initiation defect 2), et a permis l’identification de leur homologue fonctionnel chez les mammifères (Tableau I) [14]. Ce résultat et son extension à la famille des protéines regroupant Rec114 chez S. cerevisiae, Rec7 chez S. pombe et PHS1 (poor homologous synapsis 1) chez Z. maydis [14], a dévoilé, pour la première fois, une claire conservation des protéines de cassures de l’ADN en méiose au cours de l’évolution. Ce sont des approches similaires de génétique et de « phylo-génomique», menées en collaboration par nos équipes en 2016, qui nous ont conduit à l’identification du chaînon manquant, avec la caractérisation d’une famille de protéines présentant de fortes similarités avec les TopoVIB d’archées et impliquées dans l’initiation de la recombinaison méiotique. En prenant comme point de départ l’identification, grâce à un crible génétique, d’une séquence codant, chez la plante A. thaliana, une protéine indispensable à la formation des CDB méiotiques, une nouvelle famille de protéines a pu être identifiée. Cette famille compte des membres chez les plantes, dans le règne animal, mais également des homologues distants chez les levures S. cerevisiae et S. pombe ainsi que chez la drosophile. Ces homologues distants correspondent à des protéines déjà caractérisées pour leur rôle dans la formation des cassures double brin méiotiques, S. cerevisiae Rec102, S. pombe Rec6 et D. melanogaster Mei-P22 (Tableau I) [2, 3]. En ce qui concerne la séquence primaire, la conservation de la famille de protéines que nous avons identifiée avec TopoVIB est faible et difficilement détectable. En revanche, les prédictions de structure tridimensionnelle révèlent des similarités significatives avec TopoVIB, au sein des deux domaines fonctionnels : le « Bergerat fold » et le « transducer » [2, 3]. Nous avons nommé ces protéines MTOPVIB (pour meiotic TOPOVIB-Like) chez les plantes, et TOPOVIBL (pour TOPOVIB-Like) chez les mammifères. Compte tenu de la similarité entre Spo11 et la sous-unité A des TopoVI (voir ci-dessus), il est tentant de proposer que l’initiation de la recombinaison méiotique chez les eucaryotes soit sous le contrôle d’un complexe de type TopoVI, dénommé TopoVI-Like (ou TopoVIL), qui impliquerait deux sous-unités A (correspondant à SPO11) et deux sous-unités B (correspondant à MTOPVIB ou TOPOVIBL). Nous avons déjà largement validé cette hypothèse en démontrant que les protéines TOPOVIBL et SPO11 de souris forment, in vitro, un complexe de taille compatible avec celle d’un hétérotétramère SPO112/TOPOVIBL2 [3]. De plus, une interaction directe entre MTOPVIB ou TOPOVIBL et SPO11 a été détectée. Comme prédit par le modèle, cette interaction implique l’extrémité C-terminale des sous-unités B méiotiques [2, 3]. Enfin, la protéine MTOPVIB d’A. thaliana est nécessaire et suffisante pour promouvoir une interaction entre les deux paralogues5, SPO11-1 et SPO11-2, tous deux nécessaires à la formation des CDB méiotiques [2, 15–17]. Chez A. thaliana, le complexe prédit impliquerait donc trois partenaires, SPO11-1, SPO11-2 et deux MTOPVIB. Les mutants de plantes et de souris mtopVIB/topoVIBL montrent tous les phénotypes attendus résultant du défaut de formation des CDB méiotiques : entre autres, une absence de recombinaison homologue, un défaut d’appariement des homologues, et la stérilité. Ces phénotypes sont similaires à ceux observés chez un mutant spo11, tant chez la plante A. thaliana que chez la souris, et en accord avec l’hypothèse de la formation d’un complexe de type TopoVI responsable de l’activité catalytique de formation des CDB méiotiques. |
Le complexe « TopoVI-Like » porte l’activité de CDB de l’ADN lors de la méiose : mode d’action proposé Le complexe TopoVIL qui apparaît issu phylogénétiquement des complexes TopoVI d’archées, résulte sans doute d’une duplication de gènes survenue très tôt lors de l’évolution des eucaryotes [10]. Les protéines codées par les gènes Spo11 et MTOPVIB/TopoVIBL ont évolué et on observe une divergence nettement plus prononcée au niveau de la sous-unité B que de la sous-unité A (correspondant à Spo11). Au cours de ce processus évolutif, et en relation avec une fonction lors de la méiose, le complexe TopoVIL a dû acquérir des propriétés biochimiques en partie modifiées. L’activité méiotique est, en première analyse, une activité de coupure de l’ADN qui, contrairement à une activité de type TopoII (topoisomerase II), n’impliquerait pas d’étape de religature. Compte tenu du rôle clef que joue la sous-unité B pour le cycle de coupure/ligature des TopoII, on peut supposer que celle-ci ait subi des modifications importantes compatibles avec la fonction méiotique. En effet, la divergence au sein de la famille MTOPVIB/TOPOVIBL est telle que la capacité de fixation et d’hydrolyse de l’ATP des sous-unités méiotiques B est incertaine, plusieurs motifs du « Bergerat fold » n’étant pas conservés (chez A. thaliana et M. musculus) voire absents (chez S. cerevisiae et D. melanogaster) [13]. En revanche, il est frappant de constater la forte conservation entre Spo11 et TopoVIA, à mettre en relation avec le fait que l’activité catalytique de coupure de l’ADN est portée par la sous-unité A des TopoVI. En prenant en compte les données acquises concernant, d’une part, la biochimie des TopoII et, d’autre part, le contrôle génétique des CDB méiotiques, plusieurs modes d’action non exclusifs du complexe TopoVIL peuvent donc être envisagés. Les données biochimiques sur la TopoVI montrent que la présence d’ATP, mais pas son hydrolyse, est nécessaire pour l’activité de coupure de l’ADN [18] (Figure 3A). La fixation de l’ATP induit la dimérisation du domaine de liaison de l’ATP GHKL et un changement de conformation de la protéine nécessaires pour la coupure de l’ADN. Cependant la relation entre fixation de l’ATP et coupure de l’ADN n’est pas stricte car d’autres topoisomérases de type II ne requièrent pas l’ATP pour la coupure de l’ADN [19]. En revanche, le cycle de coupure/ligature requiert l’ATP pour la majorité des TopoII. Selon la capacité de MTOPVIB/TOPOVIBL à fixer et à hydrolyser, ou non, l’ATP (des propriétés qui seront essentielles à analyser), on peut donc, à ce stade, envisager une activé méiotique de cassures avec ou sans religature. Les données génétiques incitent en outre à envisager un mode d’action qui pourrait être plus complexe. Chez toutes les espèces étudiées, plusieurs autres protéines sont indispensables pour la formation des CDB méiotiques (Tableau I). Leurs rôles et activités sont inconnus, mais une ou plusieurs de ces protéines pourraient être directement impliquées pour l’activité catalytique de formation des CDB par le complexe TopoVIL. Un certain nombre de données d’analyse d’interactions protéiques réalisées chez les levures sont en accord avec une telle hypothèse [20, 21]. En conséquence, un scénario simple est que les CDB sont formées par le complexe TopoVIL au sein duquel la sous-unité B n’aurait qu’un rôle de stabilisation et/ou d’organisation du complexe, sans fixation d’ATP (Figure 3B). Il n’est pas nécessaire d’envisager dans ce cas de figure une dimérisation des sous-unités B et les autres protéines requises pourraient être impliquées directement ou indirectement dans l’accessibilité de l’ADN et/ou la conformation du complexe par exemple. Alternativement, la formation des CDB méiotiques pourrait nécessiter des propriétés de dimérisation des sous-unités B et/ou de changement conformationnel. Celles-ci pourraient impliquer la fixation d’ATP et/ou l’interaction avec un ou plusieurs autres partenaires identifiés. Ce mode d’action serait plus proche, d’un point de vue moléculaire, de celui d’une TopoVI et l’on pourrait même envisager dans ces conditions que le complexe TopoVIL ait une activité de religature (Figure 3C). L’induction de CDB potentiellement réversibles a sans doute une signification importante quant au maintien de la stabilité du génome. La régulation de la formation de CDB pourrait alors s’opérer à deux niveaux : l’activation de l’activité catalytique de Spo11 et l’orientation de la réaction coupure/ligature. |
Les approches génétiques et phylogénétiques que nous avons menées ont permis d’identifier, chez différents eucaryotes, une nouvelle protéine nécessaire à la formation des CDB et à la recombinaison lors de la méiose. Cette découverte apporte un maillon manquant qui était recherché depuis des années. Elle ouvre la perspective de pouvoir analyser, par des approches biochimiques, les mécanismes moléculaires impliqués dans la formation des CDB. Ces données établissent également clairement la relation évolutive entre les topoisomérases et l’activité de formation des CDB lors de la méiose. Aux niveaux moléculaire et structural, il sera extrêmement intéressant d’analyser comment une activité de type topoisomérase a évolué vers une activité de coupure irréversible de l’ADN. Il est par ailleurs remarquable, et intriguant, de constater cette spécificité de l’évolution de la TopoVI et qu’aucune autre activité de type TopoII n’ait suivi une évolution similaire. Ces questions croisent et recoupent celles liées à la compréhension de l’émergence de la reproduction sexuée lors de l’évolution des eucaryotes et du passage de l’état haploïde à l’état diploïde [22]. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
L’IJPB bénéficie du soutien du Labex Saclay Plant Sciences-SPS (ANR-10-LABX-0040-SPS). BdM et TR sont financés par le Centre National pour la Recherche Scientifique (CNRS) et l’European Research Council Executive Agency under the European Community’s Seventh Framework Programme (FP7/2007-2013 Grant Agreement no. [322788]).
|
Footnotes |
1.
Baudat F, de Massy B. SPO11: une activité de coupure de l’ADN indispensable à la méiose . Med Sci (Paris). 2004; ; 20 : :213.–218. 2.
Vrielynck N, Chambon A, Vezon D, et al. A DNA topoisomerase VI-like complex initiates meiotic recombination . Science. 2016; ; 351 : :939.–943. 3.
Robert T, Nore A, Brun C, et al. The TopoVIB-Like protein family is required for meiotic DNA double-strand break formation . Science. 2016; ; 351 : :943.–949. 4.
Esposito MS, Esposito RE. The genetic control of sporulation in Saccharomyces. I. The isolation of temperature-sensitive sporulation-deficient mutants . Genetics. 1969; ; 61 : :79.–89. 5.
Cao L, Alani E, Kleckner N. A pathway for generation and processing of double-strand breaks during meiotic recombination in S. cerevisiae . Cell. 1990;; 61 : :1089.–10101. 6.
Klapholz S, Waddell CS, Esposito RE. The role of the SPO11 gene in meiotic recombination in yeast . Genetics. 1985; ; 110 : :187.–216. 7.
Bergerat A, de Massy B, Gadelle D, et al. An atypical topoisomerase II from Archaea with implications for meiotic recombination . Nature. 1997; ; 386 : :414.–417. 8.
Vos SM, Tretter EM, Schmidt BH, et al. All tangled up: how cells direct, manage and exploit topoisomerase function . Nat Rev Mol Cell Biol. 2011; ; 12 : :827.–841. 9.
Keeney S, Giroux CN, Kleckner N. Meiosis-specific DNA double-strand breaks are catalyzed by Spo11, a member of a widely conserved protein family . Cell. 1997; ; 88 : :375.–384. 10.
Malik S-B, Ramesh M a, Hulstrand AM, et al. Protist homologs of the meiotic Spo11 gene and topoisomerase VI reveal an evolutionary history of gene duplication and lineage-specific loss . Mol Biol Evol. 2007;; 24 : :2827.–2841. 11.
de Massy B. Initiation of meiotic recombination: how and where? Conservation and specificities among eukaryotes . Annu Rev Genet. 2013; ; 47 : :563.–599. 12.
Diaz RL, Alcid AD, Berger JM, et al. Identification of residues in yeast Spo11p critical for meiotic DNA double-strand break formation . Mol Cell Biol. 2002; ; 22 : :1106.–1115. 13.
Robert T, Vrielynck N, Mézard C, et al. A new light on the meiotic DSB catalytic complex . Semin Cell Dev Biol. 2016; ; 54 : :165.–176. 14.
Kumar R, Bourbon HM, de Massy B. Functional conservation of Mei4 for meiotic DNA double-strand break formation from yeasts to mice . Genes Dev. 2010; ; 24 : :1266.–1280. 15.
Grelon M, Vezon D, Gendrot G, et al. AtSPO11-1 is necessary for efficient meiotic recombination in plants . EMBO J. 2001; ; 20 : :589.–600. 16.
Hartung F, Wurz-Wildersinn R, Fuchs J, et al. The catalytically active tyrosine residues of both SPO11-1 and SPO11-2 are required for meiotic double-strand break induction in Arabidopsis . Plant Cell. 2007; ; 19 : :3090.–3099. 17.
Stacey NJ, Kuromori T, Azumi Y, et al. Arabidopsis SPO11-2 functions with SPO11-1 in meiotic recombination . Plant J. 2006; ; 48 : :206.–216. 18.
Buhler C, Lebbink JHG, Bocs C, et al. DNA topoisomerase VI generates ATP-dependent double-strand breaks with two-nucleotide overhangs . J Biol Chem. 2001; ; 276 : :37215.–37222. 19.
Chen SH, Chan N-L, Hsieh T. New mechanistic and functional insights into DNA topoisomerases . Annu Rev Biochem. 2013; ; 82 : :139.–170. 20.
Miyoshi T, Ito M, Kugou K, et al. A central coupler for recombination initiation linking chromosome architecture to S phase checkpoint . Mol Cell. 2012; ; 47 : :722.–733. 21.
Maleki S, Neale MJ, Arora C, et al. Interactions between Mei4, Rec114, and other proteins required for meiotic DNA double-strand break formation in Saccharomyces cerevisiae . Chromosoma. 2007; ; 116 : :471.–486. 22.
Goodenough U, Heitman J. Origins of eukaryotic sexual reproduction . Cold Spring Harb Perspect Biol. 2014; :6.. 23.
Stanzione M, Baumann M, Papanikos F, et al. Meiotic DNA break formation requires the unsynapsed chromosome axis-binding protein IHO1 (CCDC36) in mice . Nat Cell Biol. 2016; ; 18 : :1208.–1220. |