| |
| Med Sci (Paris). 33(5): 469–473. doi: 10.1051/medsci/20173305004.Du neuf dans l’oncogenèse moléculaire des lymphomes : rôle du gène SETD2 dans les lymphomes T de l’intestin David Vallois,1 Annalisa Roberti,1 Bettina Bisig,1 Philippe Gaulard,2 and Laurence de Leval1* 1Institut de pathologie, Centre hospitalier universitaire Vaudois, Lausanne, Suisse 2Inserm U955, Université Paris-est, département de pathologie, hôpital Henri-Mondor, Créteil, France |
Lymphomes T périphériques Les lymphomes T périphériques (LTP) dérivés de cellules T (ou NK, natural killer) matures regroupent une trentaine de maladies rares, qui représentent ensemble moins de 15 % des hémopathies lymphoïdes malignes [1]. Ces néoplasies se manifestent sous forme de proliférations tissulaires, touchant les ganglions lymphatiques, la peau ou d’autres organes (région nasale, intestins, rate, etc.), ou de leucémies. La majorité des LTP non cutanés sont des maladies agressives, souvent résistantes aux polychimiothérapies classiques et ont un mauvais pronostic vital, avec une survie à 5 ans de moins de 30 %. La pathogenèse des LTP est multifactorielle et complexe. Au plan génétique, jusqu’il y a peu, un très petit nombre de translocations ou de mutations récurrentes étaient connues. Récemment, l’analyse de ces tumeurs par différentes techniques de séquençage à haut débit a mis en évidence de nombreuses et diverses anomalies génétiques récurrentes, confirmant l’hétérogénéité moléculaire de ces maladies et mettant aussi en évidence l’implication de caractéristiques communes. Parmi les mutations les plus fréquentes, citons celles touchant différents modulateurs épigénétiques impliqués dans la méthylation de l’ADN, TET2 (Tet methylcytosine dioxygenase 2), DNMT3A (DNA [cytosine-5]-methyltransferase 3A) et IDH2 (isocitrate dehydrogenase [NADP(+)] 2, mitochondrial), particulièrement retrouvées dans les lymphomes angio-immunoblastiques et autres lymphomes ganglionnaires dérivés de lymphocytes T auxilliaires (helper) folliculaires (TFH) [2]. Ces altérations, conduisant globalement à une hyperméthylation de l’ADN, sont fréquemment associées à des mutations touchant d’autres gènes impliqués dans diverses voies de signalisation ou processus métaboliques. Notamment, des mutations à l’origine de variants du gène codant la GTPase1, RhoA (ras homolog family member A ; mutation G17V) ou ciblant d’autres gènes de la voie de signalisation du récepteur antigénique des cellules T (TCR) sont détectées dans la majorité des lymphomes TFH, généralement aussi mutés pour TET2 [2, 3] (→).
(→) Voir la Nouvelle de L. Couronné, m/s n° 5, mai 2014, page 488
|
Les lymphomes T dérivés des cellules T intraépithéliales de l’intestin sont des néoplasies de phénotype cytotoxique, très agressives, pour lesquelles les options thérapeutiques sont peu nombreuses et globalement inefficaces. La forme la plus fréquente, le lymphome T associé à une entéropathie (LTAE, précédemment désigné LTAE de type I) survient souvent comme complication de la maladie coeliaque, liée à l’intolérance au gluten. Le second type, le lymphome T intestinal monomorphe épithéliotrope (LTIME, précédemment nommé LTAE de type II) [4], survient de manière sporadique sans association avec la maladie coeliaque et son incidence est moindre que celle du LTAE en Europe. Le LTIME est plus fréquemment dérivé de cellules T exprimant l’isoforme gamma-delta du TCR, tandis que le LTAE est plus souvent dérivé de cellules T alpha-bêta. Pour le LTAE, le modèle de lymphomagenèse implique des étapes successives relativement bien caractérisées sur le plan clinique et morphologique (maladie coeliaque, maladie coeliaque réfractaire, lymphome T intraépithélial, LTAE), et l’acquisition progressive d’aberrations génomiques (gains et pertes de régions chromosomiques) [5]. Peu de données étaient disponibles sur le LTIME. à partir d’un séquençage à haut débit de l’exome (WES, whole exome sequencing) d’échantillons appariés d’ADN tumoral/normal de 15 patients atteints de LTIME [4], nous avons caractérisé le paysage mutationnel (Figure 1A), les variations du nombre de copies (CNV) et les pertes d’hétérozygotie (LOH, loss of heterozygosity) des tumeurs. Notre étude a mis en évidence des altérations récurrentes du gène SETD2 (SET domain containing 2) présentes dans quasiment tous les cas (14/15 échantillons, soit 93 %). Il s’agissait de mutations inactivatrices et/ou de délétions du locus génique correspondant en 3p21, souvent bialléliques (deux mutations dans 7 cas, et une mutation avec perte du locus 3p21 dans 3 cas). SETD2 code une lysine méthyltransférase (H3K36 triméthyltransférase2,), qui est la seule enzyme chez l’homme à catalyser la triméthylation de la lysine en position 36 de l’histone H3 (transformation de H3K36me2 en H3K36me3) [4, 6] (Figure 1 A,B). Afin de confirmer les conséquences fonctionnelles des altérations génétiques, nous avons évalué par immunohistochimie semi-quantitative l’expression de la lysine méthyltransférase SETD2, ainsi que les niveaux de l’histone H3 diméthylée (H3K36me2) et triméthylée (H3K36me3) sur des coupes histologiques des tumeurs. En accord avec l’inactivation attendue de SETD2 (conséquence de mutations délétères), la majorité des tumeurs montraient un niveau réduit de H3K36me3 (Figure 1C), souvent concomitant d’une réduction d’expression de SETD2. À noter que la seule perte d’un allèle par délétion n’entraîne pas d’effet fonctionnel par elle-même. En revanche, la présence d’un allèle muté, même en présence d’un allèle fonctionnel, peut entraîner une diminution de H3K36me3, suggérant un effet dominant de ces mutations inactivatrices. En outre, tous les lymphomes étudiés portaient une ou plusieurs mutations activatrices ciblant un ou plusieurs éléments de la voie de signalisation JAK/STAT (janus kinase/ signal transducers and activators of transcription). Le séquençage ciblé d’échantillons tumoraux supplémentaires a confirmé la grande fréquence des mutations de SETD2 et de la voie JAK/STAT sur une plus grande série de LTIME. Les altérations génétiques caractéristiques des LTIME n’ont pas été identifiées dans les 8 cas de LTAE étudiés.
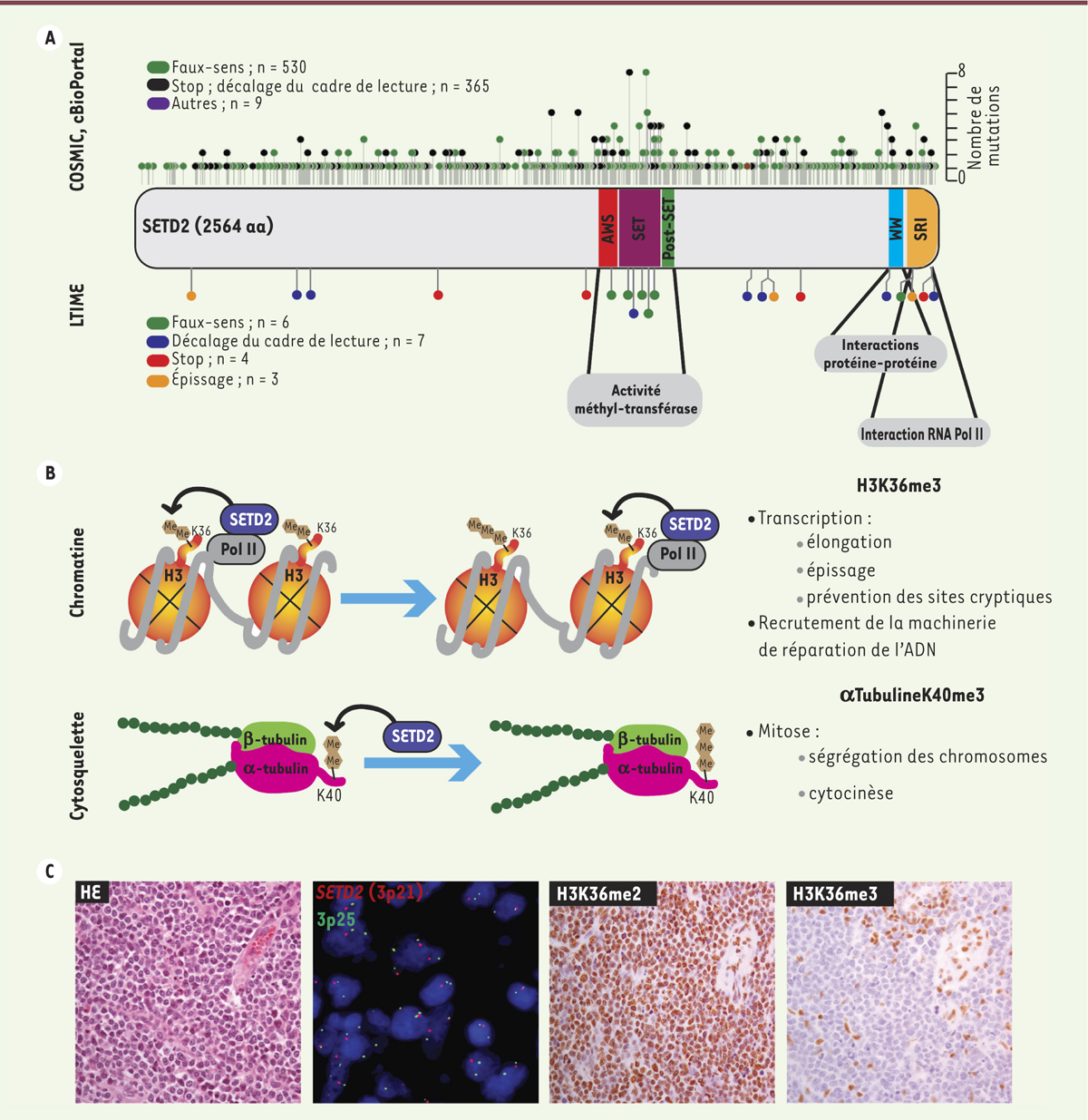
 | Figure 1.
Schématisation de la protéine SETD2 (SET domain containing 2) et de ses fonctions. (A) Les mutations répertoriées dans COSMIC ( catalogue of somatic mutations in cancer) et cBioPortal, deux bases de données listant les mutations génétiques dans les cancers, ou identifiées dans notre étude sur 15 LTIME [ 4], sont indiquées et positionnées sur le schéma de la protéine SETD2. (B) Fonctions de SETD2 dans le remodelage des nucléosomes et l’organisation des microtubules du fuseau mitotique. (C) Illustration de l’histopathologie d’un cas de lymphome T intestinal monomorphe épithéliotrope (LTIME). La coloration à l’HE (hématoxyline-éosine) indique que la tumeur est constituée de nappes monotones de lymphocytes de taille moyenne, en l’absence de composante inflammatoire significative. SETD2 (3p21)/3p25 : hybridation in situ fluorescente (FISH) illustrant une délétion hétérozygote de SETD2 ; dans la majorité des noyaux on visualise un seul signal rouge, correspondant au locus de SETD2 en 3p21, contre deux signaux verts, représentant la région de contrôle conservée en 3p25. Di- et triméthylation de l’histone H3 (H3K36me2 et me3) : analyse immunohistochimique témoignant d’une perte de fonction de l’enzyme SETD2 (en sus de la délétion détectée par FISH, le séquençage a révélé la présence d’une mutation inactivatrice sur l’autre allèle) ; expression conservée de la forme diméthylée (me2) de la lysine 36 de l’histone H3, alors que la forme triméthylée (me3) est complètement absente dans les cellules tumorales (en présence de contrôles internes positifs : une petite structure vasculaire et quelques cellules réactives et stromales). AWS : associated with SET ; SRI : Set2 Rpb1 interacting ; Pol II : ARN polymérase II. |
|
SETD2 – structure et fonction La protéine SETD2 présente trois domaines conservés [6] (Figure 1A): le trio AWS (associated with SET)-SET-PostSET, le domaine WW et le domaine Set2 Rpb1 interacting. Le premier domaine catalyse habituellement le transfert d’un ou plusieurs groupes méthyles à des résidus lysine ou arginine d’histones ou d’autres protéines. Le deuxième domaine est impliqué dans des interactions protéine-protéine et le troisième domaine interagit spécifiquement avec la forme hyperphosphorylée en C-teminal de Rpb1, la plus grande sous-unité de l’ARN polymérase II. La fonction la mieux caractérisée de SETD2 est l’ajout d’un groupe méthyle à l’histone H3 diméthylée H3K36me2. Lors de la transcription, SETD2 est recrutée par la polymérase II au niveau des sites transcriptionnellement actifs pour y déposer sa marque [6]. Celle-ci sera ensuite lue par d’autres protéines la reconnaissant spécifiquement et dont l’action induira le résultat fonctionnel. La présence de l’histone H3 tri-méthylée H3K36me3 facilite l’élongation, participe à la sélection des exons, à l’épissage des introns et évite l’utilisation de sites d’initiation cryptiques de la transcription. La rétention d’introns et la transcription à partir de sites cryptiques sont des éléments majeurs de la tumorigenèse, conduisant à l’inactivation de gènes comme TP53 (tumor protein 53) ou la phosphatase PTEN (phosphatase and tensin homolog). La triméthylation de la lysine 36 de l’histone H3 (H3K36me3) augmente également lors de la transition des phases G1/S du cycle cellulaire. Son absence due à l’inactivation de SETD2 empêche la machinerie de réparation de l’ADN de localiser les sites endommagés lors de la réplication de l’ADN, ce qui résulte en une accumulation d’erreurs et une instabilité génomique [6]. Une autre fonction de SETD2 découverte récemment, est la triméthylation de la lysine 40 de l’α-tubuline. Cette marque épigénétique apparaît sur le fuseau mitotique [7]. En cas d’inactivation de SETD2, l’absence de cette marque perturbe la répartition des chromosomes en anaphase ainsi que la cytocinèse, deux processus essentiels au passage de l’information génétique durant la mitose. SETD2 est donc à la fois impliquée dans le remodelage de la chromatine et l’organisation du cytosquelette (Figure 1B). |
Les carcinomes à cellules claires du rein (CRcc) sont caractérisés par une perte récurrente du bras court du chromosome 3 en 3p21. L’identification du gène SETD2 au niveau de ce locus a conduit à sa caractérisation, en 2010, comme gène suppresseur de tumeur, inactivé dans environ 11 % des cas de CRcc et dans des lignées cellulaires dérivées de ces tumeurs [6]. L’inactivation de SETD2 par des mutations bialléliques ou par la mutation d’un allèle avec la perte de l’autre allèle a été ensuite observée chez des patients atteints de leucémie aiguë. Depuis lors, des mutations de SETD2, la plupart inactivatrices (stops, décalages du cadre de lecture ou délétions du locus), sont mises en évidence dans un nombre croissant de tumeurs solides (Tableau I). Du point de vue clinique, une corrélation inverse entre le niveau d’expression de SETD2 et le stade tumoral a été observé dans une étude sur le cancer du sein [5]. Chez les patients atteints de CRcc, les mutations somatiques de SETD2 sont associées à un plus grand risque de rechute [6]. De plus, on observe une hétérogénéité de la perte de la marque H3K36me3 au sein du tissu tumoral, mais aussi une diminution plus fréquente de celle-ci dans les métastases, comparées à la tumeur primaire. Ces données suggèrent que l’inactivation de SETD2 favorise à la fois l’émergence et la croissance des tumeurs [8] (→).
(→) Voir la Synthèse de H. Chaib, m/s n° 8-9, août-septembre 2011, page 725
Tableau 1.
|
Mutations ponctuelles et petites insertions /délétions
|
Variation du nombre de copies
|
| TYPE DE CANCER |
% de cas mutés (nombre de cas testés) |
% de cas avec délétion (nombre de cas testés) |
| Cancer du rein |
7,23 (2408) |
0,78 (1027) |
|
| Carcinome rénal à cellules clairesa [5] |
10,49 (1478) |
1,33 (528) |
|
| Cancers pulmonaires |
1,25 (1838) |
0,25 (1185) |
|
| Adénocarcinome |
2,19 (775) |
0,49 (411) |
|
| Carcinome épidermoïde |
0,76 (655) |
ND |
|
| Carcinome à petites cellules |
0,83 (121) |
ND |
|
| Cancers cutanés |
5,23 (1223) |
0,16 (620) |
|
| Mélanome malin |
4,63 (820) |
0,19 (532) |
|
| Sarcomes |
|
|
| Sarcomes des tissus mous |
3,81 (683) |
ND |
|
| Sarcomes osseux |
0,84 (595) |
ND |
|
| Cancers gynécologiques |
|
| Carcinome séreux de l’ovaire |
1,08 (651) |
ND |
|
| Adénocarcinome de l’endomètre |
4,38 (548) |
0,19 (530) |
|
| Cancers digestifs |
|
|
| Cancer de l’oesophage |
1,39 (1155) |
0,45 |
|
| Adénocarcinome gastrique |
2,66 (826) |
ND |
|
| Adénocarcinome colorectal |
3,9 (1373) |
0,26 (389) |
|
| Adénocarcinome canalaire du pancréas |
0,78 (1279) |
ND |
|
| Tumeurs stromales gastro-intestinalesb [5] |
8,13 (123) |
ND |
|
| Cancer du seinc [5] |
2,34 (2222) |
0,006 (1745) |
|
| Cancer de la prostate |
1,4 (1282) |
ND |
|
| Tumeurs du système nerveux central |
1,02 (2251) |
0,27 (1093) |
|
| Cancers de la thyroïde |
0,43 (697) |
ND |
|
| Hémopathies malignes |
1,4 (4142) |
2,67 (15) |
|
| Leucémie myéloïde aiguë |
1,15 (871) |
ND |
|
| Leucémies lymphoblastiques aiguës |
3,23 (620) |
ND |
|
| Lymphome diffus à grandes cellules B |
2,89 (277) |
ND |
|
| Leucémie lymphoïde chronique |
0,5 (803) |
ND |
|
| Lymphome-leucémie à cellules T de l’adulte |
2,42 (372) |
ND |
|
| Lymphome T hépatosplénique [9] |
23,5 (68) |
0 (20) |
|
| Lymphome T intestinal monomorphe épithéliotrope [4] |
70,4 (27) |
30,8 (39) |
Fréquence des mutations et délétions de SETD2 dans une sélection de cancers. Donnees issues de COSMIC (catalogue of somatic mutations in cancer) et de references selectionnees. |
|
Conclusions et perspectives Nos données montrent pour la première fois l’implication de SETD2 dans le développement de lymphomes T matures. La très grande prévalence des mutations de SETD2 observée dans les LTIME (> 90 %) ainsi que leurs fréquences alléliques élevées suggèrent que ces mutations représentent un événement précoce et essentiel au développement tumoral. Une publication très récente rapporte également des mutations récurrentes de SETD2 dans 24 % des lymphomes T hépatospléniques [9], une autre forme de lymphome T dérivé de cellules T gamma/delta. L’inactivation de SETD2 semble donc fréquente dans les lymphomes T gamma/delta, dérivés du système immunitaire inné, tandis que les mutations dans d’autres régulateurs épigénétiques tels TET2, DNMT3A et IDH2 sont associées à des lymphomes du système immunitaire adaptatif. L’inactivation de SETD2 dans les cellules cancéreuses les rend particulièrement vulnérables à l’effet de la molécule AZD1775 (ou MK1775) qui interfère avec le métabolisme des déoxynucléotides triphosphate (dNTP). Par son action inhibitrice sur la kinase WEE1, ce composé entraîne la dégradation de RRM2 (ribonucleotide reductase regulatory subunit M2), une enzyme clef pour la synthèse des dNTP et dont l’expression dépend de la présence de la triméthylation de la lysine 36 de l’histone H3. Dans des cellules déficientes en SETD2 et donc pauvres en H3K36me3, où l’expression de RRM2 est déjà fortement diminuée, AZD1775 (MK1775) induit un appauvrissement en dNTP et la mort cellulaire. Cette molécule, actuellement testée dans plusieurs études cliniques de phase 2 pour des cancers solides, pourrait constituer une nouvelle option thérapeutique pour certains lymphomes T dont les LTIME. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
De Leval L, Gaulard P Pathology and biology of peripheral T-cell lymphomas . Histopathology. 2011; ; 58 : :49.–68. 2.
Vallois D, Dobay MPD, Morin RD, et al. Activating mutations in genes related to TCR signaling in angioimmunoblastic and other follicular helper T-cell–derived lymphomas . Blood. 2016; ; 128 : :1490.–1502. 3.
Couronné L, Bastard C, Bernard OA Mutations récurrentes des gènes RHOA et FYN dans les lymphomes T périphériques . Med Sci (Paris). 2014; ; 30 : :488.–492. 4.
Roberti A, Dobay MP, Bisig B, et al. Type II enteropathy-associated T-cell lymphoma features a unique genomic profile with highly recurrent SETD2 alterations . Nat Commun. 2016; ; 7 : :12602.. 5.
Foukas PG, de Leval L Recent advances in intestinal lymphomas . Histopathology. 2015; ; 66 : :112.–136. 6.
Li J, Duns G, Westers H, et al. SETD2: an epigenetic modifier with tumor suppressor functionality . Oncotarget. 2016; ; 7 : :50719.–50734. 7.
Park In Y, Powell Reid T, Tripathi Durga N, et al. Dual chromatin and cytoskeletal remodeling by SETD2 . Cell. 2016;; 166 : :950.–62. 8.
Chaib H, Prébet T, Vey N, et al. Une nouvelle classe de cibles thérapeutiques dans le traitement du cancer ? Histone methyltransferases. Med Sci (Paris). 2011; ; 27 : :725.–732. 9.
McKinney M, Moffitt AB, Gaulard P, et al. The genetic basis of hepatosplenic T-cell lymphoma . Cancer Discov. 2017; ; 7 : :369.–379. |