| |
| Med Sci (Paris). 33(3): 238–245. doi: 10.1051/medsci/20173303009.Les multiples facettes de l’autophagie au cours du développement Céline Jenzer1 and Renaud Legouis1* 1Institut de biologie intégrative de la cellule (I2BC), CEA, CNRS, Université Paris-Sud, Université Paris-Saclay, avenue de La Terrasse, 91198Gif-sur-Yvette Cedex, France |
Historiquement, l’autophagie a été identifiée, par microscopie électronique, comme un processus cellulaire déclenché dans des conditions de stress et tout particulièrement en réponse à une carence alimentaire. L’ensemble des observations réalisées a révélé que la macroautophagie (que nous appellerons autophagie dans cette revue) est un processus de dégradation du matériel cytoplasmique par fusion de vésicules à double membrane, les autophagosomes, avec le lysosome. De nombreux cribles génétiques ont ensuite permis l’identification d’une quarantaine de gènes, appelés ATG (autophagy-related gene), essentiels à la biogenèse des autophagosomes (Figure 1). La majorité de ces acteurs moléculaires sont recrutés de façon séquentielle et ne se lient que transitoirement aux autophagosomes. Les protéines de la machinerie autophagique peuvent être subdivisées en différents groupes fonctionnels [34] (→) :
(→) Voir la Nouvelle de E. Morel, page 217 de ce numéro
 | Figure 1.
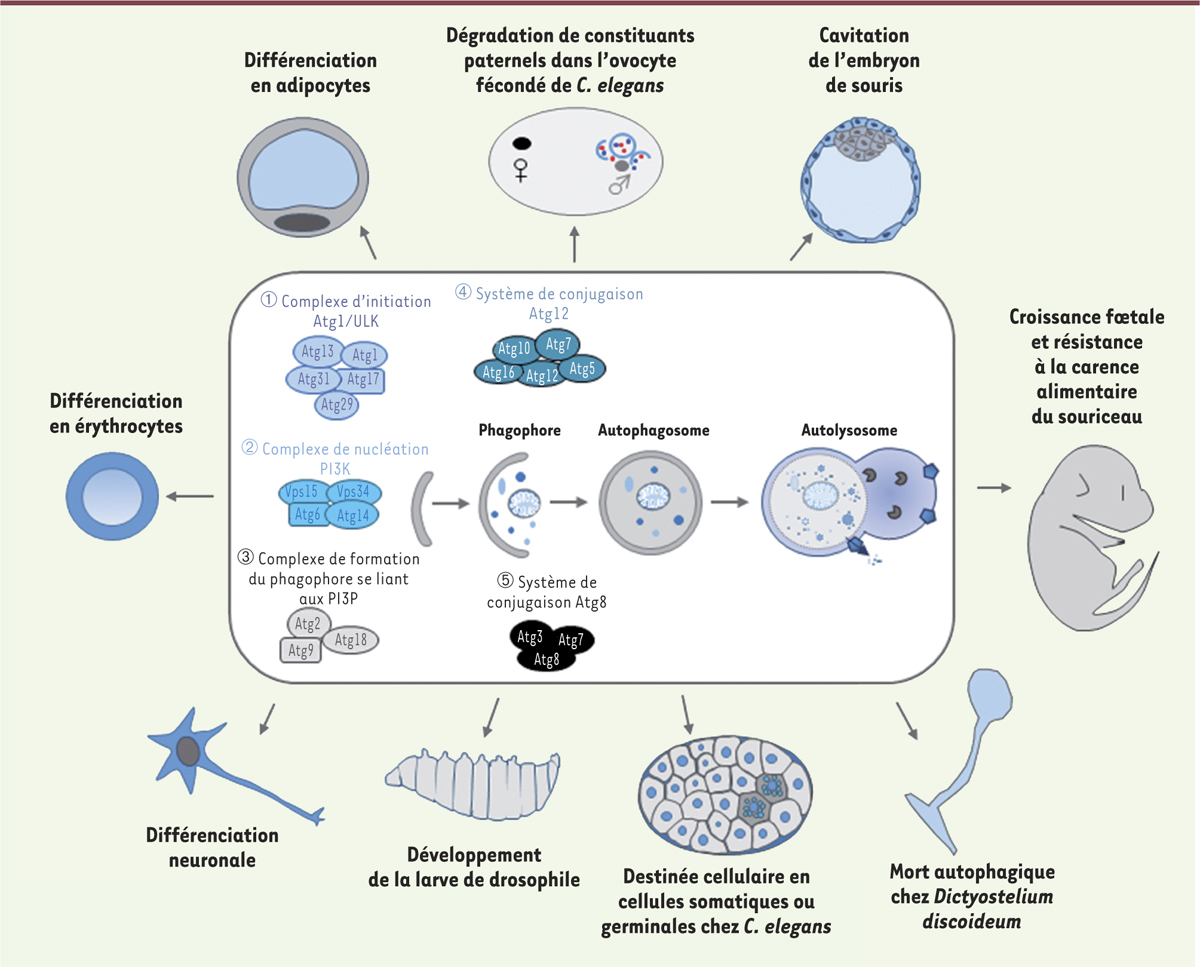
Diverses fonctions de l’autophagie au cours du développement. L’autophagie joue des rôles physiologiques variés durant différents stades du développement des mammifères, de la drosophile ou du nématode. Elle est en effet impliquée dans la dégradation de composants d’origine paternelle juste après la fécondation, dans la croissance du fœtus, dans la résistance à la carence alimentaire des souriceaux à la naissance, la cavitation de l’embryon de souris, la dégradation d’agrégats protéiques dans l’embryon de Caenorhabditis elegans et le développement de la larve de drosophile. Elle est également essentielle à l’organogenèse et à la différenciation de divers types cellulaires comme les neurones, les érythrocytes ou les adipocytes ainsi qu’à la mort autophagique chez Dictyostelium discoideum. Au centre de la figure, le schéma regroupe les différentes étapes de la voie autophagique. L’autophagie est initiée par la formation du phagophore qui s’étend progressivement autour du matériel cytoplasmique à dégrader. Cette étape d’élongation se termine par la formation de l’autophagosome. La maturation des autophagosomes nécessite leur fusion avec les lysosomes formant ainsi un autolysosome pour permettre la dégradation des constituants cytoplasmiques. Les étapes allant de l’induction de l’autophagie par la naissance du phagophore à la formation de l’autophagosome sont contrôlées principalement par cinq groupes de protéines : le complexe Atg1 impliqué dans l’initiation du processus, le complexe PI3K (phosphoinositide 3-kinase) permettant la nucléation du phagophore, le complexe de liaison aux PI3P (phosphatidylinositol 3-phosphate) pour la formation du phagophore constitué notamment d’Atg9, le système de conjugaison d’Atg12 et le système de conjugaison d’Atg8 contrôlant l’élongation du phagophore pour former l’autophagosome. Atg : autophagy-related gene ; ULK : Unc-51-like autophagy activating kinase. |
le complexe Atg1 qui régule l’induction de la formation de l’autophagosome, Atg9 jouant un rôle dans la délivrance des membranes nécessaires à l’expansion du phagophore ; le complexe PI3K (phosphoinositide 3-kinase) de classe III constitué notamment de la protéine Atg6, complexe qui est impliqué au stade de la nucléation de la membrane d’isolation et de la séquestration efficace des constituants cytoplasmiques ; enfin, les deux systèmes de conjugaison des protéines Atg12 et Atg8, importants pour l’extension du phagophore et la fermeture des membranes pour former l’autophagosome, comprenant notamment Atg16, Atg5, Atg3 et Atg7 (Figure 1). Il est maintenant clair que l’autophagie joue de multiples fonctions dans de nombreux processus physiologiques et pathologiques comme le contrôle de la qualité des protéines et des organites intracellulaires, la longévité, la suppression de la formation de tumeurs, l’élimination de pathogènes intracellulaires ainsi que la réponse immunitaire. Depuis quelques années, l’autophagie a également été impliquée dans différents processus développementaux notamment lors de l’embryogenèse, la différenciation cellulaire, l’organogenèse, le vieillissement ou la mort cellulaire programmée. En effet, l’inactivation du processus autophagique dans différents organismes modèles tels que les champignons, le nématode, la drosophile et la souris, entraîne l’apparition d’anomalies dans de multiples mécanismes impliqués dans le développement. Une déficience générale de l’autophagie ou l’absence de dégradation d’un acteur participant spécifiquement au développement par autophagie sélective peut être à l’origine de ces défauts. Cette revue présente les connaissances actuelles sur les rôles de l’autophagie dans le développement (Figure 1). À partir d’exemples pris essentiellement chez la souris, la drosophile, le nématode (Figure 2) ou le poisson zèbre, nous illustrerons le caractère pléiotropique de l’autophagie au cours du développement précoce et tardif, ou de la mort cellulaire programmée (voir Encadré). Pour des contraintes de place, nous n’aborderons pas ici ses implications dans la différenciation de nombreux types cellulaires et l’organogenèse.
 | Figure 2.
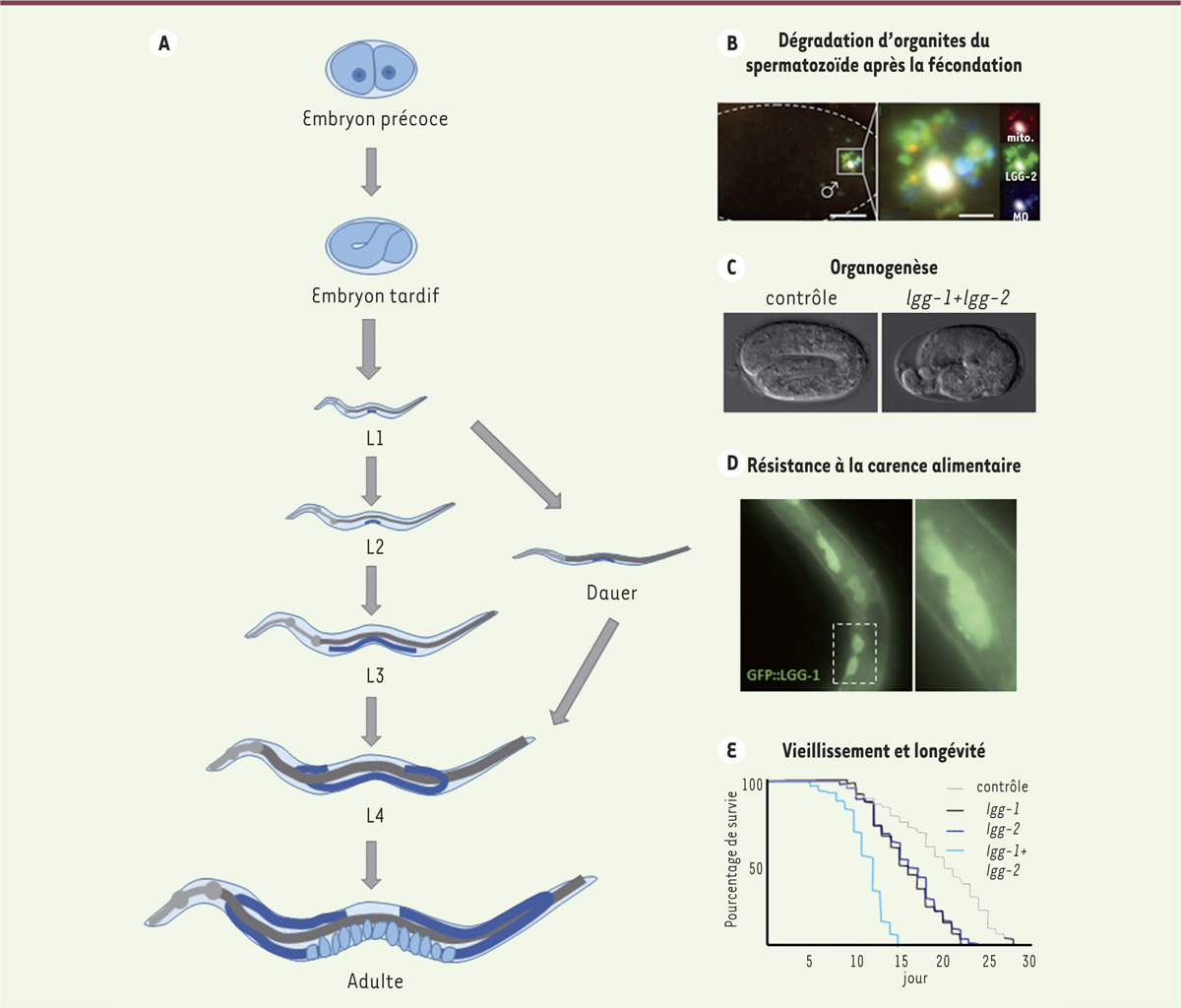
Implications physiologiques de l’autophagie au cours du développement de Caenorhabditis elegans. A. Le cycle de développement de Caenorhabditis elegans se compose d’un stade embryonnaire suivi de 4 stades larvaires (L1, L2, L3 et L4) avant le passage à l’âge adulte. La ligne gris clair représente le pharynx et celle en gris foncé, l’intestin du ver. La ligne bleu foncé schématise la lignée germinale, les ovocytes sont représentés en bleu clair. L’autophagie est essentielle au cours de nombreux processus du développement. Elle participe, entre autres, à la dégradation d’organites du spermatozoïde après la fécondation, à l’organogenèse, à la résistance à la carence nutritionnelle des larves L1, à l’entrée en stade dauer, au vieillissement et à la longévité des adultes. B. Juste après la fécondation, dans l’embryon précoce de Caenorhabditis elegans au stade 1 cellule, la protéine autophagique LGG-2, l’homologue d’Atg8, s’accumule au niveau des organites paternels à dégrader comme les mitochondries (mito, en rouge) et d’autres organites membranaires (MO, en bleu). C. Une absence des protéines autophagiques LGG-1/2 (par inactivation des gènes correspondants lgg-1/2) dans les embryons tardifs de Caenorhabditis elegans entraîne des défauts d’organogénèse. D. Des structures ponctuées correspondant à des autophagosomes apparaissent dans des cellules de larves L4 en conditions de carence alimentaire. Elles correspondent à la protéine LGG-1 rendue fluorescente par fusion avec la GFP (green fluorescent protein). E. Le graphique montre que la longévité des adultes diminue en absence des protéines autophagiques LGG-1/2 (par inactivation des gènes correspondants lgg-1/2). |
|
Éloge de la diversité : des multiples modèles pour comprendre les fonctions des protéines autophagiques au cours du développement Les processus développementaux sont, en très grande majorité, étudiés chez les métazoaires et dans différents modèles animaux. Beaucoup de travaux sur les fonctions autophagiques au cours du développement ont utilisé les organismes modèles comme le ver Caenorhabditis elegans, la mouche Drosophila melanogaster, le poisson Danio rerio, le poulet Gallus gallus et la souris Mus musculus. Plus ponctuellement, des études ont aussi utilisé les embryons de bovins. Des travaux sur les modèles végétaux [33] peuvent également apporter des données très complémentaires sur la conservation des relations entre autophagie et processus développementaux qui ont été décrits chez les animaux. Plusieurs modèles unicellulaires ont été source de résultats importants pour l’autophagie développementale, tels la sporulation chez la levure Saccharomyces cerevisiae ou les interactions cellulaires chez l’amibe Dictyostelium discoideum. Afin d’identifier les fonctions ancestrales et éventuellement non autophagiques de plusieurs protéines Atg, les protozoaires dont le parasite Toxoplasma gondii constituent également un modèle original. |
|
« Pourquoi, j’ai mangé mon père » L’autophagie est impliquée dès les premières étapes du développement comme l’indiquent plusieurs études récentes portant sur les cellules germinales et la fécondation. Elle semble importante pour la biogenèse de l’acrosome, un organite accolé au noyau du spermatozoïde et essentiel à la fécondation. L’absence d’Atg7 dans les cellules de la lignée germinale de la souris entraîne en effet des anomalies au niveau de la formation de l’acrosome et, en conséquence, une infertilité presque complète de l’animal [ 1]. Chez la souris femelle, l’autophagie permet également de préserver la fertilité en maintenant la viabilité des cellules dormantes de la lignée germinale au cours de l’embryogenèse [ 2]. Chez les métazoaires, l’information génétique mitochondriale est uniquement héritée de la mère. Cependant, lors de la fécondation, des constituants d’origine paternelle, en particulier des mitochondries, peuvent pénétrer dans l’ovocyte en même temps que le noyau du spermatozoïde. La dégradation des mitochondries paternelles est donc un évènement majeur du stade embryonnaire précoce empêchant les effets potentiellement délétères de mitochondries portant des mutations dans leur génome (hétéroplasmie mitochondriale). Chez Caenorhabditis elegans, nous avons montré que l’autophagie était impliquée dans la dégradation de ces organites d’origine paternelle lors de la fécondation (Figure 2) [3]. Des autophagosomes se forment, juste après la fécondation, autour des mitochondries et autres organites spécifiques des spermatozoïdes qui sont essentiels à leur fertilité. Dans les mutants de l’autophagie, ces constituants paternels persistent dans l’embryon tardif et la larve. Ce processus développemental stéréotypé a été appelé allophagie pour « allogeneic organelle autophagy ». Une protéine essentielle à la mitophagie, la prohibitine 2, a récemment été impliquée dans la dégradation des mitochondries paternelles chez Caenorhabditis elegans [4]. Chez la drosophile, dans les spermatides, les mitochondries fusionnent entre elles pour former une structure appelée « dérivé mitochondrial » qui semble être dégradée par autophagie après la fécondation [5]. Les données divergent pour les embryons de souris. Dans une étude, les mitochondries des spermatozoïdes qui entrent dans l’ovocyte au moment de la fécondation semblent immédiatement marquées par l’ubiquitine mais elles ne sont pas internalisées par des autophagosomes [6]. Dans une autre étude, la machinerie de la mitophagie apparaît néanmoins essentielle à l’élimination des mitochondries paternelles [7]. Enfin, il existe, chez le porc et le singe, un mécanisme d’élimination par autophagie sélective des mitochondries paternelles [8]. L’autophagie est donc l’un des processus dégradatifs qui participent à l’élimination des mitochondries paternelles mais son importance semble varier selon les espèces « Tout sur ma mère » L’autophagie est également impliquée dans la dégradation de constituants d’origine maternelle lors des premiers stades du développement embryonnaire. Elle permet la transition de ressources essentiellement maternelles vers de nouvelles, produites par le zygote. Ainsi chez la souris, l’activité de l’autophagie, faible dans les ovocytes, est fortement augmentée 4 heures après la fécondation ainsi qu’à la fin du stade 2 cellules jusqu’au stade 8 cellules [ 9]. L’absence d’ Atg5 dans des souriceaux homozygotes ( Atg5-/-) descendant de souris hétérozygotes pour Atg5 n’est pas létale au stade embryonnaire en raison de la présence des protéines maternelles. En revanche, l’élimination de cet apport maternel spécifiquement dans les ovocytes provoque une létalité embryonnaire entre les stades 4 et 8 cellules témoignant de l’implication essentielle de l’autophagie à ce stade développemental. Dans des embryons de bovins cultivés in vitro, l’induction de l’autophagie entraîne une augmentation du taux de survie des blastomères [ 10]. L’autophagie est également impliquée au début de l’embryogenèse du nématode Caenorhabditis elegans. En effet, elle permet l’élimination dans les cellules somatiques des granules-P, des composants ribonucléo-protéiques spécifiques de la lignée germinale [11]. Les protéines PGL-1 et PGL-3 (P-granules 1/3), composant les granules-P, sont impliquées dans la différenciation des cellules en cellules germinales. Elles sont dégradées par autophagie spécifiquement dans les cellules somatiques. Dans des mutants de l’autophagie, ces composants s’accumulent en agrégats dans les cellules somatiques. Chez Caenorhabditis elegans, l’identification de protéines réceptrices/adaptatrices (suppressor of ectopic P granule [SEPA] et sequestosome 1 [SQST-1]) a permis de montrer que les granules-P étaient dégradés par autophagie sélective dans les cellules somatiques en conditions physiologiques [12]. À noter que cette découverte a également conduit à l’identification par criblage génétique de mutants présentant un défaut de l’élimination des granules-P, de nouveaux gènes spécifiques aux métazoaires impliqués dans l’autophagie [13]. Le rôle précis de l’autophagie dans ces étapes précoces n’est pas totalement élucidé : elle pourrait participer à l’activation du programme génétique de l’embryon en dégradant des protéines et des organites accumulés dans l’ovocyte et permettre la transcription et la traduction des gènes zygotiques. |
« Allo Maman, ici Bébé » L’autophagie a de nombreuses fonctions après l’implantation de l’embryon. Le processus autophagique, en particulier la protéine Atg9, serait important pour la croissance du fœtus de souris [ 14]. En effet, la croissance fœtale de mutants homozygotes pour Atg9 est retardée par rapport à celle de fœtus sauvages ou hétérozygotes. L’absence d’autophagie fonctionnelle dans le fœtus entraîne ainsi un poids à la naissance plus faible des souriceaux. Une telle observation avait été réalisée précédemment sur des souris mutantes pour les gènes Atg3, Atg5 et Atg7 avec, en plus, une aggravation du phénotype lorsque les fœtus sont en condition d’hypertension maternelle [ 14]. L’hypertension maternelle au cours de la gestation a pour conséquence chez les fœtus une privation en nutriments et oxygène. Une hypothèse expliquant cette aggravation du phénotype serait que l’autophagie permettrait un apport d’énergie au fœtus dans ces conditions de carences alimentaires. Une étude récente met en évidence l’implication chez la souris de la protéine Atg13 dans le développement embryonnaire : les embryons mutants pour cette protéine sont significativement plus petits [ 15]. Dans l’embryon de poulet, l’autophagie, en particulier Atg7, est essentielle lors de la transition épithélio-mésenchymateuse (TEM, qui correspond à une transformation de cellules épithéliales vers une forme mésenchymateuse). L’inactivation de l’autophagie entraîne en effet un retard de développement en raison de défauts affectant le processus de TEM. La délamination de cellules1, lors de la formation de la ligne primitive déterminant l’axe longitudinal, permet de générer les trois couches cellulaires desquelles dériveront tous les organes : l’endoderme, le mésoderme et l’ectoderme [16]. L’autophagie est ainsi importante dans la morphogenèse cardiaque au cours du développement du poisson zèbre [17]. Son inactivation, notamment de Atg5 ou Atg7, a pour conséquence une augmentation de la létalité cellulaire, des défauts de structures cardiaques et une diminution du taux de survie des embryons. Enfin, l’autophagie, en particulier les protéines Beclin-1 (homologue d’Atg6 chez les mammifères) et LC3 (microtubule-associated protein 1A/1B-light chain 3), semble être impliquée dans le développement du placenta humain dans des conditions de carence en glucose et en oxygène [18], en protégeant les trophoblastes (des cellules placentaires) de l’apoptose induite par les stress que représentent l’hypoxie ou la restriction en nutriments. « Il faut manger pour vivre » Le passage rapide d’un stade développemental au suivant peut s’accompagner de variations très importantes d’apport en nutriments et en facteurs de croissance qui peuvent être source de stress pour la cellule. L’autophagie est alors essentielle à la production de ressources énergétiques de remplacement afin de protéger la cellule de ces conditions. Chez les mammifères, le processus autophagique joue un rôle important juste après la naissance. Il est fortement activé un ou deux jours après la naissance dans la majorité des tissus du nouveau-né [19]. L’inactivation d’Atg5 dans les souris ayant bénéficié de l’apport maternel du gène (voir plus haut) entraîne la mort des animaux juste après la naissance [19]. Il en est de même pour l’inactivation dans l’embryon d’Atg3, Atg7, Atg9, Atg12 et Atg16. Le niveau en acides aminés est diminué dans le plasma et les tissus de ces nouveau-nés déficients pour l’autophagie, ce qui suggère que l’autophagie permettrait le maintien d’une réserve en acides aminés à ce stade de développement. Au cours de cette période, les souris ne sont plus alimentées. En effet, les nutriments ne sont plus délivrés par la circulation placentaire mais pas encore par l’allaitement. L’autophagie permet donc de résister à cette carence alimentaire en générant des acides aminés et des acides gras à partir des composants cellulaires [9]. Plusieurs études ont démontré que l’autophagie est également impliquée dans le développement larvaire de Caenorhabditis elegans. Juste après l’éclosion des embryons, quatre stades larvaires, appelés L1, L2, L3 et L4, précèdent le passage au stade adulte (Figure 2). En cas de restriction nutritionnelle, les larves éclosent mais leur développement est arrêté au premier stade larvaire L1. Elles restent cependant viables pendant plusieurs jours. L’inhibition de l’autophagie diminue leur survie [20]. Le processus autophagique permet en effet de générer l’énergie nécessaire à la survie des animaux. De même, des conditions environnementales défavorables comme l’augmentation de la densité de la population, la limitation en nutriments et des températures élevées, induisent la formation d’un stade L3 alternatif, appelé dauer, permettant à la larve de résister. Cette étape de transformation requiert la participation de l’autophagie [20, 21]. L’autophagie est également essentielle à la métamorphose de la drosophile lors de la pupaison, c’est-à-dire lorsque les larves s’encapsulent dans la pupe avant de devenir adultes. À ce moment, elles cessent de se nourrir et l’autophagie induite par l’hormone ecdysone permet alors leur survie durant cette période de carence alimentaire [22]. « Vivre et laisser mourir » De nombreuses études ont montré que l’autophagie est impliquée dans la mort cellulaire programmée, un processus essentiel au remodelage des tissus ou au développement du système immunitaire. Ces changements cellulaires rapides, qui répondent à des stimulus environnementaux ou hormonaux, nécessitent des dégradations de composants cellulaires spécifiques. Les liens entre l’apoptose et l’autophagie sont particulièrement complexes. En effet, en fonction du type de cellule et du contexte, l’autophagie peut (1) induire une mort cellulaire indépendamment de l’apoptose ; (2) agir en collaboration avec l’apoptose ; (3) réguler l’apoptose ; et (4) être régulée par l’apoptose (Tableau I). La mort cellulaire autophagique (également appelée mort de type II) a été initialement décrite comme une mort associée à la présence de vésicules autophagiques. Mais l’observation d’autophagosomes au cours de la mort par apoptose a conduit à redéfinir cette mort de type II comme une forme indépendante de l’apoptose pouvant être bloquée par l’inhibition pharmacologique ou génétique de la machinerie autophagique. Chez l’amibe Dictyostelium discoideum, la mort autophagique est un processus développemental induit par carence alimentaire [ 23]. Chez Caenorhabditis elegans, la dégradation d’une partie des cellules germinales fait intervenir l’autophagie en collaboration avec l’apoptose [ 24].
Tableau I.
| Relations entre l’autophagie et l’apoptose |
Processus |
Espèces |
|
Pas d’interaction
|
Activation de l’oncogène H-Ras |
Cellules de mammifères |
|
Mort en carence alimentaire |
Dictyostelium discoideum
|
|
Ovogenèse en absence d’apoptose |
Caenorhabditis elegans
|
|
Intestin |
Drosophila melanogaster
|
|
|
Collaboration
|
Glande salivaires lors du développement larvaire |
Drosophila melanogaster
|
|
|
Activation de l’apoptose par l’autophagie
|
Ovogenèse |
Drosophila melanogaster
|
|
Dégradation de cellules apoptotiques |
Poulet |
|
Dégradation de neuroblastes Q |
Caenorhabditis elegans
|
|
Dégradation de facteurs anti-apoptotiques |
Cellules de mammifères |
|
|
Inhibition de l’apoptose par l’autophagie
|
Dégradation de facteurs pro-apoptotiques |
Cellules de mammifères |
|
|
Inactivation de l’autophagie par l’apoptose
|
Inhibition de Beclin-1 par Bcl-2 (B-cell lymphoma 2) |
Cellules de mammifères |
|
Clivage de Beclin-1 par la caspase-3 |
Cellules de mammifères |
|
Clivage d’Atg3 par la caspase-8 |
Cellules de mammifères |
Relations fonctionnelles entre l’autophagie et l’apoptose. Plusieurs études ont démontré qu’il existait des relations étroites et complexes entre autophagie et apoptose au cours du processus de mort cellulaire. En effet, l’autophagie joue des rôles contradictoires en fonction du tissu ou de l’organisme permettant la mort indépendamment de l’apoptose ou en collaboration. L’autophagie peut parfois réguler positivement ou négativement la mort. Réciproquement, l’apoptose semble elle aussi être capable de moduler l’autophagie. |
Plusieurs études réalisées chez la drosophile montrent que les relations entre autophagie et apoptose diffèrent en fonction du contexte cellulaire. Lors de la métamorphose, la dégradation de l’intestin par autophagie est indépendante de l’apoptose. L’inactivation de gènes Atg entraîne ainsi un retard de cette dégradation et l’expression forcée d’ULK-1 (Unc-51-like autophagy activating kinase 1), l’homologue d’Atg1 chez les mammifères, suffit à induire la mort cellulaire [25]. La dégradation des glandes salivaires requiert, en revanche, la conjonction de l’apoptose et de l’autophagie [26] et, plus tard, au cours de l’ovogenèse, l’autophagie contrôle la mort par apoptose en permettant l’activation de caspases [27]. Un rôle de l’autophagie dans la mort apoptotique lors de la cavitation de l’embryon de souris [28] et dans la rétine de poulet a également été identifié [29]. L’autophagie génèrerait l’ATP nécessaire à l’exposition de la phosphatidylsérine à la surface des cellules apoptotiques. Enfin, chez Caenorhabditis elegans, différentes équipes ont montré l’implication des protéines autophagiques dans l’apoptose de cellules embryonnaires et larvaires [30, 31]. L’autophagie est également impliquée dans deux autres types de mort cellulaire récemment décrits, la nécroptose (nécrose programmée induite par la liaison de ligands à leur récepteur) et l’autose, une nouvelle forme de mort cellulaire non apoptotique régulée par l’ATPase Na+/K+. Cependant, les liens de causalité entre protéines autophagiques et mécanismes d’induction de la mort ont encore été très peu étudiés. Il est aujourd’hui évident qu’il existe des différences essentielles entre mort cellulaire autophagique et mort cellulaire en présence d’acteurs de l’autophagie. Les mécanismes par lesquels l’autophagie participe à la mort de la cellule restent encore à élucider. |
Initialement décrite comme un processus permettant la survie en conditions de stress, il est aujourd’hui clairement établi que l’autophagie est impliquée dans divers processus développementaux mais également dans la différenciation et la mort cellulaire programmée. Le
Tableau II
récapitule, pour les protéines de la machinerie autophagique les plus étudiées, leurs fonctions dans les différentes étapes du processus de formation des autophagosomes ainsi que leurs rôles au cours du développement.
Tableau II.
| Complexe |
Protéine |
Fonction dans la cascade autophagique |
Implications physiologiques |
Espèces |
| Complexe d’initiation Atg1/ULK |
Atg1 |
Induction de la formation de l’autophagosome |
Développement larvaire |
D. melanogaster
|
|
Différenciation des érythroblastes |
Souris |
|
Longévité |
D. melanogaster
|
|
Longévité |
C. elegans
|
|
Différenciation neuronale |
Souris |
| Atg13 |
Activation de l’activité kinase d’Atg1 |
Développement embryonnaire |
Souris |
|
| Complexe de nucléation PI3K |
Vps34 |
Nucléation de la membrane d’isolation |
Différenciation des lymphocytes |
Souris |
| Atg6 / Beclin1 |
Augmentation de l’activité kinase du complexe PI3K |
Développement larvaire |
D. melanogaster
|
|
Longévité |
D. melanogaster
|
|
Développement du placenta |
Homme |
|
Morphogenèse cardiaque |
Souris |
|
Longévité |
C. elegans
|
|
Formation de la cavité amniotique |
Souris |
|
Tumorigenèse |
Souris |
|
| Vésicules Atg9 |
Atg9 |
Expansion du phagophore |
Croissance du fœtus |
Souris |
|
| Système de conjugaison d’Atg12 |
Atg12 |
Élongation du phagophore |
Survie à la naissance |
Souris |
|
Dégradation des protéines maternelles |
Souris |
|
Croissance du fœtus et survie à la naissance |
Souris |
|
Morphogenèse cardiaque |
Poisson zèbre |
| Atg5 |
Ancrage d’Atg12 aux membranes autophagiques |
Différenciation en adipocytes |
Cellules de mammifères |
|
Neurogenèse |
Souris |
|
|
|
Vieillissement |
D. melanogaster
|
|
|
|
Formation de la cavité amniotique |
Souris |
|
| Système de conjugaison d’Atg8/LC3 |
Atg8 |
Biogenèse des autophagosomes et recrutement de substrats autophagiques |
Dégradation de composants paternels |
C. elegans
|
|
Détermination destinée cellulaire |
C. elegans
|
|
Développement du placenta |
Homme |
|
Survie des L1 en carence alimentaire |
C. elegans
|
|
Survie des larves dauer |
C. elegans
|
|
Différenciation des cellules souches neuronales |
Souris |
|
Longévité |
D. melanogaster
|
|
Dégradation des neuroblastes Q |
C. elegans
|
| Atg3 |
Ancrage d’Atg8 aux membranes autophagiques |
Croissance du fœtus |
Souris |
|
Survie à la naissance |
Souris |
| Atg7 |
Ancrage d’Atg8 aux membranes autophagiques |
Transition épithélio-mésenchymateuse (TEM) |
Poulet |
|
Formation de l’acrosome |
Souris |
|
Morphogenèse cardiaque |
Poisson zèbre |
|
Croissance du fœtus et survie à la naissance |
Souris |
|
Différenciation des érythroblastes |
Souris |
|
Maintenance des cellules souches hématopoïètiques |
Souris |
|
Adipogenèse |
Souris |
|
Neurogenèse |
Souris |
|
Morphogenèse cardiaque |
Souris |
|
Formation du tube cardiaque |
Poulet |
|
Longévité |
C. elegans
|
Récapitulatif des rôles physiologiques des protéines autophagiques impliquées dans diverses étapes de la cascade autophagique. Les protéines autophagiques essentielles à différentes étapes du processus de formation des autophagosomes ont des fonctions physiologiques variées au cours du développement. Ce tableau récapitule pour les principales protéines autophagiques, leurs fonctions dans la cascade et leurs rôles principaux au cours du développement de différents organismes modèles. Atg : autophagy-related gene ; ULK : Unc-51-like autophagy-activating kinase ; PI3K : phosphoinositide 3-kinase ; LC3 : microtubule-associated protein 1A/1B-light chain 3. |
L’implication de l’autophagie dans ces processus développementaux a été démontrée principalement par des études descriptives fondées sur l’utilisation de mutants de gènes de l’autophagie. De nombreuses questions restent en suspens sur la fonction précise de ce processus, les voies d’induction, les substrats dégradés et les mécanismes moléculaires impliqués. L’autophagie canonique permet parfois la production de ressources énergétiques telles que des acides aminés ou des acides gras en conditions de stress et notamment de carences nutritionnelles. L’autophagie peut également être sélective et impliquée dans la dégradation de protéines spécifiques essentielles au développement. Il serait ainsi intéressant d’identifier les cibles précises de l’autophagie dans ces différents processus. Des rôles non canoniques ont été décrits pour des protéines autophagiques. Certaines sont en effet recrutées au niveau du phagosome au cours de la phagocytose de cellules apoptotiques indépendamment de la formation d’autophagosomes dans un mécanisme qui a été appelé LAP (LC3-associated phagocytosis) [32]. Ces observations démontrent donc la complexité des rôles de l’autophagie, qu’ils soient canoniques ou non, en fonction du processus ou du type cellulaire. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Les auteurs remercient l’ensemble de l’équipe pour les travaux de recherches présentés dans cette revue et les nombreux échanges scientifiques sur cette thématique. Merci particulièrement à Emmanuel Culetto pour sa relecture du manuscrit. Faute de place, de nombreuses études ne sont pas présentées ici et nous nous en excusons auprès de leurs auteurs. Nos travaux sont financés par l’Agence Nationale de la Recherche et la Fondation ARC (allocation de 4e année de thèse de Céline Jenzer) que nous remercions vivement pour leur soutien.
|
Footnotes |
1.
Wang H, Wan H, Li X, et al. Atg7 is required for acrosome biogenesis during spermatogenesis in mice . Cell Res. 2014; ; 24 : :852.–869. 2.
Gawriluk TR, Hale AN, Flaws JA, et al. Autophagy is a cell survival program for female germ cells in the murine ovary . Reprod Camb Engl. 2011; ; 141 : :759.–765. 3.
Al Rawi S, Louvet-Vallée S, Djeddi A, et al. Postfertilization autophagy of sperm organelles prevents paternal mitochondrial DNA transmission . Science. 2011; ; 334 : :1144.–1147. 4.
Wei Y, Chiang WC, Sumpter R, et al. Prohibitin 2 is an inner mitochondrial membrane mitophagy receptor . Cell. 2017; ; 168 : :224.–238.e10. 5.
Politi Y, Gal L, Kalifa Y, et al. Paternal mitochondrial destruction after fertilization is mediated by a common endocytic and autophagic pathway in Drosophila . Dev Cell. 2014; ; 29 : :305.–320. 6.
Luo SM, Ge ZJ, Wang ZW, et al. Unique insights into maternal mitochondrial inheritance in mice . Proc Natl Acad Sci USA. 2013; ; 110 : :13038.–13043. 7.
Rojansky R, Cha MY, Chan DC. Elimination of paternal mitochondria in mouse embryos occurs through autophagic degradation dependent on PARKIN and MUL1 . eLife. 2016; ; 5. 8.
Song WH, Yi YJ, Sutovsky M, et al. Autophagy and ubiquitin-proteasome system contribute to sperm mitophagy after mammalian fertilization . Proc Natl Acad Sci USA. 2016; ; 113 : :E5261.–E5E70. 9.
Tsukamoto S, Kuma A, Murakami M, et al. Autophagy is essential for preimplantation development of mouse embryos . Science. 2008; ; 321 : :117.–120. 10.
Song BS, Yoon SB, Kim JS, et al. Induction of autophagy promotes preattachment development of bovine embryos by reducing endoplasmic reticulum stress . Biol Reprod. 2012; ; 87 : (8), :1.–11. 11.
Zhang Y, Yan L, Zhou Z, et al. SEPA-1 mediates the specific recognition and degradation of P granule components by autophagy in C. elegans . Cell. 2009; ; 136 : :308.–321. 12.
Yang P, Zhang H. You are what you eat: multifaceted functions of autophagy during C. elegans development . Cell Res. 2014; ; 24 : :80.–91. 13.
Tian Y, Li Z, Hu W, et al. C. elegans screen identifies autophagy genes specific to multicellular organisms . Cell. 2010; ; 141 : :1042.–1055. 14.
Kojima T, Yamada T, Akaishi R, et al. Role of the Atg9a gene in intrauterine growth and survival of fetal mice . Reprod Biol. 2015; ; 15 : :131.–138. 15.
Kaizuka T, Mizushima N. Atg13 is essential for autophagy and cardiac development in mice . Mol Cell Biol. 2016; ; 36 : :585.–595. 16.
Lu WH, Wang G, Li Y, et al. Autophagy functions on EMT in gastrulation of avian embryo . Cell Cycle Georget Tex. 2014; ; 13 : :2752.–2764. 17.
Lee E, Koo Y, Ng A, et al. Autophagy is essential for cardiac morphogenesis during vertebrate development . Autophagy. 2014; ; 10 : :572.–587. 18.
Saito S, Nakashima A. Review. The role of autophagy in extravillous trophoblast function under hypoxia . Placenta. 2013; ; 34 ((suppl)) : :S79.–S84. 19.
Kuma A, Hatano M, Matsui M, et al. The role of autophagy during the early neonatal starvation period . Nature. 2004; ; 432 : :1032.–1036. 20.
Alberti A, Michelet X, Djeddi A, et al. The autophagosomal protein LGG-2 acts synergistically with LGG-1 in dauer formation and longevity in C. elegans . Autophagy. 2010; ; 6 : :622.–633. 21.
Meléndez A, Tallóczy Z, Seaman M, et al. Autophagy genes are essential for dauer development and life-span extension in C. elegans . Science. 2003; ; 301 : :1387.–1391. 22.
Tracy K, Baehrecke EH. The role of autophagy in Drosophila metamorphosis . Curr Top Dev Biol. 2013; ; 103 : :101.–125. 23.
Giusti C, Tresse E, Luciani M-F, et al. Autophagic cell death: analysis in Dictyostelium . Biochim Biophys Acta. 2009; ; 1793 : :1422.–1431. 24.
Wang L, Pan X, He L, et al. Vps41, a protein involved in lysosomal trafficking, interacts with caspase-8 . Acta Biochim Pol. 2013; ; 60 : :37.–42. 25.
Denton D, Shravage B, Simin R, et al. Autophagy, not apoptosis, is essential for midgut cell death in Drosophila . Curr Biol. 2009; ; 19 : :1741.–1746. 26.
Berry DL, Baehrecke EH. Autophagy functions in programmed cell death . Autophagy. 2008; ; 4 : :359.–360. 27.
Nezis IP, Shravage BV, Sagona AP, et al. Autophagic degradation of dBruce controls DNA fragmentation in nurse cells during late Drosophila melanogaster oogenesis . J Cell Biol. 2010; ; 190 : :523.–531. 28.
Qu X, Zou Z, Sun Q, et al. Autophagy gene-dependent clearance of apoptotic cells during embryonic development . Cell. 2007; ; 128 : :931.–946. 29.
Mellén MA, de la Rosa EJ, Boya P. The autophagic machinery is necessary for removal of cell corpses from the developing retinal neuroepithelium . Cell Death Differ. 2008; ; 15 : :1279.–1290. 30.
Huang S, Jia K, Wang Y, et al. Autophagy genes function in apoptotic cell corpse clearance during C. elegans embryonic development . Autophagy. 2013; ; 9 : :138.–149. 31.
Li W, Zou W, Yang Y, et al. Autophagy genes function sequentially to promote apoptotic cell corpse degradation in the engulfing cell . J Cell Biol. 2012; ; 197 : :27.–35. 32.
Green DR, Oguin TH, Martinez J. The clearance of dying cells: table for two . Cell Death Differ. 2016; ; 23 : :915.–926. 33.
Michaeli S, Galili G, Genschik P, et al. Autophagy in plants: what’s new on the menu? Trends Plant Sci. 2016; ; 21 : :134.–144. 34.
Morel E. La formation de l’autophagosome : un nouveau défi pour le biologiste cellulaire . Med Sci (Paris). 2017; ; 33 : :217.–220. |