| |
| Med Sci (Paris). 33(2): 123–126. doi: 10.1051/medsci/20173302003.Les règles de la biodiversité en temps profond Éric Lewitus1* and Hélène Morlon1 1Institut de Biologie, École Normale Supérieure, 46, rue d’Ulm, 75005Paris, France |
L’évolution vue à travers l’arbre du vivant La vie sur Terre est étonnamment diversifiée. On estime à plus de dix millions le nombre d’espèces, chaque espèce jouant un rôle unique dans les écosystèmes de notre planète. Ces espèces descendent toutes d’un même ancêtre commun, et sont apparues par accumulation des processus de mutation et spéciation. L’arbre du vivant (ou phylogénie) est une représentation du lien de parenté entre les espèces. À l’ère génomique, les phylogénies sont généralement construites en alignant des séquences de gènes homologues (descendant d’un même gène ancestral) d’espèces différentes et en reconstruisant la façon dont ces séquences ont évolué au fil du temps. En utilisant le principe de l’horloge moléculaire pour les gènes, et si possible également des spécimens fossiles datés [1] (→), on peut reconstruire non seulement la relation de parenté entre les espèces mais aussi leur temps de divergence (Figure 1A). Les phylogénies moléculaires datées ouvrent une fenêtre sur l’évolution de la diversité de la vie et sont nos guides les plus puissants pour comprendre comment les espèces ont évolué. Elles sont utilisées, par exemple, pour étudier l’évolution de vecteurs de maladies infectieuses [2], les réponses de la diversité biologique aux changements environnementaux [3], ou encore la diversification des traits des espèces qui leur confère leur rôle unique [4].
(→) Voir la Synthèse de R. Debruyne et V. Barriel, m/s n° 5, mai 2006, page 502
 | Figure 1.
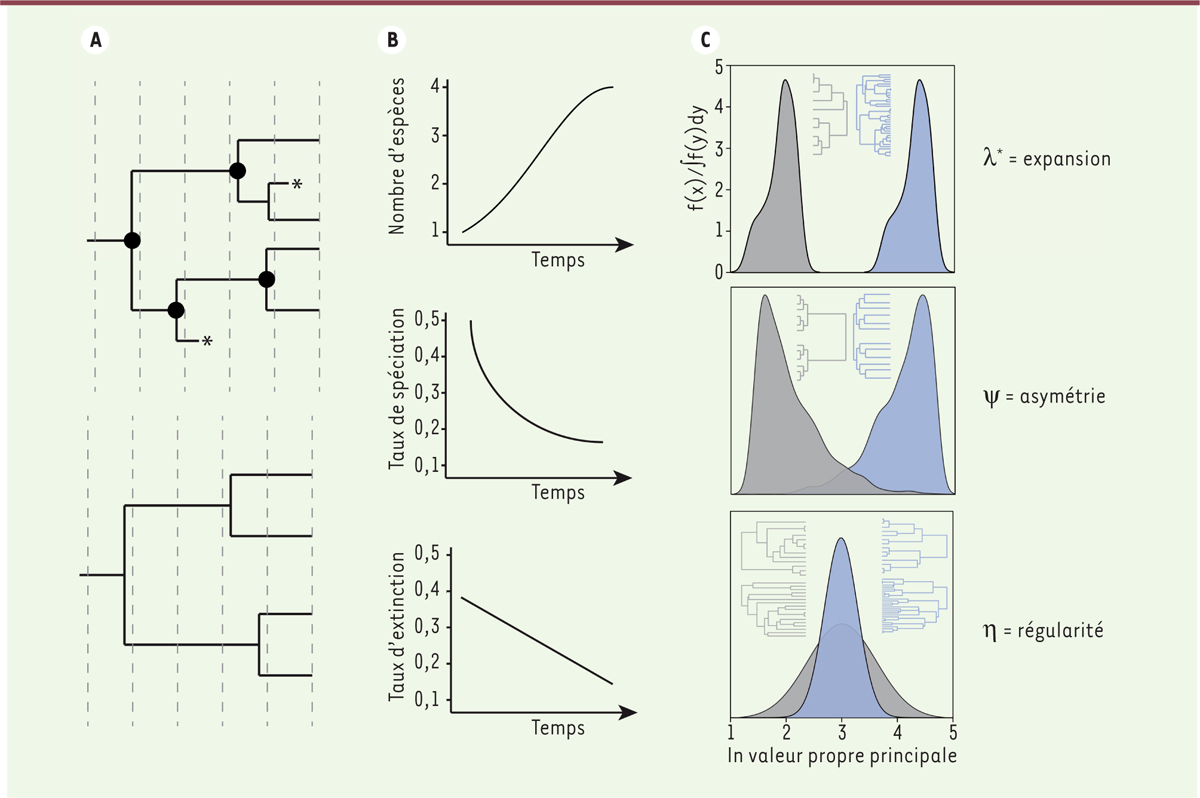
Inférer l’histoire évolutive du vivant à partir de phylogénies. (A, haut) À mesure que le clade évolue, les espèces divergent (points), s’éteignent (étoiles), ou survivent. (A, en bas) La phylogénie reconstruite à partir des espèces actuelles représente une histoire partielle des événements de spéciation dans ce clade. (B) La modélisation permet, à partir d’une phylogénie, de déduire des éléments de l’histoire évolutive d’un clade, comme la variation au cours du temps du nombre d’espèces, du taux de spéciation et du taux d’extinction ont varié au cours du temps. (C) Le profil de densité spectrale résume de manière exhaustive la forme d’une phylogénie. Les profils pour différentes espèces de clades peuvent être directement comparés et chaque profil peut être caractérisé par le logarithme (ln) de sa valeur propre principale (λ*), son asymétrie (ψ), et sa hauteur de pic (η). |
Les arbres phylogénétiques sont un enregistrement des événements de spéciation et d’extinction. Ils sont donc utiles pour comprendre comment la diversité des espèces d’un clade1 a évolué au cours du temps et pour analyser les différents facteurs écologiques et environnementaux qui ont influencé cette évolution [5]. Cependant, les phylogénies reconstruites à partir des espèces actuelles ne donnant qu’une image partielle de l’évolution passée, il est nécessaire de développer des modèles permettant de combler les informations manquantes. Au cours des dernières décennies, on a consacré beaucoup de travail au développement et à l’application de tels modèles. Cela a révélé, par exemple, que la diversification chez les oiseaux a accéléré au cours des 50 derniers millions d’années [6], que les vitesses de spéciation des mammifères sont supérieures dans les régions tropicales que dans les régions tempérées [7], et que la viviparité entraîne des radiations2 chez les poissons [8]. Notre compréhension actuelle de l’évolution est donc fondée à la fois sur les phénomènes microévolutionnaires de l’héritage génétique et sur l’étude de l’histoire phylogénétique des espèces. |
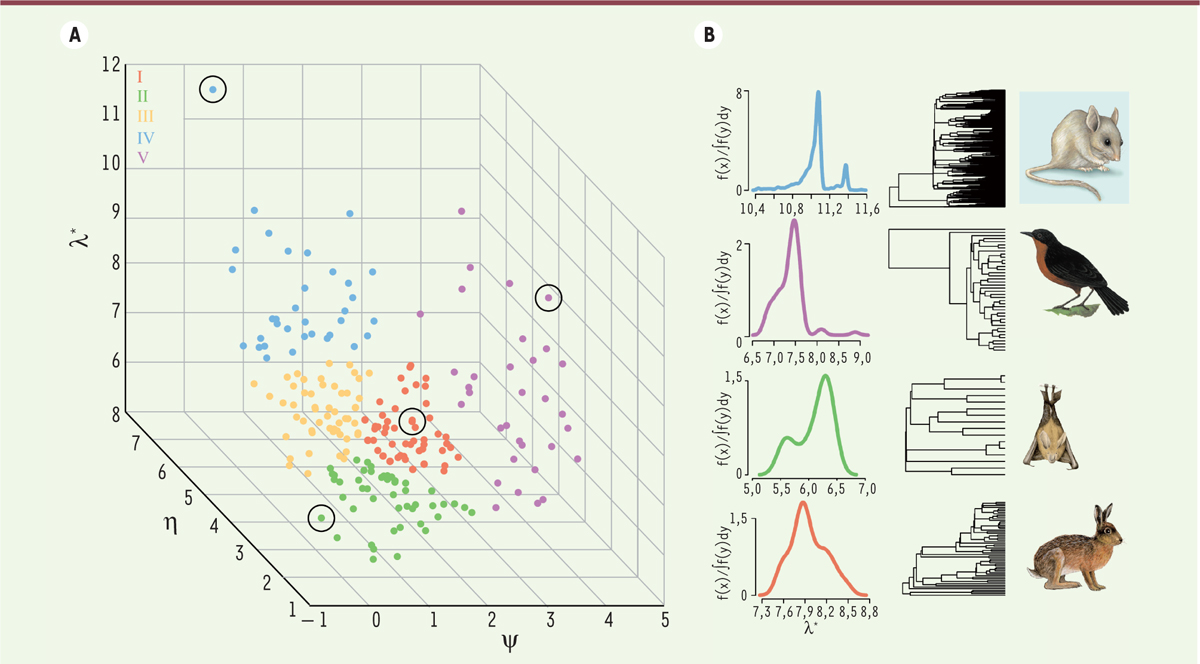
À la recherche de lois universelles de l’évolution du vivant Bien entendu, comme dans tout domaine d’étude, il y a un intérêt irrésistible à découvrir les principes généraux de la diversification des espèces. Existe-t-il des lois universelles sur la façon dont la biodiversité évolue ? Pourquoi certaines espèces prospèrent-elles et d’autres disparaissent-elles ? Y a-t-il des limites à la rapidité ou à la lenteur de la diversification des espèces ? Certains processus écologiques ou environnementaux, tels que les extinctions de masse ou des périodes climatiques extrêmes, laissent-ils des signatures discernables dans les phylogénies ? Ces questions sont presque aussi anciennes que la théorie de l’évolution elle-même. Les réponses à ces questions, cependant, sont insaisissables. Pour la plupart, c’est en raison des lacunes dans les méthodes disponibles pour comparer les histoires phylogénétiques des clades (groupes d’espèces). La plupart des méthodes phylogénétiques ont utilisé des statistiques sommaires, qui réduisent l’intégralité de l’histoire phylogénétique d’un clade à un seul nombre, ou des approches de model-fitting, qui sont idéales pour tester des hypothèses sur la dynamique de diversification dans les clades, mais moins pour comparer l’évolution des clades entre eux sans hypothèse a priori. Nous avons développé une nouvelle approche, qui nous a permis de résumer la totalité de l’information contenue dans la forme de la phylogénie par une distribution simple, appelée profil de densité spectrale phylogénétique [9]. Plus précisément, le profil de densité spectrale est le spectre des valeurs propres de la matrice laplacienne3 (graph Laplacian matrix), construite à partir des distances d’évolution entre les nœuds d’un arbre phylogénétique (Figure 1). Le profil peut être utilisé pour mesurer les similitudes entre les arbres phylogénétiques représentant différents clades et donc pour grouper ces clades sur la base de la similarité de leurs phylogénies. En outre, les profils peuvent être résumés par trois paramètres : le logarithme de la valeur propre principale, indicatif de l’expansion phylogénétique (λ*) ; l’asymétrie, indicative de la distribution des événements de ramification de la racine aux feuilles de l’arbre (ψ) ; et la hauteur du pic, indicative de l’hétérogénéité des longueurs de ramification. Ces trois paramètres permettent de placer les phylogénies dans un espace tri-dimensionnel, que nous appelons espace phylogénétique. Dans notre travail récent [10], nous avons utilisé cette approche pour comparer les phylogénies de 214 familles de vertébrés, couvrant environ 500 millions d’années et quelques 12 000 espèces. Nous avons identifié cinq principaux modes de diversification chez les vertébrés. En plaçant les phylogénies dans l’espace phylogénétique, nous avons caractérisé le paysage de diversification des vertébrés (Figure 2). Plus précisément, nous voyons que l’espace phylogénétique des vertébrés est fortement contraint, occupant seulement environ 40 % de tout l’espace possible. Différentes régions de l’espace occupé sont gouvernées par des processus évolutifs différents (par exemple, la dépendance à la température), de sorte que, où que se trouve une famille dans l’espace phylogénétique, nous pouvons (statistiquement) identifier le processus susceptible de l’avoir conduite là. Nous avons de plus utilisé l’espace phylogénétique pour tracer les trajectoires que les phylogénies prennent au fil du temps. En moyenne, les phylogénies des vertébrés au niveau familial prennent une trajectoire contrainte, passant par différents stades macroévolutionnaires, chacun caractérisé par une forme phylogénétique particulière et un processus évolutif. Remarquablement, cette trajectoire est différente pour les mammifères comparativement aux autres vertébrés : la diversification chez les mammifères est beaucoup plus susceptible d’être influencée par les tendances à long terme de la température.
 | Figure 2.
La forme de l’espace phylogénétique. (A) Phylogénies de 214 familles de vertébrés placées sur l’espace phylogénétique à l’aide de statistiques résumantes issues de leurs profils de densité spectrale (λ*, ψ, η). Sur la base de la forme de leurs phylogénies, on peut identifier cinq principaux groupes de clades, correspondant à cinq modes de diversification. Chaque point correspond à la phylogénie d’une famille de vertébrés et est coloré en fonction de son mode de diversification. (B) Profil, phylogénie et espèce représentative pour quatre clades appartenant à des groupes distincts (de haut en bas : Muridae, Acanthizidae, Nycteridae et Leporidae) (les images sont issues de http://animaldiversity.org/). |
|
À quel point l’évolution de la biodiversité est-elle contrainte ? Un regard attentif sur la façon dont la biodiversité est composée révèle un mélange de clades qui sont riches et pauvres en espèces, vieux et jeunes, homogènes et inhomogènes dans leur façon de se diversifier. La biodiversité semble donc désordonnée, ne se conformant pas à des lois d’organisation au-delà de celles décrites par la descendance par la sélection naturelle. Cependant, notre travail [10] révèle une règle générale d’organisation : la diversification des espèces chez les vertébrés est contrainte en raison de compromis dans les modes de diversification, aboutissant à une trajectoire évolutive moyenne à travers l’espace phylogénétique. Des processus écologiques et environnementaux particuliers peuvent amener les clades à s’écarter de la trajectoire moyenne vers d’autres régions de l’espace phylogénétique, mais ces écarts sont contraints. Au-delà du désordre apparent, il existe des règles générales nous permettant de comprendre comment la biodiversité émerge et évolue en temps profond. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Debruyne R, Barriel V. Évolution biologique et ADN ancien . Med Sci (Paris). 2006; ; 22 : :502.–506. 2.
Popinga A, Vaughan T, Stadler T, Drummond AJ. Inferring epidemiological dynamics with bayesian coalescent inference: the merits of deterministic and stochastic models . Genetics. 2015; ; 19 : :595.–607. 3.
Condamine FL, Rolland J, Morlon H. Macroevolutionary perspectives to environmental change . Ecol Lett. 2013; ; 16 ((suppl 1)) : :72.–85. 4.
Simões M, Breitkreuz L, Alvarado M, et al. The evolving theory of evolutionary radiations . Trends Ecol Evol. 2016; ; 31 : :27.–34. 5.
Morlon H. Phylogenetic approaches for studying diversification . Ecol Lett. 2014; ; 17 : :508.–525. 6.
Jetz W, Thomas GH, Joy JB, et al. The global diversity of birds in space and time . Nature. 2012; ; 491 : :444.–448. 7.
Rolland J, Condamine FL, Jiguet F, Morlon H. Faster speciation and reduced extinction in the tropics contribute to the mammalian latitudinal diversity gradient . PLoS Biol. 2014; ; 12 : :e1001775.. 8.
Helmstetter AJ, Papadopulos AST, Igea J, et al. Viviparity stimulates diversification in an order of fish . Nat Commun. 2016; ; 7 : :11271.. 9.
Lewitus E, Morlon H. Characterizing and comparing phylogenies from their Laplacian spectrum . Syst Biol. 2015; ; 65 : :495.–507. 10.
Lewitus E, Morlon H. Natural constraints to species diversification . PLoS Biol. 2016; ; 14 : :e1002532.. |