| |
| Med Sci (Paris). 33(1): 105–108. doi: 10.1051/medsci/20173301020.Chroniques génomiques - Modification ciblée du génome : une nouvelle approche prometteuse Bertrand Jordan1,2* 1UMR 7268 ADÉS, Aix-Marseille, Université/EFS/CNRS, Espace éthique méditerranéen, hôpital d’adultes la Timone, 264, rue Saint-Pierre, 13385Marseille Cedex 05, France 2CoReBio PACA, case 901, parc scientifique de Luminy, 13288Marseille Cedex 09, France MeSH keywords: Systèmes CRISPR-Cas, Protéine CFTR, Édition de gène, Ciblage de gène, Thérapie génétique, Génomique, Humains, Mutagenèse dirigée, Acides nucléiques peptidiques, génétique, méthodes, tendances, physiologie |
La large diffusion de la technique CRISPR-Cas9 [1] (→) et son emploi dans au moins deux essais cliniques de thérapie génique [2] (→) laissaient penser que cette méthode précise et efficace allait à elle seule révolutionner ce domaine. Mais voici qu’apparaît une approche très différente, fondée sur l’emploi d’entités appelées PNA (peptide nucleic acids), qui permet elle aussi une correction précise de l’altération présente dans un gène (et non simplement l’ajout d’un exemplaire fonctionnel) et semble présenter des avantages importants. Deux articles, dont les auteurs se recouvrent en bonne partie, détaillent son emploi pour corriger la mutation CFTR (cystic fibrosis transmembrane conductance regulator, responsable de la mucoviscidose) chez la souris [3] et, plus récemment, un gène de bêta-globine muté (responsable de la thalassémie), également dans un modèle souris [4]. Dans les deux cas, la correction a pu être réalisée in vivo avec une efficacité suffisante pour envisager des applications cliniques. Ce sont donc là des résultats importants qui méritent un examen approfondi.
(→) Voir la Nouvelle de H. Gilgenkrantz, m/s n° 12, décembre 2014, page 1066
(→) Voir la Chronique génomique de B. Jordan, m/s n° 11, décembre 2016, page 1035
|
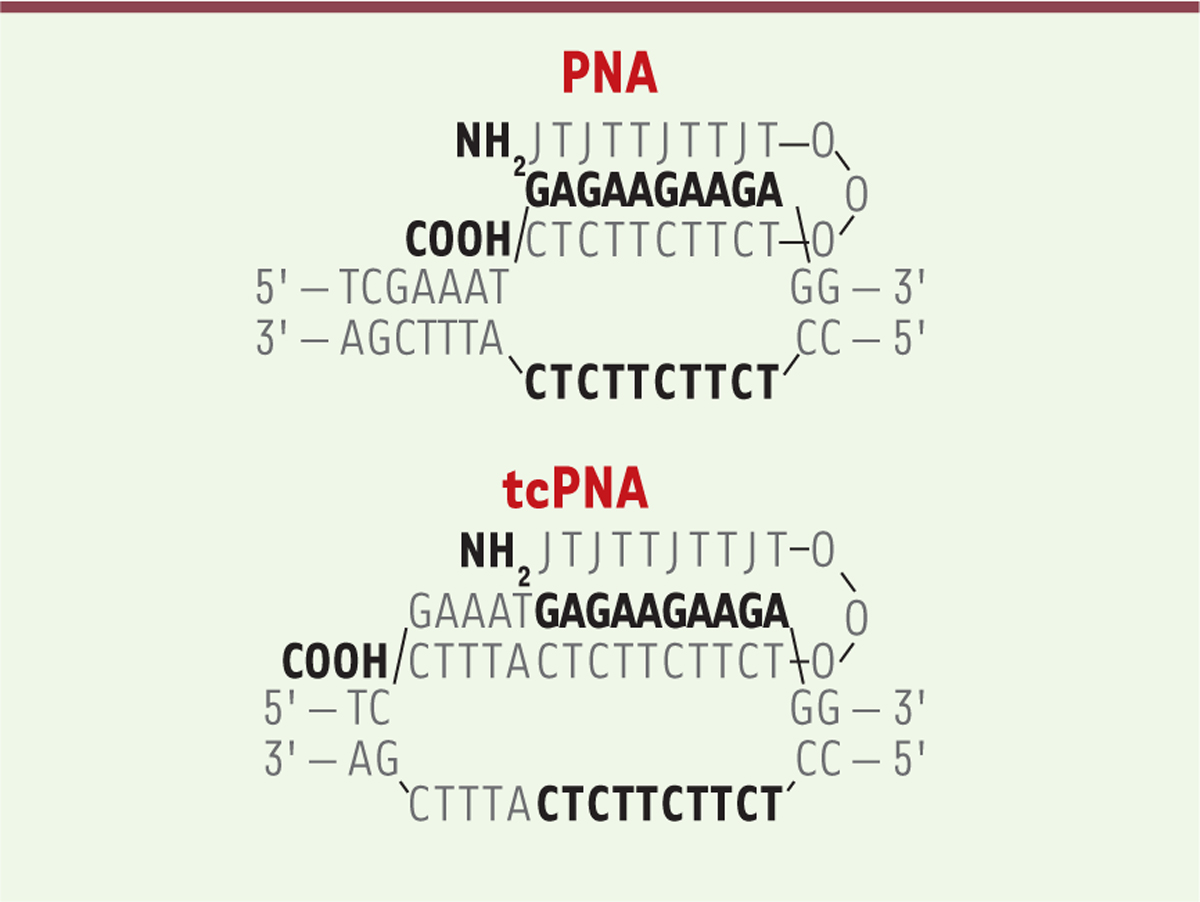
Les PNA, un outil aux multiples usages Comme leur nom l’indique, ces molécules sont à mi-chemin entre la protéine et l’ADN : les PNA sont formés d’un squelette de type peptidique (polyamide en général) sur lequel sont greffées les bases T, A, G et C. Celles-ci peuvent former des liaisons hydrogène classiques avec un brin d’ADN complémentaire, et les hybrides ainsi formés de manière spécifique sont plus stables que les hybrides ADN/ADN du fait que le squelette du PNA ne porte pas de charge, contrairement au squelette phosphate-ribose de l’ADN ou de l’ARN. De plus, ces molécules sont très résistantes à la dégradation dans l’environnement cellulaire et peuvent facilement être modifiées à leurs extrémités. Les PNA, dont l’apparition date d’un quart de siècle [5], fournissent des « super-sondes » qui ont trouvé de nombreux usages en recherche et dans le domaine du diagnostic [6, 7]. Dans le cas présent, il s’agit d’exploiter leur capacité à se fixer spécifiquement en un point donné de l’ADN cellulaire, et à y former une triple hélice qui déclenche, à son voisinage, les mécanismes d’excision et de réparation homologue (HDR, homology directed repair). Pour ce faire on utilise un PNA qui comporte deux parties reliées par un linker : la partie COOH-terminale (rappelons que la chaîne est de type peptidique) ouvre la double hélice et se fixe de manière classique (liaisons Watson-Crick) au brin complémentaire de l’ADN cellulaire (grâce à sa stabilité supérieure à celle du duplex ADN/ADN), tandis que la partie NH2-terminale se fixe par liaison de type Hoogsteen1 à ce duplex (Figure 1). On a donc une ouverture locale de l’ADN, avec d’un côté une triple hélice très stable (appelée PNA clamp), de l’autre un simple brin non apparié. La présence de cette structure déclenche alors les mécanismes cellulaires d’excision, qui favorisent ensuite la recombinaison avec un segment d’ADN introduit en même temps que le PNA. La première démonstration remonte à plus de dix ans [8], mais l’efficacité de la modification était alors très faible, et il a fallu de nombreux perfectionnements pour aboutir à des résultats permettant d’envisager des applications cliniques. L’un de ces changements consiste en l’allongement de la séquence du PNA à son extrémité COOH pour ajouter quelques bases dénommées tail clamp qui forment une double hélice adjacente à la triple hélice, augmentent la spécificité et favorisent le déclenchement des mécanismes d’excision [9]. On passe ainsi du PNA au tcPNA (Figure 1).
 | Figure 1.
Fixation d’un PNA (en haut) et d’un tcPNA (en bas) sur l’ADN cellulaire. Le PNA ouvre le double brin, se fixe sur un brin en formant une double hélice Watson-Crick, et se replie grâce au linker (O-O-O) pour se fixer (en orientation parallèle) sur cette double hélice et former un triplex. « J » indique la pseudocytosine qui remplace la cytosine et favorise la formation de la triple hélice. « O » figure une unité du linker flexible (acide 8-amino-2,6-dioxaoctanoïque). PNA : peptide nucleic acids ; tc : tail clamp. |
|
Correction de la mutation F508del dans le gène CFTRIl s’agit bien sûr de la plus fréquente des mutations responsables (à l’état homozygote) de la mucoviscidose, que l’on cherche à corriger par la méthode indiquée ci-dessus. Dans le travail que je vais brièvement décrire [3], les auteurs ont introduit une modification importante pour augmenter l’efficacité de la correction : ils utilisent des tcPNA qui ont été incorporés, en même temps que l’ADN « donneur » portant la séquence corrigée, dans des nanoparticules biodégradables PLGA (poly[lactic-co-glycolic] acid) dont la surface a été modifiée en y conjuguant des peptides qui favorisent leur pénétration dans les cellules [10]. Cette combinaison leur permet de gagner un ordre de grandeur en efficacité de correction. Dans un premier temps, des cellules d’une lignée d’épithélium bronchique humain portant la mutation ont été traitées par des nanoparticules contenant un tcPNA ciblé au voisinage de la mutation F508del ainsi qu’un ADN « donneur » long de 50 nucléotides et portant la séquence que l’on veut introduire. La fréquence de cellules corrigées (vues par PCR, et confirmées par séquençage) est d’environ 1 %, et pour les clones « corrigés », la fonction d’efflux de chlore est rétablie. Une deuxième série d’expériences utilisant les nanoparticules modifiées pour améliorer leur pénétration donne cette fois une efficacité de 7 %, qui peut être portée à 25 % par des traitements répétés : on arrive donc à des valeurs tout à fait respectables. Les auteurs sont alors passés aux expériences in vivo, sur la souris. Il existe en effet une lignée de souris portant la délétion F508 dans le gène de la protéine CFTR murine ; leur phénotype (à l’état homozygote) est beaucoup moins marqué que chez l’homme mais néanmoins mesurable au niveau du transport du chlore. Il a fallu bien sûr redéfinir le tcPNA et l’ADN donneur en fonction des séquences murines ; les souris ont alors été traitées par application intranasale de nanoparticules PLGA modifiées contenant le tcPNA et l’ADN donneur. Des mesures non invasives ont alors montré un rétablissement partiel du transport du chlore ; un séquençage intensif (deep sequencing) de l’ADN d’épithélium nasal a montré un taux de correction de 5,7 % pour ce tissu, et de 1,2 % pour le poumon. Les différents témoins négatifs (notamment avec des tcPNA ciblant d’autres gènes) ont bien donné les résultats attendus. Enfin, un ensemble de tests approfondis recherchant des modifications parasites (off-target effects) ou des coupures non spécifiques n’ont montré aucun phénomène de ce type. Au total, cet article détaillé et comportant de nombreux contrôles2 montre de manière convaincante que la correction de mutations par ciblage de PNA est réalisable in vitro et même in vivo, avec une efficacité suffisante pour commencer à envisager des applications cliniques et une absence apparente d’effets secondaires. On aimerait cependant en avoir une démonstration dans un domaine où il y ait réellement correction d’une pathologie : c’est ce qu’apporte l’article suivant. |
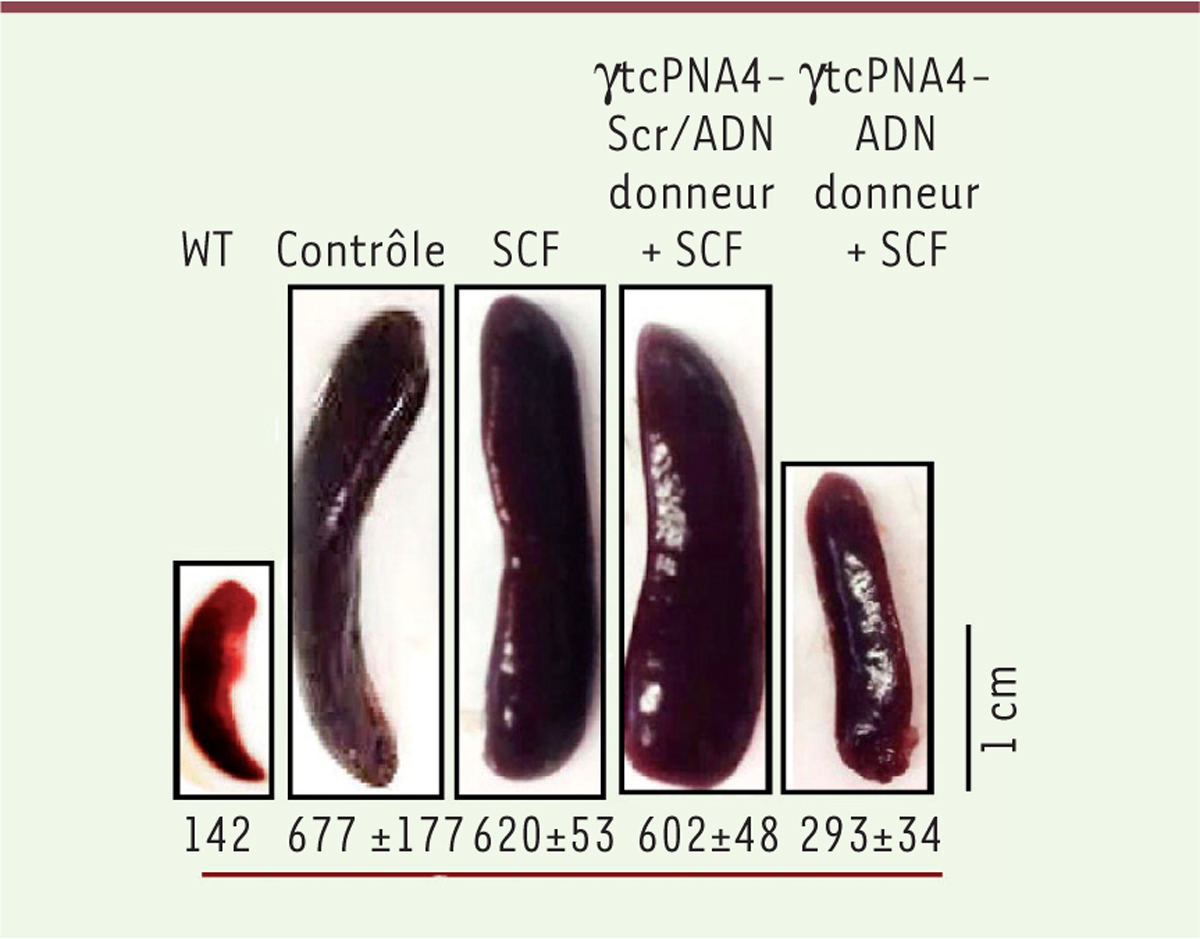
Correction de la bêta-thalassémie Le deuxième article [4], paru fin octobre 20163,, porte cette fois sur la correction chez la souris d’une mutation responsable de la bêta-thalassémie, la mutation IVS2-654 située dans un intron, qui empêche l’épissage correct de l’ARN messager de la globine. Les réactifs utilisés sont pour l’essentiel les mêmes que dans l’article précédent [3] à part une substitution en position gamma sur le PNA qui augmente encore son affinité pour l’ADN. Dans toute la première partie du travail, les auteurs utilisent des souris portant un transgène GFP (green fluorescent protein) dans lequel a été inséré l’exon 2 de la globine humaine (portant la mutation IVS2-654), de sorte que seules les cellules « corrigées » peuvent produire la protéine et présenter une fluorescence : on a ainsi un système rapporteur d’emploi aisé permettant de suivre la correction de la mutation. Le traitement ex vivo de cellules de moelle osseuse provenant de ces souris corrige la mutation avec une efficacité de 1,6 % ; ce chiffre monte à 7,2 % si l’on sépare et traite les cellules exprimant à leur surface le marqueur CD117 qui correspond à la protéine c-Kit. Si l’on traite alors les cellules par le ligand de c-Kit, la cytokine SCF (stem cell factor), l’efficacité monte à 15 %. Les auteurs ont fait l’hypothèse que SCF stimule la réparation d’ADN et ont vérifié expérimentalement ce point. À partir de ce moment, ils peuvent ajouter à leur protocole l’emploi de SCF (qui est commercialement disponible) et ainsi augmenter l’efficacité de la correction. La suite du travail est effectuée sur un modèle de souris thalassémique. Il s’agit d’animaux chez lesquels le gène murin de la bêta-globine a été remplacé par un gène humain portant la mutation IVS2-654. Les souris homozygotes ne sont pas viables, les hétérozygotes ont un phénotype thalassémique modéré avec des paramètres cliniques bien définis. Les animaux sont traités par quatre injections intraveineuses successives de SCF et de nanoparticules, espacées de deux jours. Seules les souris qui ont reçu des nanoparticules contenant le gamma-tcPNA et l’ADN donneur montrent une amélioration mesurable de l’anémie (augmentation de la concentration d’hémoglobine, amélioration de la morphologie des cellules sanguines, importante correction de la splénomégalie) (Figure 2). Le séquençage de la moelle osseuse totale montre une fréquence de correction proche de 4 %, avec une fréquence off target totale (pour les sept séquences les plus semblables à la séquence-cible) de 0,0032 %. Pour terminer, les auteurs ont également traité des cellules humaines CD34+ et obtenu une fréquence de correction de 5 % (et, off target, de 0,000012 % pour les six sites les plus homologues).
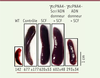 | Figure 2.
De gauche à droite, rate des souris normales, traitées avec des nanoparticules vides (contrôle), plus la cytokine SCF (SCF), avec l’ensemble des réactifs mais une séquence de PNA brouillée (scrambled, Scr), et avec l’ensemble des réactifs. On voit que la correction de quelques % des cellules réduit considérablement la splénomégalie. En bas, masse des rates (mg) (moyenne de 3 souris sauf pour WT). PNA : peptide nucleic acids ; SCF : stem cell factor ; WT : wild type (extrait partiel de la figure 4 de [ 4], licence © Creative Commons). |
Avec ce modèle souris qui semble très proche de la situation humaine, on obtient donc par un procédé simple (injection intraveineuse) la correction d’une proportion significative des cellules sanguines avec une récupération partielle du phénotype normal. On est donc assez proche d’une situation d’intervention clinique, même s’il reste bien des étapes à franchir avant d’en arriver là. |
Une technique promise à un bel avenir ? Voici donc une nouvelle approche qui permet de cibler un gène muté et de corriger l’anomalie qu’il porte, tout comme la technique CRISPR-Cas9 [2]. Comme pour cette dernière, l’approche est générique : pour cibler un autre gène, il suffit de changer la séquence du PNA (et bien sûr, celle de l’ADN donneur). Un avantage important est que l’on n’introduit pas de nucléase dans les cellules traitées : le PNA se borne à ouvrir l’ADN en formant une triple hélice, et ce sont les mécanismes intrinsèques de la cellule (système d’excision et de réparation) qui vont se charger du reste. On peut donc espérer que les coupures parasites (effets off target) seront très peu fréquentes, et cela semble bien être le cas à voir les valeurs avancées [4]. Cela reste bien sûr à confirmer. Au passif de cette technique de genome editing, il faut noter que l’obtention d’une efficacité de quelques pourcents a nécessité de multiples mises au point, notamment un design sophistiqué des PNA (du PNA en deux parties reliées par un linker au tcPNA puis au gamma-tcPNA), et l’emploi de nanoparticules modifiées en surface pour assurer la pénétration des réactifs dans les cellules. Même si la synthèse de PNA peut être assurée par des entreprises4,, la fabrication des entités très particulières employées dans ces travaux et leur inclusion dans des nanoparticules sophistiquées requièrent des compétences pointues. Cela laisse penser que la mise en œuvre de cette méthode sera plus délicate que celle du système CRISPR, et qu’elle ne connaîtra pas la même diffusion. Néanmoins, la démonstration apportée par ces deux articles de son efficacité pour une correction in vivo de mutations délétères [3, 4] laisse à penser que nous n’avons pas fini d’entendre parler des PNA en thérapie génique. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Gilgenkrantz H. La révolution des CRISPR est en marche . Med Sci (Paris). 2014; ; 30 : :1066.–1069. 2.
Jordan B. Les débuts de CRISPR en thérapie génique . Med Sci (Paris). 2016; ; 32 : :1035.–1037. 3.
McNeer NA, Anandalingam K, Fields RJ, et al. Nanoparticles that deliver triplex-forming peptide nucleic acid molecules correct F508del CFTR in airway epithelium . Nat Commun. 2015; ; 6 : :6952.. 4.
Bahal R, Ali McNeer N, Quijano E, et al. In vivo correction of anaemia in β-thalassemic mice by γPNA-mediated gene editing with nanoparticle delivery . Nat Commun. 2016; ; 7 : :13304.. 5.
Nielsen PE, Egholm M, Berg RH, Buchardt O Sequence-selective recognition of DNA by strand displacement with a thymine-substituted polyamide . Science. 1991; ; 254 : :1497.–1500. 6.
Ray A, Nordén B Peptide nucleic acid (PNA): its medical and biotechnical applications and promise for the future . FASEB J. 2000; ; 14 : :1041.–1060. 7.
Nielsen PE. PNA technology. Mol Biotechnol. 2004; ; 26 : :233.–248. 8.
Rogers FA, Vasquez KM, Egholm M, Glazer PM Site-directed recombination via bifunctional PNA-DNA conjugates . Proc Natl Acad Sci USA. 2002; ; 99 : :16695.–16700. 9.
Schleifman EB, Bindra R, Leif J, et al. Targeted disruption of the CCR5 gene in human hematopoietic stem cells stimulated by peptide nucleic acids . Chem Biol. 2011; ; 18 : :1189.–1198. 10.
Gupta A, Bahal R, Gupta M, et al. Nanotechnology for delivery of peptide nucleic acids (PNAs) . J Control Release. 2016; ; 240 : :302.–311. |