| |
| Med Sci (Paris). 32(10): 898–902. doi: 10.1051/medsci/20163210025.Chroniques génomiques- HGP-write : après la lecture, l’écriture ? Bertrand Jordan1* 1UMR 7268 ADÉS, Aix-Marseille, Université/EFS/CNRS, Espace éthique méditerranéen, hôpital d’adultes la Timone, 264, rue Saint-Pierre, 13385 Marseille Cedex 05, France 2CoReBio PACA, case 901, parc scientifique de Luminy, 13288 Marseille Cedex 09, France MeSH keywords: Coûts et analyse des coûts, ADN, Génome humain, Projet génome humain, Humains, Analyse de séquence d'ADN, biosynthèse |
Début juin 2016, un article dans la revue Science [1] proposait un projet très ambitieux, visant rien moins que la synthèse complète de l’ADN humain, une entreprise sans commune mesure avec la récente synthèse d’un génome bactérien minimal par l’équipe de Craig Venter [2] : 3,3 gigabases (Gb)1 au lieu d’une demie mégabase (Mb) ! Cet article avait été précédé par une réunion confidentielle organisée le 10 mai, qui avait donné lieu à quelques échos [3]. Les signataires ne sont pas des inconnus : on y trouve George Church, grand génomiste et co-développeur du système CRISPR-Cas9, et Jef Boeke, chef d’orchestre d’un projet de synthèse du génome de la levure sur lequel je reviendrai. Parmi les vingt-trois autres auteurs, plusieurs industriels (biotechnologie, logiciels, capital-risque), et de nombreux universitaires appartenant à des institutions prestigieuses (Harvard, John Hopkins, Columbia, Yale, New York, etc.), dont beaucoup de spécialistes de la bio-ingénierie et de la biologie des systèmes, mais aussi quelques cliniciens. Il semble donc qu’il faille prendre au sérieux ce projet – dont Le Monde s’est aussi fait l’écho [4] – en dépit de sa démesure. Démesure qui apparaît à l’aune des travaux effectivement réalisés jusqu’ici dans ce domaine : la construction d’une bactérie « minimale » portant 473 gènes sur un génome long de 531 kilobases (kb) a fait l’objet d’une chronique récente [2] ; rappelons qu’elle a demandé une dizaine d’années de travail à un laboratoire très performant. Et le projet levure (Saccharomyces cerevisiae) coordonné par Jef Boeke (Institute for systems genetics, Langone medical center, université de New York) associe une dizaine d’équipes à travers le monde et a impliqué une soixantaine de thésards pour réaliser, en sept ans, la synthèse du chromosome III de cet organisme [5] – environ trois cents kb2,. Il est amusant de remarquer que cette situation correspond précisément à celle qui prévalait lors des débuts du programme Génome (Figure 1) : en 1992, la plus longue séquence connue était justement celle du chromosome III de S. cerevisiae. La tâche – séquencer l’ensemble du génome humain – semblait pour beaucoup hors de portée ; pourtant elle fut réalisée en une dizaine d’années, et personne ne peut aujourd’hui nier qu’elle ait révolutionné la Biologie. Il est évidemment tentant, et les auteurs ne s’en privent pas, d’insister sur ce parallèle et d’affirmer que le projet HGP-write
3 impulsera une révolution dans les techniques de synthèse de l’ADN et aura de multiples retombées. Ce n’est pourtant qu’une hypothèse, à mon avis passablement optimiste…
 | Figure 1.
Le problème du séquençage, vu en 1992, alors que la plus longue séquence connue était celle du chromosome III de la levure. La séquence est supposée écrite dans des annuaires téléphoniques de 1 000 pages, chaque page portant 15 000 signes (d’après DOE primer on molecular genetics, juin 1992). |
|
Mais revenons aux buts annoncés par Boeke, Church et leurs nombreux collaborateurs [1]. La synthèse totale de l’ADN humain permettrait évidemment de le modifier à volonté et, en supposant qu’il reste fonctionnel, d’obtenir des lignées de cellules thérapeutiques résistantes aux virus et incapables de donner naissance à des cancers, d’accélérer la mise au point de vaccins et de médicaments grâce à des lignées et des organoïdes, et même de produire des organes transplantables car non immunogéniques. Il ne peut s’agir là que de projets à long terme : cela suppose de maîtriser la synthèse des 3 gigabases (Gb) de notre ADN, de savoir quelles modifications apporter à sa séquence pour obtenir les propriétés désirées et, bien sûr, d’être capable de l’introduire dans des cellules dont il prendrait le commandement. Des objectifs intermédiaires moins ambitieux (Pilot projects) sont donc présentés : ils vont de la synthèse de grands locus géniques (avec les séquences avoisinantes) pour comprendre leur régulation, à la construction de « petits » chromosomes comme le 21 (47 Mb, tout de même) et à diverses applications géniques, qui supposent néanmoins de résoudre le problème du transfert de ces grands ADN dans les cellules à traiter. Notons encore la construction de lignées cellulaires sécurisées (ultrasafe cells) présentant, grâce à leur ADN, résistance aux virus, au cancer, absence de prions, stabilité (élimination des éléments répétés) et incapacité à former des cellules germinales – lignées qui constitueraient le matériau de départ pour diverses thérapies cellulaires. Les auteurs évoquent enfin la synthèse d’un « génome humain de référence » portant en chaque point variable l’allèle le plus fréquent dans la population et permettant l’obtention des cellules correspondantes. Tout cela relève pour le moment de la science-fiction et semble être l’aboutissement d’un brainstorming intense lors de la réunion tenue en mai ; cela dit, il serait imprudent d’écarter ces perspectives sous prétexte qu’elles ne sont actuellement pas réalisables. En pratique, les auteurs espèrent réunir cent millions de dollars, à partir de sources « publiques, privées, philanthropiques et industrielles » pour lancer le projet dès 2016, et admettent que le coût total est difficile à estimer mais qu’il devrait être inférieur à trois milliards de dollars (le montant généralement admis pour le programme Génome). Il est en réalité impossible de donner un chiffre crédible à ce stade, car tout repose sur l’hypothèse d’une réduction massive du coût de la synthèse et de l’assemblage de grands segments d’ADN, réduction très probable mais dont l’ampleur et le rythme sont impossibles à déterminer à l’avance – et sur un pari quant à des étapes techniques cruciales. |
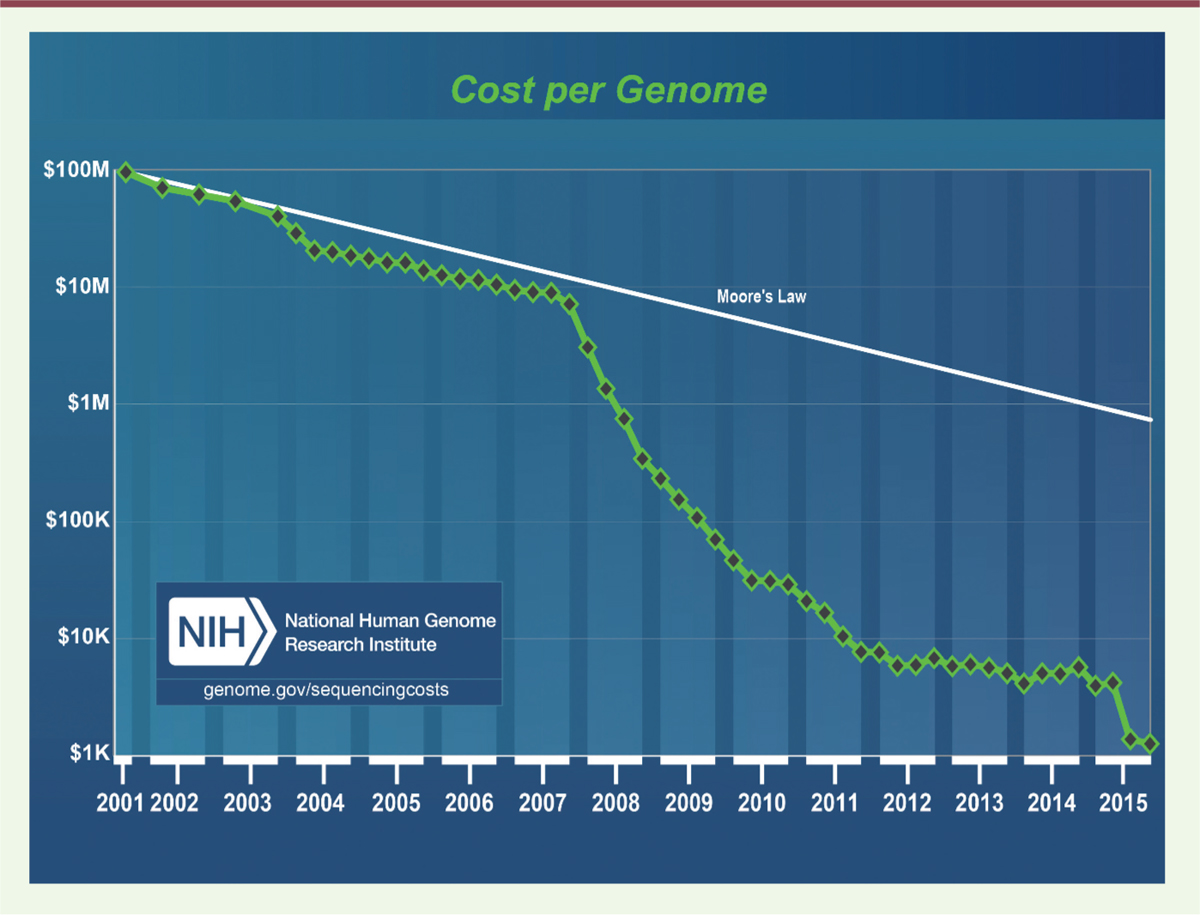
Les principaux problèmes techniques Le coût actuel de la synthèse d’ADN double brin, proposée par plusieurs entreprises4,, est de l’ordre de 0,50 dollars par paire de bases pour des segments de 10 kb, ce qui correspond à 1,5 milliards (toujours en dollars) pour un génome humain haploïde de 3 Gb. Et l’on fait abstraction ici de tout le travail d’assemblage, de vérification, du fait que les segments doivent être chevauchants etc. Il faut sans doute multiplier ce chiffre par 10 ou 100 pour s’approcher du coût réel. HGP-write requiert effectivement d’énormes progrès techniques et des baisses de coût vertigineuses. Certes, cela a été le cas pour le séquençage d’ADN, notons cependant que la plus forte décroissance n’a commencé que début 2008 (à cette époque, la lecture d’un génome coûtait encore 5 millions de dollars), bien après la fin du programme Génome, et que le « génome à 1 000 dollars » est tout récent5 (voir Figure 2).
 | Figure 2.
Coût du séquençage. Noter que la forte décroissance n’a commencé que début 2008, avec l’arrivée des nouvelles techniques de séquençage, six ans après la fin du programme Génome Humain (© graphique du NIH [National Institutes of Health]). |
Il faut noter aussi que nulle part dans l’article la question de la réintroduction dans des cellules de l’ADN synthétisé n’est abordée. Il s’agit pourtant d’un point essentiel : si Venter a choisi de travailler sur le mycoplasme Mycoplasma discoides, c’est en grande partie parce que cet organisme se prête bien au transfert de génomes (bactériens) entiers en raison de l’absence de paroi [2] ; de même, on sait depuis assez longtemps transformer la levure avec des chromosomes. Pour les cellules de mammifère, l’introduction de grands segments d’ADN reste très problématique. D’autant plus qu’il ne s’agit pas d’introduire de l’ADN nu, mais un complexe ADN/protéines ressemblant le plus possible à un chromosome. Remarquons à cet égard que le clonage de mammifères n’a jamais été réalisé avec de l’ADN, mais seulement par injection de noyaux entiers. On commence certes à parler de MAC (mammalian artificial chromosomes) [6], mais leur introduction dans des cellules par MMCT (microcell-mediated chromosome transfer) reste très délicate et limitée à des tailles de l’ordre de la mégabase. Et bien sûr, il ne sert à rien de synthétiser un génome humain si l’on ne peut pas observer son fonctionnement dans une cellule – d’ailleurs, comme pour le mycoplasme ou la levure, la réussite éventuelle d’une synthèse repose entre autres sur une construction par étapes avec vérification à chacune d’elles du bon fonctionnement du génome « semi-synthétique » déjà assemblé. En raison de ces lourdes hypothèques, le succès de HGP-write n’est donc pas garanti. |
L’ombre de l’humain synthétique L’annonce de HGP-write a fait un certain bruit et suscité des réactions mitigées, notamment de la part de Francis Collins (maintenant directeur des National Institutes of Health [NIH]), qui a mis en cause la faisabilité technique du projet et a souligné qu’il soulève de sérieux problèmes éthiques. Et, de fait, l’article de Science [1] consacre une place importante aux aspects éthiques et sociétaux, tout en s’en tenant à des considérations très générales. Cela pourrait sembler superflu pour les travaux tels qu’ils sont présentés, d’autant plus que leur aboutissement n’est pas pour demain ; mais bien sûr, la perspective d’un ADN humain synthétique et de son introduction dans des cellules ouvrirait, si elle était réalisée, celle de la fabrication d’une cellule germinale ou d’un embryon animés par un ADN construit au laboratoire. C’est déjà apparent en filigrane lorsque l’article évoque des lignées résistantes au cancer ou aux virus : propriétés intéressantes pour une cellule, certes, mais combien plus pour un organisme ! La personnalité de George Church renforce ces inquiétudes : il est assez proche du courant transhumaniste6, et a publié en 2012 un livre au titre éloquent : Regenesis: how synthetic biology will reinvent nature and ourselves [7] (Figure 3). Ces craintes sont notamment exprimées dans un article de la très technophile MIT Technology Review [8] ; quant à Nature Biotechnology, on y trouve un éditorial assez fouillé [9] qui fait preuve d’un certain scepticisme, remarque que les génomes bactériens synthétiques de Craig Venter ne semblent pas avoir suscité beaucoup de recherches nouvelles, et indique que les applications les plus intéressantes du point de vue industriel (correspondant en gros au Pilot Projects de HGP-write) requièrent de grands segments d’ADN – mais ne nécessitent pas, et de loin, un génome de 3 gigabases. L’éditorial se termine en évoquant lui aussi les inquiétudes liées à la perspective du passage de la cellule à l’embryon, quelles que soient les dénégations des promoteurs du projet.
 | Figure 3.
Le récent livre de George Church (avec E. Regis, journaliste scientifique). « Comment la biologie synthétique va réinventer la Nature et nous-mêmes » [ 7]. |
|
Certes, le projet de séquençage du génome humain a lui aussi suscité bien des réserves lors de son lancement [10], et l’on peut constater aujourd’hui qu’il a été réalisé dans les délais annoncés et que ses retombées justifient très largement l’investissement consenti. Mais rien ne dit qu’il en serait de même pour HGP-write : les inconnues techniques sont plus importantes (surtout au niveau de l’obtention de cellules dirigées par l’ADN synthétique), et les retombées moins évidentes, alors qu’il était clair, dès 1990, qu’une étude systématique du génome donnerait un grand coup d’accélérateur à la génétique médicale. Les questions éthiques posées, en cas de succès, par l’éventualité du passage à l’embryon sont autrement plus graves que celles que soulevait la perspective d’une connaissance intime de notre génome. Au total il semble que ce projet soit mal parti, malgré la qualité de ses promoteurs et leur audience médiatique, et qu’il serait imprudent de parier sur son démarrage effectif et encore plus sur sa réussite. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Boeke JD, Church G, Hessel A, et al. The genome project-write . Science. 2016; ; 353 : :126.–127. 2.
Jordan B. Synthétique, vous avez dit « Synthétique » ? Med Sci (Paris). 2016; ; 32 : :651.–653. 5.
Annaluru N, Muller H, Mitchell LA, et al. Total synthesis of a functional designer eukaryotic chromosome . Science. 2014; ; 344 : :55.–58. 6.
Martella A, Pollard SM, Dai J, Cai Y. Mammalian synthetic biology: time for big MACs . ACS Synth Biol. 2016 ; Apr 20 [Epub ahead of print]. 7.
Church G. Regenesis: how synthetic biology will reinvent nature and ourselves . New York: : Basic Books; , 2012. 9.
Rewriting our future (Editorial) . Nat Biotechnol. 2016; ; 34 : :673.. 10.
Jordan B. Feu sur le quartier général : le génome en balance ? Med Sci (Paris). 1990; ; 6 : :906.–908. |