| |
| Med Sci (Paris). 32(10): 849–860. doi: 10.1051/medsci/20163210018.Le profilage ribosomique
Une technique nouvelle génération pour l’étude de la traduction au cours d’une infection virale Juliana Blin1 and Emiliano P. Ricci1,2,3,4,5* 1CIRI, international center for infectiology research, université de Lyon, 46, allée d’Italie, Lyon, France 2Inserm, U1111, Lyon, France 3École normale supérieure de Lyon, 46, allée d’Italie, 69007Lyon, France 4Université Claude Bernard Lyon 1, centre international de recherche en infectiologie, Lyon, France 5CNRS, UMR5308, Lyon, France |
Vignette (Photo © Lionel Tafforeau). Les virus sont des « parasites » intracellulaires obligatoires qui dépendent de la cellule infectée pour se multiplier. Cette dépendance se traduit à différents niveaux au cours d’une infection virale et notamment lors de la synthèse des protéines du virus. En effet, les virus sont dépourvus de ribosomes qui sont essentiels pour la synthèse de protéines à partir des ARN messagers (ARNm). La traduction est donc une étape critique du cycle de réplication virale pendant laquelle la cellule est trompée afin de traduire les ARNm viraux en dépit de certaines différences structurelles qu’ils présentent avec les ARNm cellulaires. En effet, les contraintes évolutives, induites par le volume limité des particules virales, ont conduit les acides nucléiques viraux à être organisés de façon extrêmement compacte afin de contenir un maximum d’informations génétiques dans un espace réduit [1]. L’expression des gènes viraux se fait selon des mécanismes complexes qui ne sont pas tous encore compris. La compréhension de ces mécanismes ainsi que de ceux impliqués dans la subversion de la cellule est particulièrement importante pour la mise en place de thérapies antivirales plus ciblées et efficaces. Le profilage ribosomique, ou ribosome profiling en anglais, est une technique innovante permettant de suivre à la trace la position de l’ensemble des ribosomes présents sur les ARNm à un moment donné. Elle a récemment permis de faire des avancées considérables dans l’étude de la traduction au niveau cellulaire [2], notamment au cours d’une infection virale [3]. Cette méthode s’inspire des techniques d’empreintes, ou footprinting, où l’acide nucléique, soumis à une digestion enzymatique, est protégé localement par les protéines qui lui sont associées. En comparant la séquence de départ à la séquence digérée, il est ainsi possible de retrouver très précisément la position d’un complexe protéique sur n’importe quelle séquence nucléotidique. En couplant cette approche aux méthodes de séquençage à haut-débit de nouvelle génération, on peut désormais étudier en détail la traduction de l’ensemble des ARNm exprimés dans la cellule [4]. Dans cette synthèse, nous discuterons de la manière dont le profilage ribosomique a révolutionné l’étude de la traduction à l’échelle cellulaire et de la façon dont cette technique peut nous permettre de mieux comprendre le cycle de réplication virale. Nous nous intéresserons également aux limitations et aux biais liés à cette méthode qui doivent être pris en compte afin d’obtenir des résultats de qualité. |
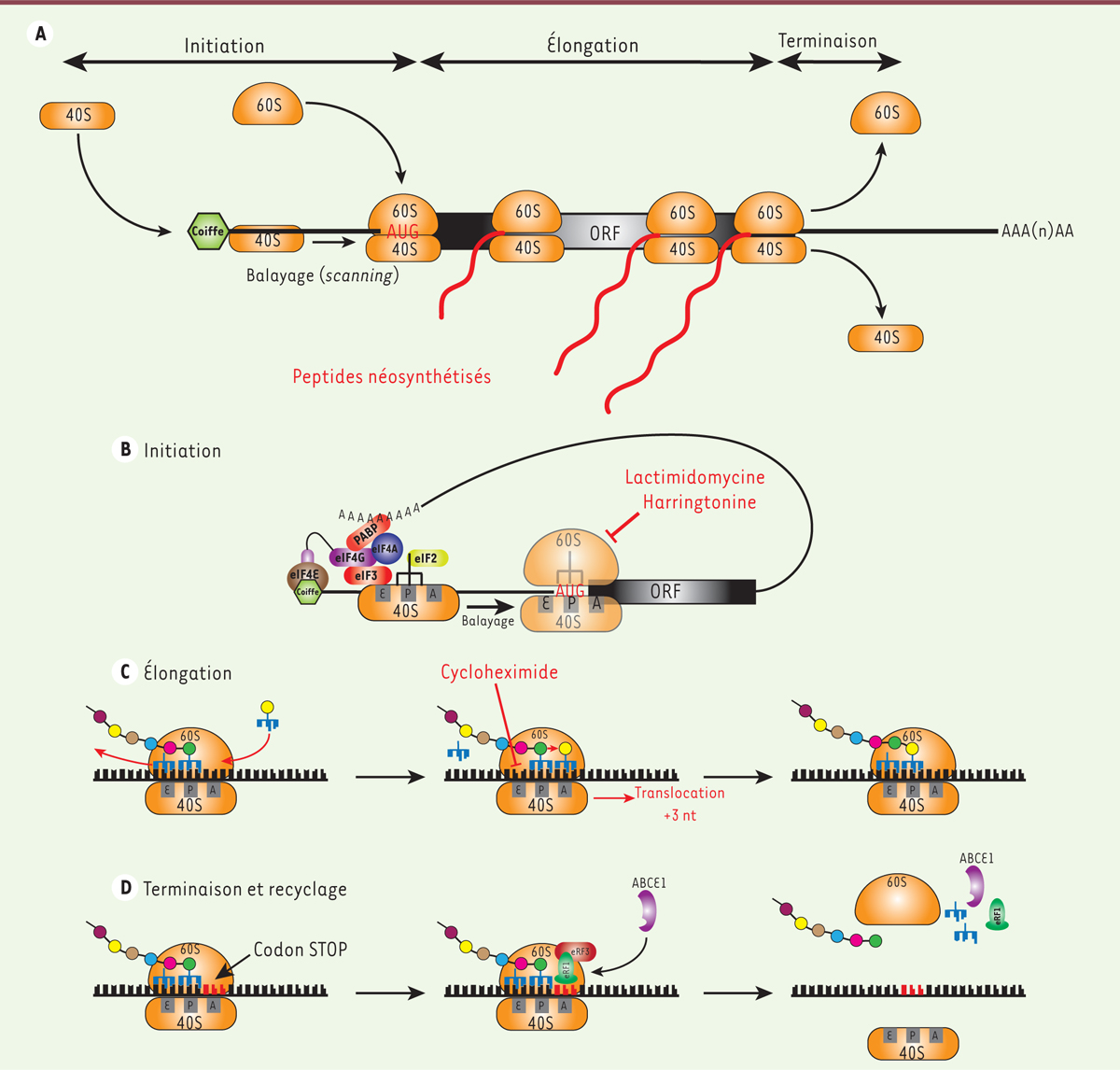
Le profilage ribosomique dans l’étude de la traduction cellulaire La synthèse des protéines est assurée par les ribosomes [43] (→), des complexes macromoléculaires composés à la fois de protéines et d’ARN ribosomaux (ARNr), capables de reconnaître les ARNm et de les traduire. Cette reconnaissance est rendue possible grâce à la présence de structures conservées telles que la coiffe méthylée (m7GpppN-, N7-méthylguanosine-triphosphate) et la queue de poly-adénosines, ou poly(A), sur les ARNm cellulaires. Le processus de traduction se divise en quatre étapes majeures que sont l’initiation, l’élongation, la terminaison et le recyclage des ribosomes et des facteurs de traduction (Figure 1A). La première étape, l’initiation, consiste à recruter un ribosome fonctionnel sur le site d’initiation de la traduction situé le plus souvent au niveau du premier codon AUG de l’extrémité 5’ de l’ARNm [5]. Pour cela, la petite sous-unité du ribosome (40S), associée à un ARN de transfert (ARNt) initiateur, et guidée par différents facteurs d’initiation (regroupés sous le nom d’eIF ou eukaryotic initiation factors), se fixe au niveau de la coiffe pour former un complexe qui va balayer l’ARNm jusqu’à atteindre un codon AUG (Figure 1B). Lorsqu’il l’atteint, le complexe s’arrête et recrute la grosse sous-unité ribosomale (60S) formant ainsi un ribosome (80S) prêt à démarrer la synthèse protéique [6]. L’étape suivante, l’élongation, consiste en l’ajout successif d’acides aminés dans la chaîne polypeptidique en cours de synthèse par le ribosome (Figure 1C). Les acides aminés sont apportés un à un par des ARNt portant chacun un acide aminé correspondant au codon en cours de lecture par le ribosome. L’élongation va ainsi se poursuivre jusqu’à ce que le ribosome atteigne un codon stop (UAG, UGA ou UAA). Lors de l’étape de terminaison, le codon stop est reconnu par le facteur de terminaison eRF1 (eukaryote release factor 1) qui, avec l’aide du facteur eRF3, catalyse l’hydrolyse de la liaison entre le dernier acide aminé incorporé et l’ARNt qui l’apporte [7] (Figure 1D). La protéine nouvellement formée est ainsi libérée du ribosome qui est ensuite dissocié grâce à l’action conjointe des facteurs ABCE1 (ATP binding cassette E1) et eRF1 (Figure 1D). Les différents composants du ribosome sont enfin recyclés pour permettre la traduction de nouveaux ARNm. La plupart des ARNm peuvent être traduits par plusieurs ribosomes en même temps, formant des structures appelées polysomes, ce qui augmente la quantité de protéines pouvant être produite à partir d’un même messager.
(→) Voir la Nouvelle de V. Marcel et al., m/s n° 1, janvier 2014, page 21
 | Figure 1.
Les différentes étapes de la traduction chez les eucaryotes. A. Vision globale des étapes principales de la traduction coiffe-dépendante : initiation, élongation et terminaison. B. Au cours de l’initiation, la petite sous-unité ribosomale (40S), associée au ARNt-initiateur méthionine et à eIF2 (eukaryotic initiation factors2), est recrutée au niveau de la coiffe par une multitude de facteurs d’initiation. Parmi ceux-ci, eIF4E lie directement la coiffe située à l’extrémité 5’ des ARNm cellulaires et interagit avec eIF4G qui joue un rôle d’échafaudage en interagissant avec de multiples autres facteurs comme eIF3 (qui permet de faire le lien avec le ribosome 40S) et PABP (poly(A)-binding protein) qui lie la queue poly(A) située à l’extrémité 3’ de l’ARNm et qui permet de circulariser l’ARNm. Une fois le ribosome recruté sur l’ARNm, il balaie la région 5’ non traduite dans le sens 5’ vers 3’ aidée par l’activité hélicase d’eIF4A qui permet de dérouler les éventuelles structures secondaires de l’ARN qui pourraient bloquer sa progression. Le balayage s’arrête lorsque le ribosome 40S arrive au niveau d’un codon AUG situé dans un bon contexte nucléotidique (aussi appelé contexte de Kozak). Une fois placé sur le codon AUG, les facteurs d’initiation sont relâchés et la grande sous-unité ribosomale (60S) est recrutée pour former un ribosome 80S. C. Lors de l’étape d’élongation, un ARNt chargé entre dans le site A du ribosome. Le ribosome catalyse la liaison peptidique entre l’acide aminé porté par l’ARNt du site A et le peptide situé sur l’ARNt du site P. Une fois la liaison effectuée, le ribosome réalise une translocation de 3 nucléotides en direction 3’ permettant le déplacement de l’ARNt qui était auparavant sur le site P vers le site E et l’arrivée de l’ARNt couplé à la chaîne peptidique dans le site P. Cette translocation libère le site A qui peut donc accueillir un nouvel ARNt chargé. D. Lorsque le ribosome arrive au niveau d’un codon stop, le facteur de terminaison eRF1 (eukaryote release factor 1) associé à eRF3 s’insère dans le site A du ribosome et induit le relargage de la protéine néosynthétisée. La dissociation des deux sous-unités ribosomales est ensuite catalysée par l’action conjointe de ABCE1 (ATP binding cassette E1) et d’eRF1. ORF : open reading frame ; nt : nucléotide. |
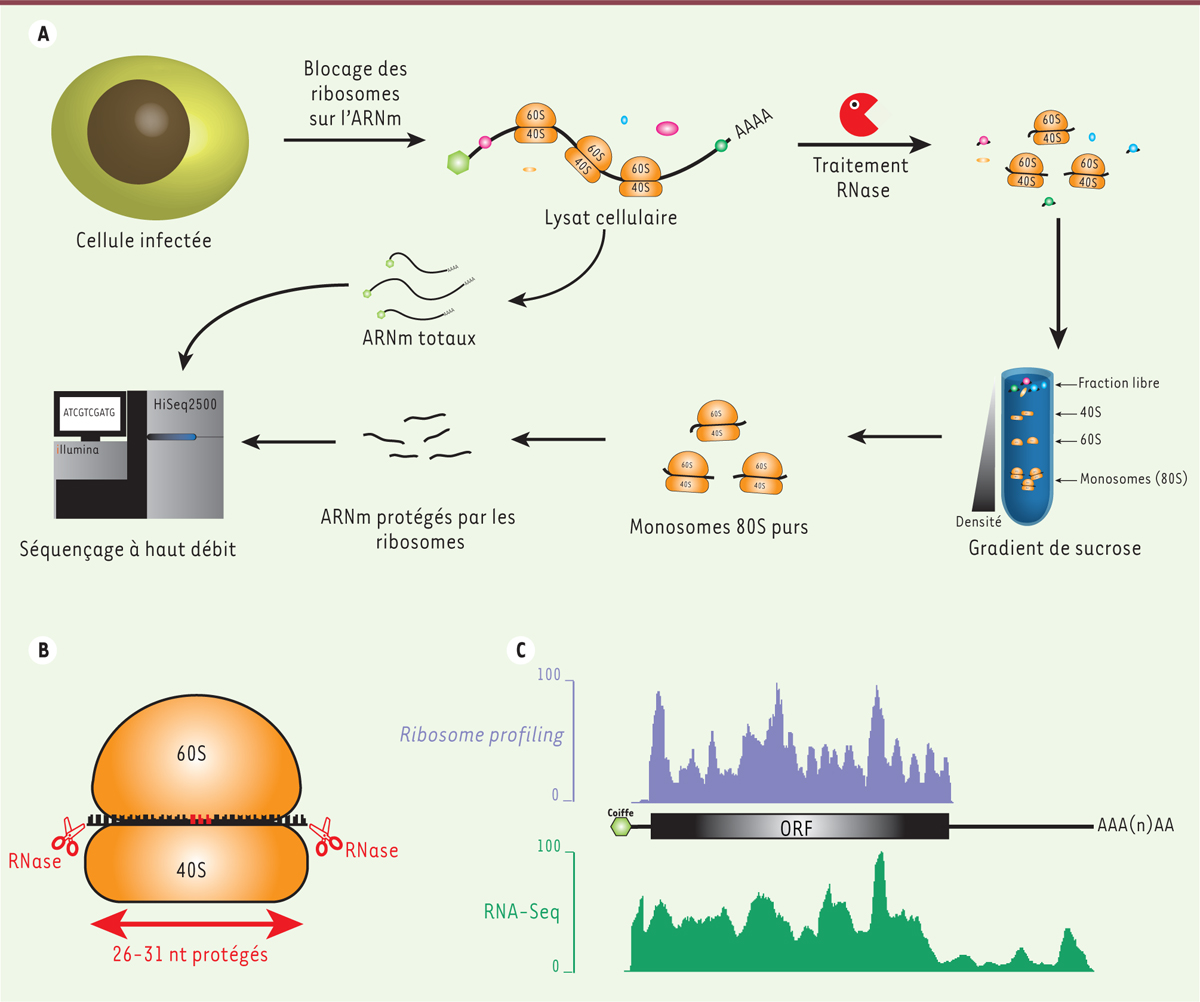
Chez les eucaryotes, différents mécanismes de contrôle de la traduction existent pour réguler la synthèse protéique en fonction des stimulus perçus par la cellule, comme lors de la différenciation cellulaire ou d’un stress [8]. L’expression individuelle d’un gène, ou de toute une classe de gènes, peut ainsi être modulée afin de permettre à la cellule de s’adapter à son environnement, en conditions physiologiques comme pathologiques. Le profilage ribosomique est une technique particulièrement utile pour étudier les mécanismes de contrôle traductionnel mis en place par la cellule pour s’adapter dans différents contextes, y compris au cours d’une infection virale [3]. Cette approche permet, notamment, d’étudier les processus moléculaires mis en jeu pour contrôler la synthèse protéique à un niveau de détail jamais égalé auparavant en s’appuyant sur le séquençage à haut-débit des ARNm en cours de traduction. L’avènement du séquençage massif parallèle, ou next generation sequencing (NGS), a en effet révolutionné de nombreux domaines de la biologie en permettant aux expérimentateurs d’avoir une vision globale des processus cellulaires faisant intervenir des acides nucléiques [9]. Le principe de ce type d’approche est de séquencer un grand nombre de petits fragments nucléotidiques, généralement plusieurs millions de fragments de quelques dizaines à quelques centaines de nucléotides de long. Cette technologie a été rapidement adoptée pour l’étude de l’expression des gènes à différents niveaux. En effet, un des avantages majeurs du NGS, comparé aux méthodes préexistantes, comme les microarrays, est qu’il permet la détection de l’ensemble des transcrits présents dans un échantillon de manière très sensible et sans aucun a priori. Ces dernières années, la grande versatilité des plates-formes de NGS et la baisse continue de leur coût d’utilisation a favorisé le développement de nombreux protocoles dédiés à l’étude des ARN dans différents contextes. Parmi ces nouvelles approches, le profilage ribosomique, qui permet donc de suivre très précisément la traduction de l’ensemble des ARNm cellulaires à un instant donné, consiste à cartographier les sites d’interaction de la sous-unité 80S des ribosomes sur les ARNm en cours de traduction (Figure 2). Cela est rendu possible par le fait que le ribosome protège l’ARNm auquel il est lié sur une zone d’environ 30 nucléotides (Figure 2B) [10]. De ce fait, les régions nues de l’ARNm sont plus sensibles à la dégradation que celles qui sont associées aux ribosomes, notamment lors d’un traitement avec des nucléases. Le séquençage à haut-débit des fragments obtenus après digestion enzymatique de polysomes permet ainsi de déterminer la position exacte de chaque ribosome associé à un ARNm et pour un ARNm donné, la quantité de ribosomes qui sont en train de le traduire [4, 11].
 | Figure 2.
Étude globale de la traduction cellulaire par profilage ribosomique. A. Les cellules ou tissus d’intérêt sont traités avec des inhibiteurs de la traduction comme le cycloheximide dans le but de figer les ribosomes sur les ARNm qu’ils traduisent. Les cellules ou tissus d’intérêt sont ensuite lysés et la fraction cytoplasmique récupérée (compartiment où a lieu la traduction des ARNm). Les lysats obtenus sont ensuite séparés en deux fractions. La première est utilisée pour extraire les ARNm et mesurer leur niveau d’expression par séquençage massif parallèle (RNA-sequencing, RNA-Seq). La deuxième fraction est incubée avec des ribonucléases (généralement avec la RNase I qui clive l’ARN simple brin quelle que soit la séquence nucléotidique) qui vont dégrader toutes les régions d’ARN accessibles à l’exception de celles qui sont associées aux protéines liant l’ARN et celles associées aux ribosomes 80S. Les ribosomes étant plus denses et volumineux que les protéines libres et autres complexes ribonucléoprotéiques, ils peuvent être purifiés sur un gradient de sucrose, sur coussin de sucrose ou par chromatographie d’exclusion. Une fois les ribosomes 80S isolés, les fragments d’ARNm protégés sont récupérés puis utilisés pour préparer des banques d’ADN complémentaire (ADNc) qui seront finalement séquencées. Une fois les résultats du séquençage obtenus, les séquences protégées par le ribosome ainsi que les séquences correspondant au RNA-Seq (ARNm en entier) sont alignées contre le génome de référence correspondant à l’espèce du matériel de départ. Une fois les alignements obtenus, l’expérimentateur peut mesurer la densité de ribosomes par molécule d’ARNm en calculant le ratio entre le nombre de séquences protégées par les ribosomes pour un ARNm donné et le nombre de séquences obtenues pour le même ARNm à partir du RNA-Seq. B. Les fragments protégés font généralement entre 26 et 31 nt (nucléotides) de long (la majorité des fragments font 28 nt de long). En conséquence, il est possible de retrouver la phase de lecture en prenant comme information la position du premier nucléotide de chaque séquence. C. Exemple de résultat obtenu par profilage ribosomique et RNA-Seq sur le gène humain codant la glycéraldéhyde-3-phosphate déhydrogénase (GAPDH). Sur le panel du haut (ribosome profiling), le nombre de fragments protégés par le ribosome est présenté en ordonnée par rapport à chaque position du transcrit codant la GAPDH présentée en abscisse. Sur le panel du bas (RNA-Seq), est présenté le nombre de fragments d’ARNm séquencés pour chaque position du transcrit codant la GAPDH. On peut observer que le signal correspondant au profilage ribosomique est limité à la région codante du transcrit alors que le signal correspondant au RNA-Seq s’étend sur les régions 5’ et 3’ non traduites. OFR : open reading frame. |
D’un point de vue pratique, la première étape du profilage ribosomique consiste à bloquer la progression de la traduction dans la cellule grâce l’ajout d’inhibiteurs de la traduction ou par incubation à froid des échantillons (Figure 2A). Ces inhibiteurs agissent soit en bloquant l’avancée des ribosomes sur les ARNm et en empêchant l’élongation, comme c’est le cas de la cycloheximide (Figure 1C), soit en ciblant l’initiation de la traduction et en figeant les ribosomes sur le codon d’initiation, comme c’est le cas de la lactimidomycine ou de l’harringtonine [12] (Figure 1B). Lorsque l’élongation est bloquée, la mesure de la densité de ribosomes associés à une région codante donne une mesure indirecte de l’efficacité de traduction du transcrit (paramètre qui dépend à la fois du nombre de ribosomes associés à l’ARNm et de la vitesse d’élongation) [13]. De plus, en étudiant l’empreinte obtenue pour chaque ribosome, il est possible de retrouver la phase de lecture de ces ribosomes sur une région codante. En couplant cette approche au séquençage des ARNm, il est ainsi possible de caractériser l’ensemble du transcriptome cellulaire et de cartographier les régions du génome qui sont codantes [14] (Figure 2C). L’utilisation d’inhibiteurs de l’initiation a plutôt pour application la localisation exacte des sites d’initiation de la traduction mais aussi la caractérisation de la vitesse d’élongation des ribosomes [11]. En effet, lors de l’ajout de ces drogues, les ribosomes en phase d’élongation vont poursuivre la traduction jusqu’à atteindre le codon stop selon un phénomène connu sous le nom de ribosome run-off. En bloquant l’élongation à différents temps après l’ajout de l’inhibiteur d’initiation, il est possible de mesurer la distance parcourue par les ribosomes depuis le site d’initiation et ainsi de déterminer la vitesse d’élongation et les sites de pause du ribosome. Depuis le développement de cette technique, en 2009 [4], l’étude de la traduction par profilage ribosomique a permis de mettre en évidence un grand nombre de mécanismes de régulation de la traduction au niveau cellulaire. Ainsi, il a été montré que les cellules faisaient régulièrement appel à des mécanismes de contrôle traductionnel originaux tels que l’utilisation de codons d’initiation non canoniques (différents du codon AUG), de petits cadres de lecture ouverts dans les régions non traduites en 5’ des ARNm, connus sous le nom d’upstream open reading frames ou uORF, ou encore de cadres de lecture alternatifs dans les ARNm [2]. Le profilage ribosomique a aussi permis de caractériser finement les changements globaux de traduction qui se produisent en cas de stress cellulaire, comme au cours d’un choc thermique [15], d’un stress oxydatif [16] ou d’une infection virale [17]. |
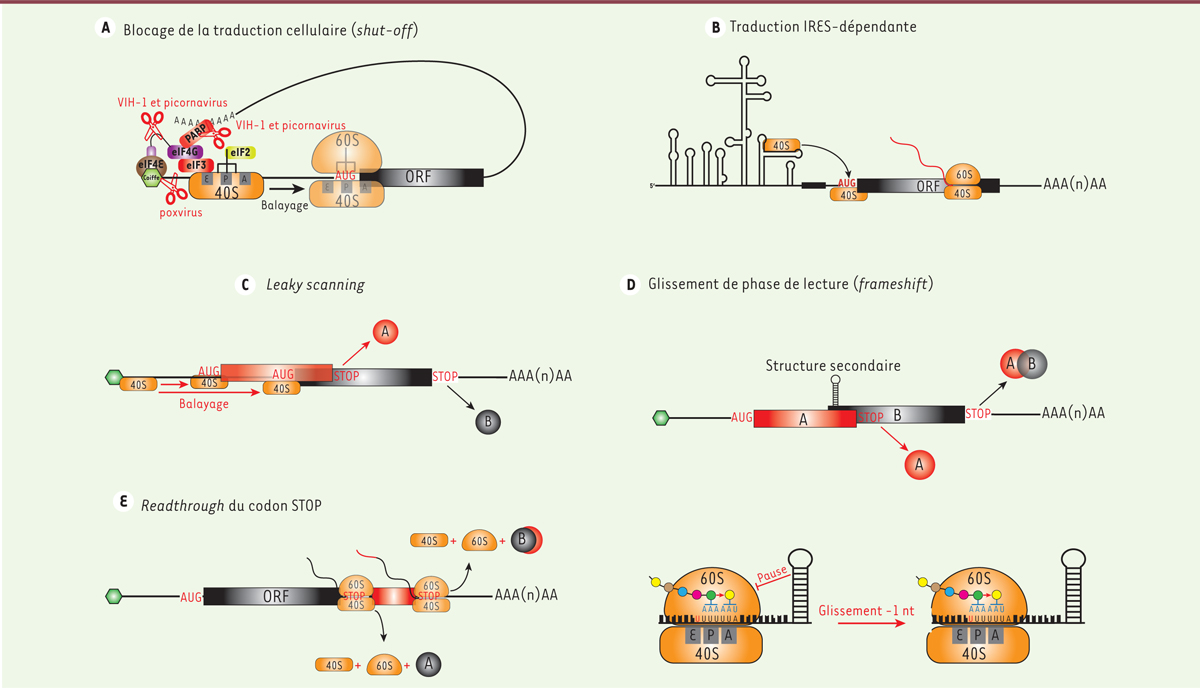
L’étude de la traduction des ARNm viraux par profilage ribosomique La traduction est une étape critique au cours d’une infection virale durant laquelle les ARNm viraux sont en compétition directe avec les ARNm cellulaires pour s’associer aux ribosomes. De nombreux ARNm viraux présentent des caractéristiques qui réduisent leur capacité à être traduits par la machinerie cellulaire, comme l’absence de coiffe ou de queue poly(A), ou encore la présence de structures secondaires essentielles pour la réplication des ARN viraux mais qui peuvent bloquer la progression des ribosomes. Cela est particulièrement vrai pour les virus à ARN. En conséquence, les virus ont évolué de manière à développer des mécanismes alternatifs pour assurer l’expression efficace de leurs protéines. Leur principale stratégie consiste à cibler les différents facteurs de traduction cellulaires. De nombreux mécanismes de subversion interviennent notamment au niveau de l’initiation de la traduction, car c’est à cette étape que la régulation est la plus importante [18]. Ainsi, la capacité à recruter les ribosomes sur les ARN viraux est capitale pour le succès de l’infection [19]. Si de nombreux mécanismes de détournement de la cellule-hôte ont déjà été mis en évidence par le passé, leurs conséquences sur l’expression des ARNm cellulaires restent aujourd’hui encore assez mal comprises. Le profilage ribosomique est une technique particulièrement prometteuse pour l’analyse approfondie des mécanismes de détournement de la cellule au cours d’une infection virale [3, 20]. Cette méthode peut notamment s’appliquer à l’étude des infections virales in vitro, sur des cultures de cellules infectées, mais aussi ex vivo, en travaillant directement à partir de tissus animaux infectés. Dans la suite de cette revue, nous allons détailler plusieurs mécanismes d’expression des gènes viraux et de détournement de la cellule-hôte qui peuvent être étudiés par le profilage ribosomique en décrivant les avantages par rapport aux techniques classiques d’étude de la traduction. Initiation de la traduction coiffe-indépendante par les IRES Les IRES ( internal ribosome entry site) correspondent à des séquences portées par les ARNm capables de recruter la sous-unité 40S du ribosome indépendamment de la présence d’une coiffe à l’extrémité 5’ du transcrit (Figure 3A). En dépit de leur grande variabilité au niveau de leur séquence primaire, la plupart des IRES possèdent des régions fortement structurées permettant une interaction directe avec le ribosome ou certains facteurs d’initiation de la traduction. La variabilité des séquences IRES se traduit aussi par une forte diversité de mécanismes moléculaires utilisés pour l’initiation de la traduction. Ainsi, toutes les IRES ne vont pas recruter les mêmes facteurs d’initiation de la traduction et certaines sont même capables de s’en affranchir totalement. Dans la cellule eucaryote, les séquences IRES sont présentes dans un nombre limité de transcrits et permettent d’assurer le maintien de la production de protéines importantes en conditions de stress. Ces séquences sont essentielles pour la synthèse des protéines virales à partir d’ARNm naturellement dépourvus de coiffe, comme c’est le cas pour de nombreux virus à ARN. Elles sont également retrouvées dans des transcrits viraux portant une coiffe et une queue poly(A). Dans ce cas, elles permettent la production de protéines virales même lorsque la traduction coiffe-dépendante est perturbée, comme par exemple lors du phénomène de host shut-off que nous décrirons dans la suite de cette revue [ 1]. Par ailleurs, les séquences IRES peuvent permettre d’accroître le nombre de protéines différentes codées par un même messager, notamment grâce à la traduction de l’ARNm viral à la fois par la voie canonique et via une IRES. Cette stratégie est retrouvée par exemple chez les virus de l’immunodéficience humaine (VIH-1 et VIH-2) qui sont capables d’exprimer plusieurs isoformes de la protéine structurale Gag en utilisant à la fois les mécanismes de traduction coiffe-dépendant et IRES-dépendant [ 21].
 | Figure 3.
Divers mécanismes de régulation de la traduction chez les eucaryotes. A. Les séquences IRES (internal ribosome entry site) permettent de recruter la petite sous-unité du ribosome (40S) indépendamment de la coiffe et de certains facteurs d’initiation de la traduction et sont insensibles aux mécanismes de blocage de la traduction mentionnés ci-dessus. B. Blocage de la traduction coiffe-dépendante cellulaire par les virus. Certains virus, comme les picornavirus, les VIH (virus de l’immunodéficience humaine) ou les poxvirus, sont capables d’induire la protéolyse de certains facteurs d’initiation de la traduction essentiels à la traduction coiffe-dépendante ou d’induire le clivage de la coiffe à l’extrémité 5’ des ARNm et d’inhiber la traduction de la plupart des transcrits cellulaires. C. Lors du leaky scanning, certains ribosomes 40S ne reconnaissent pas le premier codon AUG situé en 5’ de l’ARNm et continuent de balayer la 5’UTR (untranslated region) pour initier la traduction au niveau d’un codon AUG situé en aval. De cette façon, plusieurs protéines ou isoformes protéiques peuvent être synthétisées à partir du même ARNm. D. Le glissement de phase de lecture se produit généralement lorsque le ribosome 80S est ralenti par une structure secondaire d’ARN, se décale d’un ou deux nucléotides (nt) puis reprend l’élongation dans une autre phase de lecture. E. Lors du readthrough, les ribosomes 80S échouent à reconnaître le codon stop en incorporant un acide aminé à la place et continuent l’élongation en aval permettant la synthèse d’une isoforme de la protéine avec une extension C-terminale. ORF : open reading frame ; elF : eukaryotic initiation factors ; PABP : poly(A)-binding protein. |
Historiquement, l’étude de la traduction IRES-dépendante chez les virus a été réalisée en utilisant des gènes rapporteurs monocistroniques (portant la séquence IRES d’intérêt au niveau de la région 5’UTR [5’ untranslated region]), ou bicistroniques (portant la séquence IRES entre deux gènes rapporteurs). Ces constructions étaient ensuite utilisées dans des systèmes de traduction in vitro, ou transfectées dans des cellules en culture. Ces méthodes ont permis de caractériser l’activité IRES dans de nombreux virus en rendant possible l’étude détaillée de leurs besoins en facteurs d’initiation de la traduction et la localisation précise du site d’initiation. Cependant, lorsque l’on utilise ce type d’approche, de nombreux contrôles sont nécessaires afin de valider l’activité IRES d’une séquence donnée et d’exclure les artéfacts dus à la présence de promoteurs ou de sites d’épissage cryptiques dans la région supposée contenir une IRES. Le profilage ribosomique pourrait faciliter la détection et l’étude des séquences IRES directement au cours de l’infection virale et donc dans un contexte plus physiologique. Notamment, dans le cas de virus induisant un blocage de la traduction coiffe-dépendante, le profilage ribosomique permettrait d’identifier les ARNm cellulaires et viraux dont la traduction est résistante à ce blocage, potentiellement grâce à une activité IRES. En couplant cette approche avec des techniques complémentaires permettant une analyse poussée de l’expression des transcrits telles que le séquençage ARN ou le CAGE-Seq (cap analysis gene expression, permettant la cartographie de l’extrémité 5’ des transcrits), il est même possible d’aller encore plus loin dans la caractérisation des séquences contenant des IRES en excluant celles qui correspondent à des artéfacts liés à la présence de promoteurs ou de sites d’épissage cryptiques. Host shut-off ou modulation de la traduction des ARNm de la cellule-hôte Comme décrit précédemment, la grande majorité des ARNm cellulaires sont traduits selon le mécanisme conventionnel coiffe-dépendant (Figure 1B). En ciblant ce mode de traduction, le virus peut donc perturber la production globale des protéines cellulaires. Cette stratégie, nommée host shut-off, permet aux virus qui sont capables d’utiliser une voie alternative d’initiation de la traduction, comme les IRES, de se débarrasser de toute compétition pour le recrutement des ribosomes sur leurs ARNm. Certains virus vont par exemple cibler les zones conservées des ARNm cellulaires pour empêcher leur traduction. C’est le cas des poxvirus qui expriment des enzymes de dégradation de la coiffe permettant de réduire le pool d’ARNm traduits selon la voie coiffe-dépendante (Figure 3B) [ 22]. D’autres virus, comme les picornavirus et les rétrovirus, vont quant à eux cibler des facteurs essentiels pour l’initiation de la traduction coiffe-dépendante, tels que eIF4G et PABP ( poly(A)-binding protein), et induire leur dégradation (Figure 3B) [ 18]. Ce phénomène d’extinction de la synthèse protéique cellulaire joue aussi un rôle dans l’échappement face aux défenses immunitaires. En effet, en réduisant le taux de synthèse protéique global, l’expression de protéines nécessaires à la mise en place d’une réponse immunitaire antivirale efficace est également limitée [ 20]. Le profilage ribosomique est particulièrement utile pour étudier ce phénomène, car il permet de quantifier simultanément et précisément le taux de traduction des ARNm cellulaires et viraux au cours d’une infection. Il est ainsi possible de caractériser les mécanismes moléculaires mis en place pour induire un host shut-off et de mesurer leur impact sur la traduction cellulaire. En couplant cette approche avec la mesure des niveaux d’expression des transcrits cellulaires et viraux en temps réel par séquençage à haut débit, il a été récemment montré que le virus HSV-1 ( herpes simplex virus 1) était capable de perturber spécifiquement l’étape de maturation des ARNm cellulaires pour conduire au shut-off des protéines cellulaires et favoriser l’expression de certains de ses ARNm dont la maturation n’est pas sensible au blocage qu’il induit [ 23]. Une étude de profilage ribosomique récemment réalisée chez un coronavirus murin a, au contraire, montré un mécanisme alternatif pour augmenter la production de protéines virales lors du cycle de réplication [ 24]. Dans ce cas, la transcription des ARN viraux est tellement importante qu’ils représentent jusqu’à 90 % de l’ensemble des ARN codants dans la cellule hôte. Cette stratégie permet ainsi au virus de produire de grandes quantités de protéines virales sans que l’efficacité de traduction de ses ARNm ne soit plus importante que celle des ARNm cellulaires. Leaky scanning et initiation de la traduction à partir de codons non-AUG Un autre mécanisme de régulation de l’initiation de la traduction qui peut être étudié par profilage ribosomique est le leaky scanning (Figure 3C). Dans la voie conventionnelle, le complexe d’initiation balaye l’extrémité 5’ de l’ARNm et s’arrête au niveau du premier codon d’initiation AUG qu’il rencontre avant de former un ribosome 80S et de débuter l’élongation (Figure 1B) [ 6]. Dans le cadre du leaky scanning, une partie des sous-unités 40S ne s’arrête pas au niveau du premier codon d’initiation de la traduction mais continue de balayer l’ARNm jusqu’à atteindre le codon AUG suivant (Figure 3C). Le leaky scanning est utilisé régulièrement par les cellules eucaryotes comme mécanisme de contrôle traductionnel notamment pour réguler l’expression des protéines de réponse au stress. Ce mécanisme est aussi fréquemment utilisé par les virus, et particulièrement par les virus à ARN, afin d’assurer la traduction d’ARNm viraux polycistroniques, qui codent pour plusieurs protéines à la fois [ 1]. Ainsi, la présence de codons AUG dans un cadre de lecture différent de celui du codon initiateur canonique, qui peuvent être reconnus par leaky scanning, permet la production simultanée de différentes protéines à partir d’un même ARNm (Figure 3C). Un exemple connu est le cas des papillomavirus humains (HPV) qui utilisent le leaky scanning pour assurer la production des oncoprotéines virales E6 et E7 à partir d’un même messager [ 25]. Grâce à l’utilisation d’inhibiteurs de l’initiation de la traduction comme l’harringtonine, il est possible de figer les ribosomes au niveau des codons d’initiation auxquels ils sont associés et donc de déterminer leur position sur chaque ARNm. Ainsi, les transcrits ayant recours au leaky scanning peuvent être facilement détectés grâce à la présence d’empreintes de la sous-unité 80S multiples au niveau de chaque codon d’initiation utilisé [ 11]. Cette approche a récemment permis de mettre en évidence l’existence de nombreux sites d’initiation de la traduction non canoniques chez les ARNm cellulaires [ 26]. En effet, en raison de la dégénérescence du code génétique, l’ARNt initiateur qui s’associe préférentiellement au codon AUG peut également initier la traduction à partir de codons de séquence proche comme le CUG ou le UUG. Chez certains virus, l’initiation peut également se faire sur des codons non-AUG dans le cadre du leaky scanning. C’est le cas notamment pour le virus leucémogène murin (MuLV) qui utilise un codon CUG situé en amont du codon canonique pour produire une isoforme de sa polyprotéine Gag qui n’est pas incorporée dans les particules virales, mais qui joue un rôle important dans la dissémination des virions [ 27, 28]. Le profilage ribosomique appliqué à l’étude de la traduction au cours d’une infection virale a également permis récemment d’identifier de nombreux sites d’initiation non-canoniques chez deux virus à ADN et un virus à ARN [ 17, 24, 29]. La plupart des sites d’initiation non-AUG découverts à ce jour chez les virus sont utilisés pour la synthèse de petits peptides dont le rôle biologique n’est pas encore connu. Des études supplémentaires seront donc nécessaires afin de vérifier si ces peptides participent activement au cycle de réplication, s’ils jouent plutôt un rôle régulateur sur la traduction d’autres gènes viraux en cis ou encore s’ils correspondent à un bruit de fond. Frameshifting ou glissement de phase de lecture Le frameshifting (glissement de phase de lecture) est un mécanisme conservé chez les eucaryotes comme chez les virus qui permet l’expression de plusieurs protéines à partir d’un même ARNm (Figure 3D). De manière générale au cours de l’élongation, le ribosome se déplace le long de l’ARNm par translocation de codon en codon, soit de 3 nucléotides à chaque fois. Dans le cas d’un glissement de phase de lecture, le ribosome peut se décaler d’un ou de deux nucléotides supplémentaires ce qui induit un décalage de la phase de lecture (Figure 3D). Ce mécanisme permet notamment l’expression de protéines différentes en fonction du contexte cellulaire. Chez les virus, le frameshifting est très répandu, car il augmente sensiblement la quantité d’information génétique que peut contenir un acide nucléique [ 1]. Chez certains rétrovirus, en particulier les VIH, ce processus est essentiel pour la production des protéines virales. Il représente donc une cible thérapeutique de choix [ 30]. L’efficacité du glissement de phase de lecture étant relativement faible, il permet en outre au virus de restreindre les niveaux d’expression de certaines de ses protéines qui pourraient être toxiques pour la cellule à forte dose [ 31]. Un exemple particulièrement étudié est l’utilisation du frameshifting par le VIH-1 afin d’exprimer de manière alternée les précurseurs protéiques Gag et la protéine de fusion Gag/Pol, toutes deux essentielles pour la production de particules virales infectieuses [ 32]. La mise en évidence d’un décalage de phase de lecture peut être réalisée par des approches de prédiction in silico ou en utilisant des méthodes plus classiques de génétique. Le profilage ribosomique est particulièrement intéressant pour l’étude de ce mécanisme car il permet à la fois de détecter un décalage dans la phase de lecture pour un transcrit donné, et de quantifier le taux de traduction au niveau de chaque cadre de lecture en conditions physiologiques. En effet, en suivant la position des empreintes ribosomales laissées sur son extrémité 5’ après la digestion par les nucléases, il est possible de déterminer la phase de lecture d’un ARNm en cours de traduction [ 16]. En cas de frameshifting, la position des empreintes obtenues ne suivra pas la périodicité de trois nucléotides au niveau de la région où les deux cadres de lecture se chevauchent. Elle sera décalée de un ou deux nucléotides si le deuxième cadre de lecture s’étend en aval du cadre de lecture canonique. Récemment, cette méthode a permis d’étudier le mécanisme de frameshift utilisé par un coronavirus murin appartenant au même genre que le virus SARS-CoV à l’origine du SRAS (syndrome respiratoire aigu sévère) [ 24]. Les résultats obtenus ont permis de montrer que les ribosomes ne font pas de pause au niveau de la structure en pseudo-nœud responsable du glissement de phase dans les cellules infectées, contrairement à ce qui avait été démontré précédemment in vitro [ 33]. Ces résultats posent ainsi de nouvelles questions quant au rôle des structures secondaires de l’ARNm dans le mécanisme moléculaire impliqué dans le glissement de phase de lecture. Il sera intéressant d’étendre les résultats obtenus chez le coronavirus à d’autres virus ayant recours au frameshift comme le VIH-1 et de voir si l’absence de pause des ribosomes au cours de l’élongation observée in vivo est une caractéristique générale remettant en cause les résultats obtenus in vitro. Réinitiation de la traduction et translecture du codon stop La modulation de la traduction cellulaire peut également avoir lieu au cours des étapes tardives de la synthèse protéique comme lors de la terminaison. Notamment, dans certains cas où le codon stop est situé à proximité de séquences particulières, qui peuvent contenir ou non des structures secondaires, le ribosome peut l’ignorer et poursuivre l’élongation de la chaîne polypeptidique (Figure 3E). Ce phénomène est appelé translecture ou readthrough du codon stop et permet de synthétiser une isoforme d’une protéine avec une extension C-terminale (Figure 3E). À l’inverse, dans le cas de la réinitiation de la traduction, le ribosome reconnaît correctement le codon stop et l’étape de terminaison se fait de manière conventionnelle. Cependant, la petite sous-unité du ribosome (40S) reste accrochée à l’ARNm après l’arrêt de la synthèse protéique et reprend le balayage de l’ARNm en aval du codon stop jusqu’à atteindre un autre codon d’initiation et traduire un nouveau cadre de lecture. Ces deux mécanismes de contrôle traductionnel peuvent être détournés au cours d’une infection virale pour assurer la production de plusieurs protéines différentes à partir d’un même ARNm [ 1]. Ainsi, de nombreux génomes viraux contiennent des séquences favorisant la translecture et la réinitiation de la traduction afin d’augmenter la quantité d’information génétique que peut contenir un même ARNm viral. Le profilage ribosomique a déjà été utilisé pour étudier la translecture du codon stop chez l’homme et la drosophile [34, 35]. Les résultats obtenus ont montré que de nombreux gènes utilisent ce mécanisme de régulation pour produire des isoformes de protéines possédant des caractéristiques différentes comme, par exemple, pour leur localisation intracellulaire. Son application sur des cellules infectées pourrait permettre d’étudier l’importance de ce type de phénomène dans le cycle de réplication virale grâce à la localisation précise de l’ensemble des ribosomes en cours de traduction à un moment donné. |
Découverte de nouvelles protéines virales par profilage ribosomique Une des applications les plus importantes du profilage ribosomique dans l’étude du cycle de réplication virale consiste en la découverte de nouvelles régions codantes et la confirmation de l’utilisation de cadres de lecture ouverts (open reading frame ou ORF) prédits à partir de l’analyse bio-informatique de la séquence primaire des génomes viraux [14]. Les ORF correspondent à des portions des ARNm qui peuvent potentiellement être traduites en protéines et sont définies comme une région comprise entre deux codons stop séparés par une série de triplets. Le profilage ribosomique est particulièrement utile pour l’étude des virus complexes qui possèdent un grand génome codant pour de nombreux transcrits dont le rôle n’est pas encore caractérisé : il permet de mettre en évidence les séquences virales qui sont effectivement traduites au cours d’une infection. Les résultats obtenus avec cette méthode doivent cependant être validés par des approches complémentaires afin de suivre, en parallèle, la quantité de protéines virales produites dans la cellule telles que la spectrométrie de masse ou le western blot. Récemment, une telle approche a été appliquée à l’analyse de la synthèse des protéines lors de l’infection par le cytomégalovirus humain (HCMV). D’après des prédictions obtenues après analyses bio-informatiques, ce virus possède un génome complexe d’environ 240 kilobases contenant plus de 200 ORF d’au moins 50 acides aminés de long [36]. Pour décrypter le processus de production des protéines de ce virus, l’ensemble des transcrits produits au cours de l’infection a été séquencé et leur traduction étudiée par profilage ribosomique [17]. De plus, la position de l’extrémité 5’ de tous les transcrits viraux a été caractérisée et l’expression des protéines virales et cellulaires analysé par spectrométrie masse. Les résultats obtenus ont conduit à la caractérisation de plus de 700 régions codantes dont plus de la moitié codent des petits peptides de moins de 80 acides aminés. Les auteurs ont par ailleurs trouvé que de nombreuses régions codantes se chevauchaient soit dans la même phase de lecture, produisant des isoformes tronquées de la même protéine, soit dans des phases de lectures différentes et donc produisant des protéines différentes. Ils ont également découvert l’existence d’ARN viraux antisens (codés à partir des brins opposés du génome) dont les régions codantes se chevauchent au niveau génomique. Ce résultat est particulièrement intéressant en regard des contraintes évolutives nécessaires pour coder deux protéines fonctionnelles à partir des deux brins d’une même séquence nucléotidique. Enfin, les auteurs ont montré que de nombreux ARN auparavant prédits comme non codants par des approches bio-informatiques car ne possédant pas d’ORF suffisamment longs, sont en réalité traduits et correspondent à des ARN polycistroniques codant plusieurs peptides de moins de 90 acides aminés chacun. Même si la plupart des ORF identifiés correspondent effectivement à des protéines virales, leur rôle au cours du cycle de réplication virale reste cependant encore à être caractérisé. Il est possible que ces régions codantes correspondent à du bruit de fond traductionnel ou qu’elles participent à la régulation de la traduction ou de la stabilité de transcrits en cis sans que la protéine produite ne joue un rôle biologique (comme c’est déjà le cas de certains uORF [upstream open reading frame]). Ces résultats démontrent à quel point les génomes viraux peuvent être complexes et font appel à des mécanismes d’expression particulièrement originaux. Le développement d’approches innovantes telles que le profilage ribosomique ouvre donc de nouvelles perspectives pour l’étude de la traduction chez de tels virus. |
Limitations et biais liés au profilage ribosomique Le profilage ribosomique représente donc une avancée technique majeure pour l’étude de la traduction des ARNm. Cependant, cette approche présente un certain nombre de biais et de limitations qu’il ne faut pas négliger lors de l’analyse des résultats. Une des limitations majeures réside dans le fait que le profilage ribosomique ne permet pas d’obtenir une mesure directe de l’efficacité de traduction d’un ARNm mais uniquement de mesurer la densité de ribosomes sur un transcrit donné. En effet, dans le cas où les ribosomes seraient bloqués ou ralentis en phase d’élongation, leur densité sur la région codante de l’ARNm traduit pourrait croître sans pour autant conduire à une augmentation du nombre de protéines produites par transcrit. De plus, la présence de ribosomes sur un ARNm n’est pas nécessairement synonyme de synthèse protéique active. Il est donc préférable de valider les résultats obtenus, au moins sur une partie des transcrits d’intérêt, en utilisant des méthodes alternatives permettant de suivre les modifications de l’expression protéique telles que l’utilisation de gènes rapporteurs, le western blot ou encore la spectrométrie de masse. Par ailleurs, de nouveaux outils analytiques, comme le « ribosome release score » ou RSS, ont été développés pour discriminer, parmi les ARNm associés à des ribosomes, ceux qui codent des protéines de ceux qui sont non-codants [37]. Le RSS se base sur l’hypothèse que sur les transcrits réellement codants, le nombre de ribosomes associés en aval du codon stop décroît drastiquement, tandis que pour les ARN non-codants, l’association avec les ribosomes reste homogène le long de toute la séquence. Le profilage ribosomique est également sensible à la présence de contaminants qui peuvent créer des faux-positifs. En effet, malgré la purification des ribosomes par ultracentrifugation sur gradient ou coussin de sucrose avant de récupérer les fragments d’ARNm qui leurs sont associés, des complexes ribonucléiques de haute masse moléculaire peuvent cosédimenter et introduire des fragments d’ARN contaminants dans les fractions récoltées. Afin d’éviter ce type de contamination, il est possible de purifier les ribosomes par chromatographie d’affinité en utilisant des anticorps dirigés contre une protéine de la sous-unité ribosomale 60S [14]. Il est aussi possible d’introduire des étiquettes moléculaires, telles que les séquences tag (étiquettes) HA (hemagglutinin), FLAG (peptide de séquence AspTyrLysAspAspAspAspLys) ou GFP (green fluorescent protein), dans une protéine de la sous-unité 60S [38]. Cette approche est très utile in vivo puisqu’elle permet d’étudier la traduction spécifiquement dans un tissu donné en restreignant l’expression de la protéine ribosomale portant l’étiquette à ce tissu. De plus, des approches de bio-informatique comme le filtrage des séquences en fonction de la taille attendue pour les fragments protégés par le ribosome, connu sous le nom de « fragment length organization similarity score » ou FLOSS, améliorent significativement la qualité des données et minimisent la présence de contaminants [14]. Enfin, il est possible de préparer des échantillons contrôles en utilisant des inhibiteurs des étapes précoces de l’initiation de la traduction comme la patéamine A [17] ou des inhibiteurs non spécifiques de la traduction comme l’EDTA (acide éthylène diamine tétra-acétique). Ces drogues permettent de bloquer complètement l’association des ribosomes aux ARNm afin de vérifier si le signal observé dans les échantillons d’intérêt provient réellement d’empreintes ribosomales ou correspond à des artéfacts. Une limitation supplémentaire est introduite par les inhibiteurs de la traduction généralement utilisés pour figer les ribosomes sur les ARNm qui peuvent modifier leur distribution sur les régions codantes en introduisant un biais non négligeable [39]. Ainsi, l’utilisation de la cycloheximide aurait tendance à enrichir les empreintes de ribosomes situées à proximité du site d’initiation de la traduction ce qui ne serait pas forcément représentatif de la situation réelle dans la cellule d’intérêt avant l’ajout de la drogue. Pour éviter ce problème, des variantes du protocole n’utilisant pas d’inhibiteurs de la traduction ou au contraire utilisant un large excès d’inhibiteurs ont été développées afin de minimiser le biais de position [40]. Une étude a récemment montré que le choix de la nucléase utilisée pour obtenir les empreintes ribosomales pouvait introduire des biais significatifs dans la distribution des séquences protégées [35]. Ainsi, la RNase I a tendance à enrichir les fragments protégés au niveau du site d’initiation de la traduction, alors la nucléase micrococcale révèle la présence de sites protégés dans la région 3’UTR des transcrits. Ces résultats peuvent être expliqués par le fait que la RNAse I induit une dégradation non spécifique des ribosomes, particulièrement dans les échantillons provenant de mammifères [41]. La plupart des nucléases utilisées dans les protocoles de profilage ribosomique, telles que la Rnase I, la nucléase micrococcale et la RNase T1, agissent préférentiellement sur des substrats ARN simple brin. Certaines de ces nucléases (comme la Rnase A, la Rnase T1 et la nucléase micrococcale) ont une préférence pour digérer l’ARN en 3’ ou en 5’ de certains nucléotides : par exemple, la RNase A ne clive l’ARN qu’en 3’ des nucléotides C et U. Il est donc très probable que le choix de la nucléase impose des biais significatifs dans le profil des séquences protégées par les ribosomes. Ces biais, associés à d’autres comme la présence de structures secondaires sur l’ARNm ou les biais dus à la préparation des banques d’ADNc (ADN complémentaire) conduisent à une distribution hétérogène des séquences obtenues par profilage ribosomique le long des régions codantes (Figure 2C). Cette hétérogénéité de distribution rend difficile certaines analyses comme la détection de sites de pause des ribosomes lors de l’élongation [24]. Un paramètre supplémentaire à prendre en compte lors de l’analyse des résultats de profilage ribosomique est la profondeur de séquençage. En effet, l’analyse fine de la phase de lecture des ribosomes sur une région codante nécessite l’alignement d’un grand nombre de séquences sur le transcrit d’intérêt. Cependant, les échantillons obtenus après la purification des ribosomes 80S sont extrêmement riches en ARN ribosomiques qui, même après déplétion, peuvent représenter une fraction importante des séquences obtenues par séquençage. Cela introduit un biais non négligeable, notamment dans le cas où les ARNm viraux étudiés sont faiblement exprimés ou traduits. En effet, contrairement aux autres techniques d’analyse globale comme les microarrays où la mesure de chaque transcrit est indépendante de celle des autres, lors du séquençage à haut débit tous les transcrits sont en compétition pour être séquencés. Ainsi, un transcrit abondant va être surreprésenté en termes de fragments séquencés comparé à un transcrit peu abondant avec une couverture de séquençage beaucoup plus faible. Il faut donc adapter le nombre de fragments à séquencer au niveau d’expression des transcrits d’intérêt et aux questions biologiques posées. Notamment, l’étude de la phase de lecture nécessite une profondeur de séquençage plus importante qu’une analyse d’expression différentielle. Les protocoles de séquençage actuels étant très performants, il est possible d’obtenir des résultats de qualité en préparant au minimum trois réplicats biologiques pour chaque condition testée. En dépit de toutes ces limitations, le profilage ribosomique est un nouvel outil qui ouvre de nombreuses perspectives pour l’étude de la traduction en conditions physiologiques comme pathologiques. |
Le profilage ribosomique est une technique innovante dont l’intérêt majeur repose sur la possibilité de cartographier la position des ribosomes sur l’ensemble des ARNm présents dans une cellule et de quantifier précisément leur densité sur chaque transcrit [4]. Cette approche est particulièrement utile pour suivre les mécanismes permettant la synthèse des différentes protéines virales mis en place lors d’une infection [3, 20]. Il est aussi possible d’étudier les mécanismes de traduction des ARNm viraux, déjà décrits par le passé mais dont les détails moléculaires restaient mal définis, en suivant la localisation des ribosomes. Le profilage ribosomique ouvre également de nouvelles perspectives pour l’étude du cycle de réplication de virus particulièrement complexes, tels que les poxvirus [29] ou encore le cytomégalovirus humain (HCMV) [17], grâce à la découverte de nouvelles protéines virales et potentiellement de mécanismes de traduction des ARNm viraux encore jamais décrits. Cette approche est d’autre part très prometteuse pour l’étude des interactions entre le virus et la cellule-hôte en permettant de suivre l’impact d’une infection virale sur la synthèse protéique cellulaire [42]. Cela est particulièrement intéressant dans le cadre des phénomènes d’échappement immunitaire afin de comprendre comment le virus parvient à empêcher la mise en place d’une réponse immunitaire antivirale efficace. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Nous tenons à remercier tous les membres du laboratoire de traduction eucaryote et virale pour la relecture du manuscrit. Nous remercions la fondation FINOVI, l’ANRS (France REcherche Nord & sud Sida-hiv Hépatites), la Ligue nationale contre le cancer et l’Inserm pour leur soutien financier.
|
1.
Firth AEBrierley I. Non-canonical translation in RNA viruses . J Gen Virol. 2012; ; 93 : :1385.–1409. 2.
Brar GAWeissman JS. Ribosome profiling reveals the what, when, where and how of protein synthesis . Nat Rev Mol Cell Biol. 2015; ; 16 : :651.–664. 3.
Stern-Ginossar N. Decoding viral infection by ribosome profiling . J Virol. 2015; ; 89 : :6164.–6166. 4.
Ingolia NTGhaemmaghami SNewman JRS, et al. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling . Science. 2009; ; 324 : :218.–223. 5.
Sonenberg NHinnebusch AG. Regulation of translation initiation in Eukaryotes: Mechanisms and biological targets . Cell. 2009; ; 136 : :731.–745. 6.
Kozak M. Pushing the limits of the scanning mechanism for initiation of translation . Gene. 2002; ; 299 : :1.–34. 7.
Jackson RJHellen CUTPestova TV. Termination and post-termination events in eukaryotic translation . Adv Protein Chem Struct Biol. 2012; ; 86 : :45.–93. 8.
Gebauer FHentze MW. Molecular mechanisms of translational control . Nat Rev Mol Cell Biol. 2004; ; 5 : :827.–835. 9.
Morris DR. Ribosomal footprints on a transcriptome landscape . Genome Biol. 2009; ; 10 : :215.. 10.
Wolin SLWalter P. Ribosome pausing and stacking during translation of a eukaryotic mRNA . EMBO J. 1988; ; 7 : :3559.–3569. 11.
Ingolia NTBrar GARouskin S, et al. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments . Nat Protoc. 2012; ; 7 : :1534.–1550. 12.
Michel AMBaranov PV. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale . Wiley Interdiscip Rev RNA. 2013; ; 4 : :473.–490. 13.
Ingolia NT, Brar GA, Rouskin S, et al. Genome-wide annotation and quantitation of translation by ribosome profiling . Curr Protoc Mol Biol. 2013 ; chapter 4 : Unit-4.18. 14.
Ingolia NTBrar GAStern-Ginossar N, et al. Ribosome profiling reveals pervasive translation outside of annotated protein-coding genes . Cell Rep. 2014; ; 8 : :1365.–1379. 15.
Shalgi RHurt JAKrykbaeva I, et al. Widespread regulation of translation by elongation pausing in heat shock . Mol Cell. 2013; ; 49 : :439.–452. 16.
Gerashchenko MVLobanov AVGladyshev VN. Genome-wide ribosome profiling reveals complex translational regulation in response to oxidative stress . Proc Natl Acad Sci USA. 2012; ; 109 : :17394.–17399. 17.
Stern-Ginossar NWeisburd BMichalski A, et al. Decoding human cytomegalovirus . Science. 2012; ; 338 : :1088.–1093. 18.
Walsh DMohr I. Viral subversion of the host protein synthesis machinery . Nat Rev Microbiol. 2011; ; 9 : :860.–875. 19.
Roberts LOJopling CLJackson RJ, et al. Viral strategies to subvert the mammalian translation machinery . Prog Mol Biol Transl Sci. 2009; ; 90 : :313.–367. 20.
Walsh DMathews MBMohr I. Tinkering with translation: protein synthesis in virus-infected cells . Cold Spring Harb Perspect Biol. 2013; ; 5 : :a012351.. 21.
Ricci EPRifo RSHerbreteau CH, et al. Lentiviral RNAs can use different mechanisms for translation initiation . Biochem Soc Trans. 2008; ; 36 : :690.–693. 22.
Lloyd RE. Translational control by viral proteinases . Virus Res. 2006; ; 119 : :76.–88. 23.
Rutkowski AJErhard FL’Hernault A, et al. Widespread disruption of host transcription termination in HSV-1 infection . Nat Commun. 2015; ; 6 : :7126.. 24.
Irigoyen NFirth AEJones JD, et al. High-resolution analysis of coronavirus gene expression by RNA sequencing and ribosome profiling . PLoS Pathog. 2016; ; 12 : :e1005473.. 25.
Stacey SNJordan DWilliamson AJK, et al. Leaky scanning is the predominant mechanism for translation of Human papillomavirus type 16 E7 oncoprotein from E6/E7 bicistronic mRNA . J Virol. 2000; ; 74 : :7284.–7297. 26.
Ingolia NTLareau LFWeissman JS. Ribosome profiling of mouse embryonic stem cells reveals the complexity of mammalian proteomes . Cell. 2011; ; 147 : :789.–802. 27.
Corbin APrats ACDarlix JL, et al. A nonstructural gag-encoded glycoprotein precursor is necessary for efficient spreading and pathogenesis of murine leukemia viruses . J Virol. 1994; ; 68 : :3857.–3867. 28.
Prats ACDe Billy GWang P, et al. CUG initiation codon used for the synthesis of a cell surface antigen coded by the murine leukemia virus . J Mol Biol. 1989; ; 205 : :363.–372. 29.
Yang ZCao SMartens CA, et al. Deciphering poxvirus gene expression by RNA sequencing and ribosome profiling . J Virol. 2015; ; 89 : :6874.–6886. 30.
Bolinger CBoris-Lawrie K. Mechanisms employed by retroviruses to exploit host factors for translational control of a complicated proteome . Retrovirology. 2009; ; 6 : :8.. 31.
Finch LKLing RNapthine S, et al. Characterization of ribosomal frameshifting in Theiler’s murine encephalomyelitis virus . J Virol. 2015; ; 89 : :8580.–8589. 32.
Guerrero SBatisse JLibre C, et al. HIV-1 replication and the cellular eukaryotic translation apparatus . Viruses. 2015; ; 7 : :199.–218. 33.
Somogyi PJenner AJBrierley I, et al. Ribosomal pausing during translation of an RNA pseudoknot . Mol Cell Biol. 1993; ; 13 : :6931.–6940. 34.
Dunn JG, Foo CK, Belletier NG, et al. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster . eLife. 2013; ; 2 : :e01179.. 35.
Miettinen TPBjörklund M. Modified ribosome profiling reveals high abundance of ribosome protected mRNA fragments derived from 3' untranslated regions . Nucleic Acids Res. 2015; ; 43 : :1019.–1034. 36.
Murphy ERigoutsos IShibuya T, et al. Reevaluation of human cytomegalovirus coding potential . Proc Natl Acad Sci USA. 2003; ; 100 : :13585.–13590. 37.
Guttman MRussell PIngolia NT, et al. Ribosome profiling provides evidence that large noncoding RNAs do not encode proteins . Cell. 2013; ; 154 : :240.–251. 38.
Sanz EYang LSu T, et al. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues . Proc Natl Acad Sci USA. 2009; ; 106 : :13939.–13944. 39.
Hussmann JAPatchett SJohnson A, et al. Understanding biases in ribosome profiling experiments reveals signatures of translation dynamics in yeast . PLoS Genet. 2015; ; 11 : :e1005732.. 40.
Gerashchenko MVGladyshev VN. Translation inhibitors cause abnormalities in ribosome profiling experiments . Nucleic Acids Res. 2014; ; 42 : :e134.. 41.
Cenik CCenik ESByeon GW, et al. Integrative analysis of RNA, translation, and protein levels reveals distinct regulatory variation across humans . Genome Res. 2015; ; 25 : :1610.–1621. 42.
Piccirillo CABjur ETopisirovic I, et al. Translational control of immune responses: from transcripts to translatomes . Nat Immunol. 2014; ; 15 : :503.–511. 43.
Marcel VCatez FMertani HCDiaz JJ. Le ribosome. Med Sci (Paris). 2014; ; 30 : :21.–24. |