| |
| Med Sci (Paris). 32(10): 808–812. doi: 10.1051/medsci/20163210006.Identification d’un nouveau facteur de restriction contre l’infection par le virus de l’hépatite B Le complexe SMC5/6 Laetitia Gerossier,1* Adrien Decorsière,2 Fabien Abdul,2 and Olivier Hantz1 1Centre de recherche en cancérologie de Lyon (CRCL), Inserm U1052, CNRS 5286, université de Lyon, 151, cours Albert Thomas, 69424Lyon Cedex, France 2Department of microbiology and molecular medicine, university medical centre (CMU), rue Michel Servet 1, 1211Genève 4, Suisse MeSH keywords: Animaux, Protéines du cycle cellulaire, ADN viral, Hépatite B, Virus de l'hépatite B, Hépatocytes, Humains, Transactivateurs, Transcription génétique, métabolisme, génétique, virologie, physiologie |
L’infection par le virus de l’hépatite B (VHB) constitue un problème majeur de santé publique. En effet, malgré l’existence d’un vaccin préventif efficace, environ 240 millions de personnes sont infectées de façon chronique par le virus au niveau mondial. La situation de porteur chronique du VHB est associée à un risque élevé de développer de graves pathologies du foie comme la cirrhose ou le carcinome hépatocellulaire (CHC). Le CHC représente 95 % des tumeurs malignes primitives du foie, pour lesquelles il n’existe que peu d’options de traitement, et est responsable de 6 000 décès par an en France. L’incidence du CHC augmente du fait des infections virales, notamment celles par le virus de l’hépatite B, associées à presque la moitié des cas de CHC au niveau mondial [1]. Les traitements actuels contre l’hépatite B ne suffisent pas et ne permettent pas d’arrêter l’évolution vers la cirrhose et le cancer car l’ADN viral n’est pas éliminé des cellules [2]. Le VHB se maintient dans la cellule par formation d’un mini chromosome viral (ou ADNccc pour ADN circulaire clos de façon covalente) utilisé comme matrice pour la transcription des ARN messagers (ARNm) et de l’ARN pré-génomique du VHB. La formation et le maintien de l’ADNccc est l’élément clé qui permet au virus de persister de façon chronique dans le foie des personnes infectées. De ce fait, l’ADNccc représente une cible thérapeutique importante. |
La protéine HBx : élément clé de la transcription virale La protéine régulatrice HBx (protéine X du VHB) possède des activités pléiotropes faisant intervenir des interactions avec de multiples partenaires cellulaires nucléaires ou cytoplasmiques (pour revue voir [3]). Parmi les rôles attribués à HBx, on distingue des implications dans de nombreux processus cellulaires tels que la régulation du cycle cellulaire, l’apoptose ou des mécanismes de réparation de l’ADN. Au niveau viral, HBx est aujourd’hui reconnue comme jouant un rôle déterminant dans le maintien de l’activité transcriptionnelle de l’ADNccc. L’absence de HBx inhibe la transcription de l’ADN viral et ainsi la réplication du virus. Les données les plus récentes montrent que HBx module l’activité transcriptionnelle et les modifications épigénétiques [4] (→).
(→) Voir la Nouvelle de L. Rivière et al., m/s n° 5, mai 2016, page 455
Nos travaux précédents ont montré que HBx, en plus de son action sur la stimulation de la transcription du génome du VHB, permet la transcription de toutes les matrices d’ADN sous forme épisomale1. Cette capacité de HBx est liée à son interaction avec DDB1 (DNA damage-binding protein 1) [5], un composant d’un complexe E3 ubiquitine ligase2, qui joue un rôle crucial dans de nombreux processus cellulaires, incluant réplication, réparation de l’ADN et régulation de la chromatine. Dans ce complexe, DDB1 sert de protéine adaptatrice recrutant, directement ou indirectement, les protéines qui vont être ubiquitinées par la E3 ligase et dégradées par le protéasome [6]. Cependant la cible cellulaire de ce complexe HBx-DDB1-E3 ubiquitine ligase était jusqu’à présent inconnue. |
Identification du complexe SMC5/6 comme partenaire de HBx L’identification de la cible de HBx s’est avérée particulièrement difficile, sans doute parce que l’interaction entre HBx et cette dernière est très transitoire dans des conditions naturelles. Pour pallier ce problème, nous avons mis en place une stratégie consistant à bloquer l’ubiquitination de la cible tout en maintenant la liaison entre DDB1 et HBx [7]. Pour cela, nous avons fusionné la protéine HBx avec un mutant de DDB1, incapable de s’incorporer au complexe E3 ubiquitine ligase. Cette protéine de fusion conserve sa capacité de fixation à la cible mais celle-ci n’est plus détruite. Une purification par chromatographie d’affinité suivie d’une analyse en spectrométrie de masse a permis d’identifier cette cible. De façon remarquable, les 6 protéines composant le complexe SMC5/6 (structural maintenance of chromosomes proteins 5/6) ont été retrouvées associées à la protéine de fusion HBx-DDB1 : les 2 protéines SMC 5 et SMC 6 ainsi que les 4 protéines NSE (non SMC element) (Figure 1).
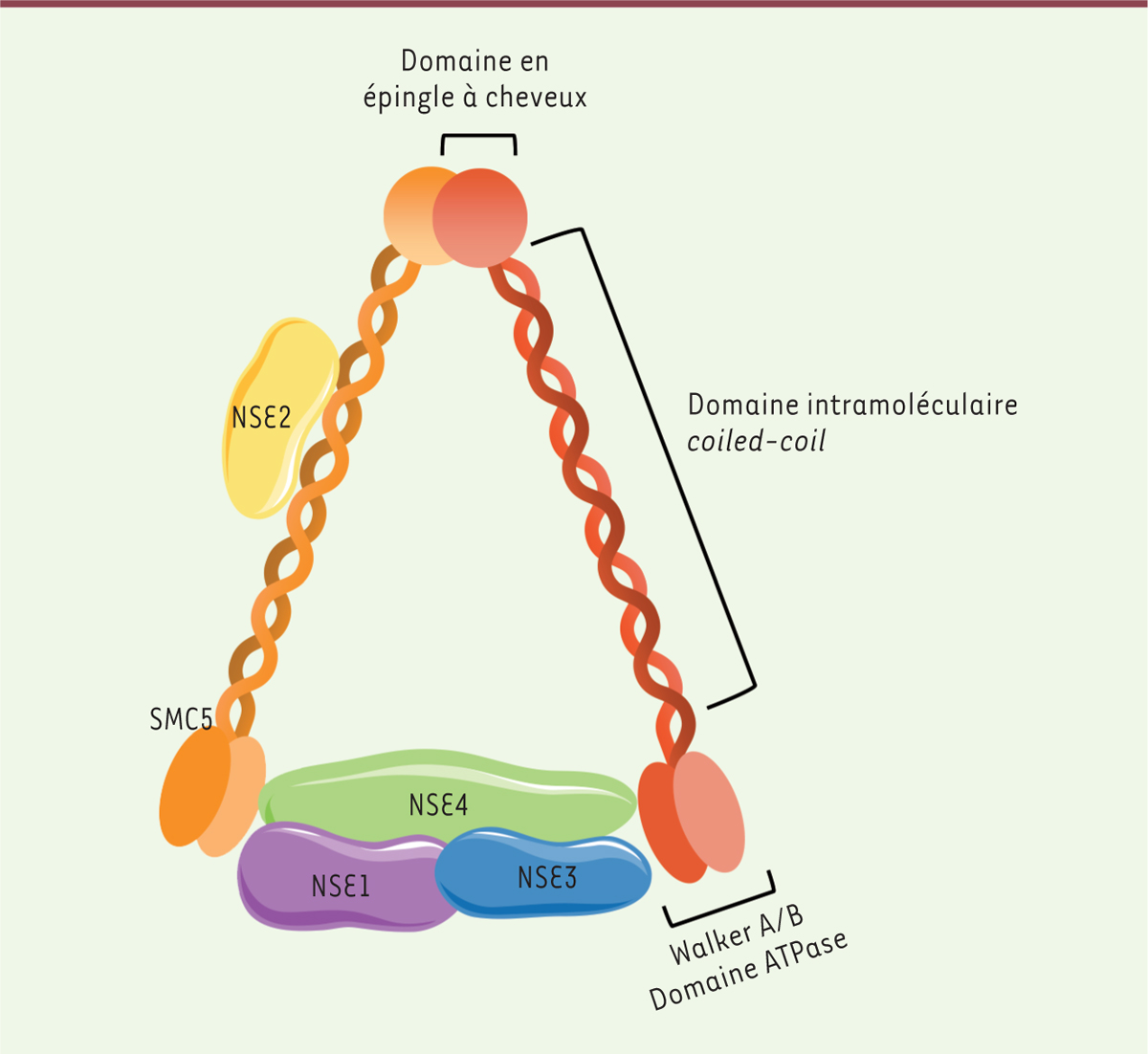
 | Figure 1.
Représentation schématique du complexe SMC5/6. Le complexe SMC5/6 appartient à la famille des SMC ( structural maintenance of chromosomes). Le complexe est composé de 2 protéines SMC5 et SMC6 formant un hétérodimère. Chaque SMC contient aux extrémités N-terminale et C-terminale un domaine globulaire contenant respectivement les motifs ATPase Walker A et Walker B (des motifs à la structure tridimensionnelle hautement conservée). Ces deux régions sont reliées par une longue queue avec des motifs coiled-coil (superhélice). Cette région est séparée en deux par un motif en épingle à cheveu permettant le repliement de la protéine et la formation d’un domaine ATPase fonctionnel. SMC 5 et SMC 6 sont associées à 4 protéines NSE ( non SMC element) portant des activités enzymatiques (activité E3 ubiquitine ligase et activité E3 SUMO ligase) (adapté de [ 12]). |
Dans des hépatocytes primaires humains infectés in vitro par un VHB sauvage, nous avons pu montrer que SMC5/6 est dégradé par HBx via son interaction avec le complexe DDB1-E3 ubiquitine ligase. L’utilisation d’un virus muté pour l’expression de HBx montre qu’en l’absence de HBx, SMC5/6 se fixe sur l’ADN épisomal du virus bloquant sa transcription. En provoquant sa dégradation, HBx active la transcription virale et permet la réplication du VHB (Figure 2). Cette dégradation implique DDB1 (ce qui est montré par l’utilisation de siARN [small interfering RNA] contre DDB1) et l’E3 ubiquitine ligase (qui peut être bloquée par le MNL4924, inhibiteur de la neddylation3).
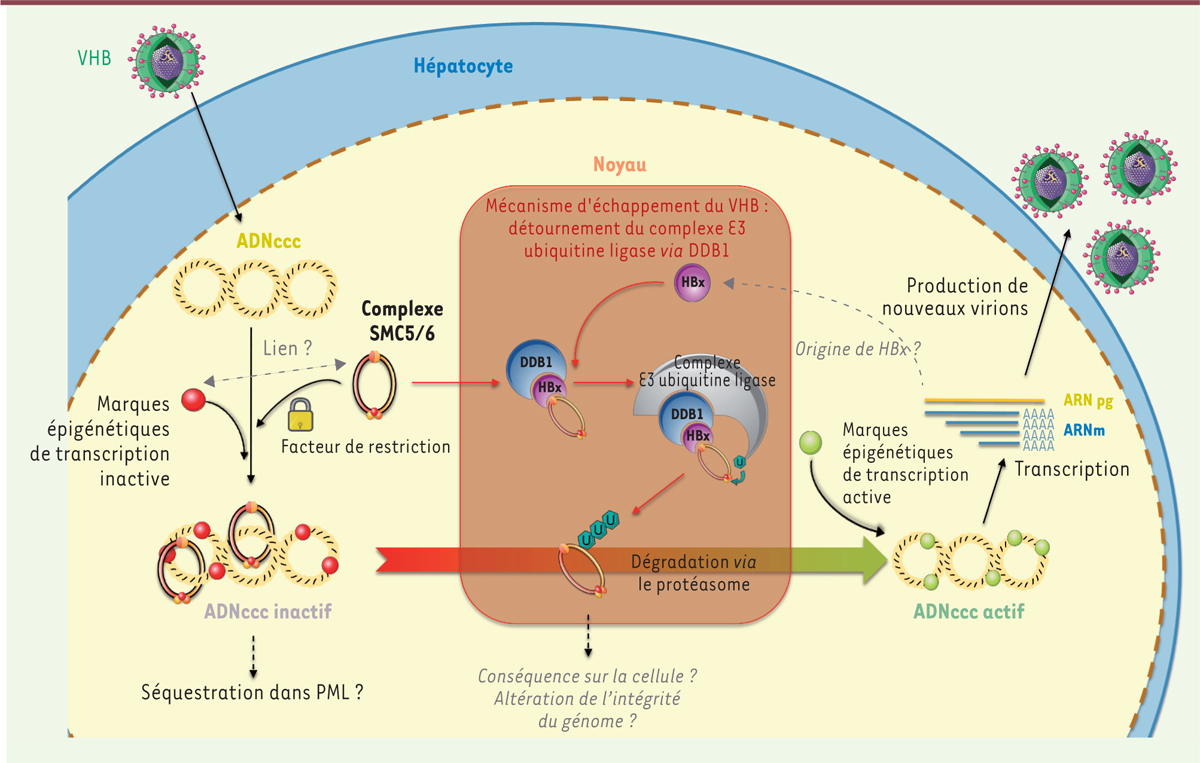
 | Figure 2.
HBx détourne le complexe E3 ubiquitine ligase pour dégrader le facteur de restriction SMC5/6. Lors d’une infection par le virus de l’hépatite B (VHB), l’ADNccc (pour circulaire clos de façon covalente) est reconnu par le facteur de restriction SMC5/6 (structural maintenance of chromosomes proteins 5/6) et subit des modifications épigénétiques conduisant à l’inhibition de la transcription de l’ADN. Le VHB a mis en place un mécanisme d’échappement pour lutter contre ce phénomène. Pour cela, la protéine virale HBx (protéine X du VHB), par le biais de son interaction avec DDB1(DNA damage-binding protein 1), recrute le complexe SMC5/6 au sein du complexe E3 ubiquitine ligase pour permettre l’ubiquitination et la dégradation de SMC5/6. Ainsi, l’ADNccc n’est plus inhibé (on observe des marques épigénétiques de transcription active), la transcription des ARN viraux et de l’ARN pré-génomique (ARN pg) est possible et permet la sécrétion de nouveaux virions. En gris apparaissent les points qui restent à éclaircir à la suite de notre étude, à savoir : quel est le lien entre le complexe SMC5/6 et la présence des modifications épigénétiques sur l’ADNccc ? La liaison entre ADN viral et SMC5/6 engendre-t-elle la séquestration de l’ADNccc dans des corps nucléaires PML (promyelocytic leukaemia nuclear bodies) ? Quelle est l’origine de HBx ? Vient-il de la particule virale ou la transcription du gène x échappe-t-elle à l’inhibition ? Et, enfin, quelles sont les conséquences de la dégradation de SMC5/6 sur la survie et l’intégrité des cellules ? U : ubiquitine. |
Ce phénomène ne serait pas limité au VHB mais concernerait tout l’ADN épisomal comme le suggéraient nos travaux précédents [8]. SMC5/6 se fixe sur l’ADN épisomal et réprime sa transcription. La dégradation de SMC6 par HBx, ou artificiellement par des shARN (small hairpin RNA), provoque la destruction du complexe et restaure la transcription de cet ADN extra-chromosomique. L’ensemble de ces résultats montre que le VHB utilise HBx pour dégrader un facteur cellulaire limitant sa capacité réplicative et que SMC5/6 agit comme un facteur de restriction4,. Ces mécanismes sont retrouvés pour plusieurs virus : la protéine V du paramyxovirus5 SV5 (simian virus 5) se fixe à DDB1 pour permettre la dégradation de STAT (signal transducer and activator of transcription) et ainsi lutter contre les réponses interférons [9] (→) ; la protéine virale X (Vpx) du virus de l’immunodéficience humaine de type 2 (VIH-2) et simienne (SIV, simian immunodeficiency virus) détourne un partenaire cellulaire et le complexe DDB1/E3 ubiquitine ligase pour dégrader le facteur de restriction SMADH1 (SAM [sterile alpha motif] domain and HD domain-containing protein 1) afin d’augmenter le pool de nucléotides disponibles et ainsi faciliter la synthèse de l’ADN viral [10] (→) ; en revanche, le virus de l’herpès simplex utilise sa protéine ICP0 (infected cell polypeptide 0), ayant sa propre activité ubiquitine ligase, pour dégrader les corps nucléaires PML (promyelocytic leukaemia nuclear bodies) séquestrant le virus [11].
(→) Voir la Nouvelle de G. Maarifi et al., m/s n° 2 février 2016, page 141
(→) Voir la Nouvelle de H. Lahouassa et al., m/s n° 11 novembre 2012, page 909
|
Une première question est : quelle est la cible directe du complexe E3 ubiquitine ligase ? En effet, toutes les sous-unités du complexe SMC5/6 ont été retrouvées associées au complexe HBx/DDB1. Tout le complexe SMC5/6 semble dégradé par la présence de HBx, mais il est connu que l’absence d’un seul de ses composants le déstabilise entièrement. Nous n’avons pas pu identifier, en l’absence d’anticorps adéquats, la protéine directement ubiquitinée par le complexe HBx/DDB1/ E3 ubiquitine ligase. Par ailleurs, quel est le rôle de SMC5/6 sur l’ADNccc ? Les mécanismes mis en jeu pour bloquer la transcription restent à élucider. SMC5/6 permet-il le recrutement de facteurs de remodelage de la chromatine entraînant des modifications post-traductionnelles des histones conduisant au blocage de l’ADN ? Ou est-ce que SMC5/6 permet de relocaliser l’ADN étranger ? Il a été montré que le complexe SMC5/6 est colocalisé avec les corps nucléaires PML. Il est donc possible d’imaginer que SMC5/6 recrute l’ADN viral ou épisomal pour le séquestrer au niveau de ces corps nucléaires. Une dernière interrogation subsiste quant à l’origine de HBx en début d’infection. Aucune étude, à notre connaissance, n’a montré la présence de la protéine HBx dans la particule virale. Or comment expliquer la levée de l’inhibition de la transcription de l’ADNccc en l’absence de HBx ? En effet, la synthèse de HBx nécessite la transcription de l’ADNccc, elle-même bloquée par SMC5/6. Peut-on imaginer que le transcrit de HBx échappe à l’inhibition de transcription ? Ou que le temps nécessaire au recrutement de SMC5/6 sur l’ADNccc suffise à la transcription précoce du gène X viral ? Toutes ces questions demeurent et ouvrent de nombreuses pistes de recherche. |
La dégradation de SMC5/6 : quelles conséquences pour la cellule ? Le rôle de facteur de restriction est nouveau pour le complexe SMC5/6. En effet, SMC5/6 appartient au groupe de protéines SMC (structural maintenance of chromosomes) au même titre que la cohésine (composée par les protéines SMC1/3) et la condensine (SMC2/4), respectivement impliquées dans la cohésion et la condensation des chromatides. Le rôle du complexe SMC5/6 n’est pas totalement défini, d’où l’absence d’un nom plus suggestif. Néanmoins, une des fonctions de SMC5/6 connue, bien que contestée, est sa participation dans les réponses aux dommages à l’ADN (DDR, pour DNA damage response) [12] en agissant comme plate-forme permettant le recrutement de protéines aux points de cassures de l’ADN [13]. Ceci implique que la dégradation de SMC5/6 par HBx pourrait avoir un impact sur la structure de la chromatine et influencer la DDR. Il est également connu que l’absence de SMC5/6 dans les souris est létale lors des stades précoces de développement. Les changements de chromatine associés et leurs impacts sur le DDR pourraient permettre une meilleure compréhension de l’activité pro-oncogénique de HBx et de la transformation cellulaire engendrée par le virus de l’hépatite B. Ceci pourrait ouvrir, à terme, une nouvelle voie de traitement pour les CHC avec une telle étiologie.
|
Note ajoutée aux épreuves Des résultats publiés très récemment confirment nos observations et l’importance de la dégradation du complexe SMC5/6 par HBx pour la réplication du VHB [14].
|
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Buendia
MA, Neuveut C. Hepatocellular carcinoma . Cold Spring Harb Perspect Med. 2015; ; 5 : :a021444.. 2.
Revill P, Testoni B, Locarnini S, Zoulim F. Global strategies are required to cure and eliminate HBV infection . Nat Rev Gastroenterol Hepatol. 2016; ; 13 : :239.–248. 3.
Benhenda S, Cougot D, Buendia MA, Neuveut C. Hepatitis B virus X protein molecular functions and its role in virus life cycle and pathogenesis . Adv Cancer Res. 2009; ; 103 : :75.–109. 4.
Rivière L, Gérossier L, Hantz O, Neuveut C. Virus de l’hépatite B et chromatine - Une protéine virale, HBx, interfère avec la machinerie épigénétique de la cellule . Med Sci (Paris). 2016; ; 32 : :455.–458. 5.
Li T, Robert EI, van Breugel PC, et al. A promiscuous alpha-helical motif anchors viral hijackers and substrate receptors to the CUL4-DDB1 ubiquitin ligase machinery . Nat Struct Mol Biol. 2010; ; 17 : :105.–111. 6.
Iovine B, Iannella ML, Bevilacqua MA. Damage-specific DNA binding protein 1 (DDB1): a protein with a wide range of functions . Int J Biochem Cell Biol. 2011; ; 43 : :1664.–1667. 7.
Decorsiere A, Mueller H, van Breugel PC, et al. Hepatitis B virus X protein identifies the Smc5/6 complex as a host restriction factor . Nature. 2016; ; 531 : :386.–389. 8.
van Breugel PC, Robert EI, Mueller H, et al. Hepatitis B virus X protein stimulates gene expression selectively from extrachromosomal DNA templates . Hepatology. 2012; ; 56 : :2116.–2124. 9.
Maarifi G, Dianoux L, Nisole S, Chelbi-Alix MK. Les paralogues de SUMO et la réponse interféron . Med Sci (Paris). 2016; ; 32 : :141.–143. 10.
Lahouassa H, Dragin L, Transy C, Margottin-Goguet F. SAMHD1 prive le VIH des nucléotides essentiels à la synthèse d’ADN viral . Med Sci (Paris). 2012; ; 28 : :909.–910. 11.
Calistri A, Munegato D, Carli I, et al. The ubiquitin-conjugating system: multiple roles in viral replication and infection . Cells. 2014; ; 3 : :386.–417. 12.
Potts PR. The Yin and Yang of the MMS21-SMC5/6 SUMO ligase complex in homologous recombination . DNA repair. 2009; ; 8 : :499.–506. 13.
Lukas J, Lukas C, Bartek J. More than just a focus: The chromatin response to DNA damage and its role in genome integrity maintenance . Nat Cell Biol. 2011; ; 13 : :1161.–1169. 14.
Murphy CM, Xu Y2, Li F1, et al. Hepatitis B virus X protein promotes degradation of SMC5/6 to enhance HBV replication . Cell Rep. 2016; ; 16 : :2846.–2854. |