| |
| Med Sci (Paris). 32(10): 797–799. doi: 10.1051/medsci/20163210002.Une nanoseringue bactérienne pour injecter des protéines dans les cellules eucaryotes Cécile Arrieumerlou1* 1Inserm U1016, Institut Cochin ; CNRS, UMR8104 ; université Paris Descartes, Sorbonne Paris Cité, 22, rue Méchain75014Paris, France MeSH keywords: Aspartate-semialdehyde dehydrogenase, Protéines bactériennes, Techniques cytologiques, Microinjections, Nanotechnologie, Transport de protéines, Protéines de fusion recombinantes, Transfection, Systèmes de sécrétion de type III, Yersinia enterocolitica, génétique, méthodes, composition chimique |
L’étude fonctionnelle des protéines peut nécessiter de faire varier leur concentration dans la cellule et de mesurer l’impact de ces modifications sur les différentes activités cellulaires. La surexpression d’une protéine est généralement obtenue par des techniques de transfection d’un ADNc (ADN complémentaire), de microinjection ou de protéofection1. Celles-ci s’avèrent souvent efficaces. Cependant, ces techniques conduisent à une surreprésentation massive et hétérogène de la protéine, ce qui rend difficile l’interprétation de données dynamiques globales de type -omique. Ces méthodes peuvent également devenir onéreuses lorsqu’elles sont utilisées à grande échelle. Au cours de l’évolution, certaines bactéries ont développé des outils sophistiqués qui leur permettent d’injecter des facteurs de virulence au travers de la membrane plasmique de leurs cellules cibles. C’est le cas, en particulier, de certaines bactéries Gram négatives comme Shigella flexneri, Salmonella typhimurium ou Yersinia enterocolitica qui utilisent un appareil de sécrétion de type III (ST3) appelé injectisome [1]. Il s’agit d’une nanostructure complexe qui est présente à la surface des bactéries et qui fonctionne comme une seringue moléculaire au contact d’une cellule eucaryote (Figure 1) [10] (→).
(→) Voir la Nouvelle de A. Loquet et al., m/s n° 11, novembre 2012, page 926
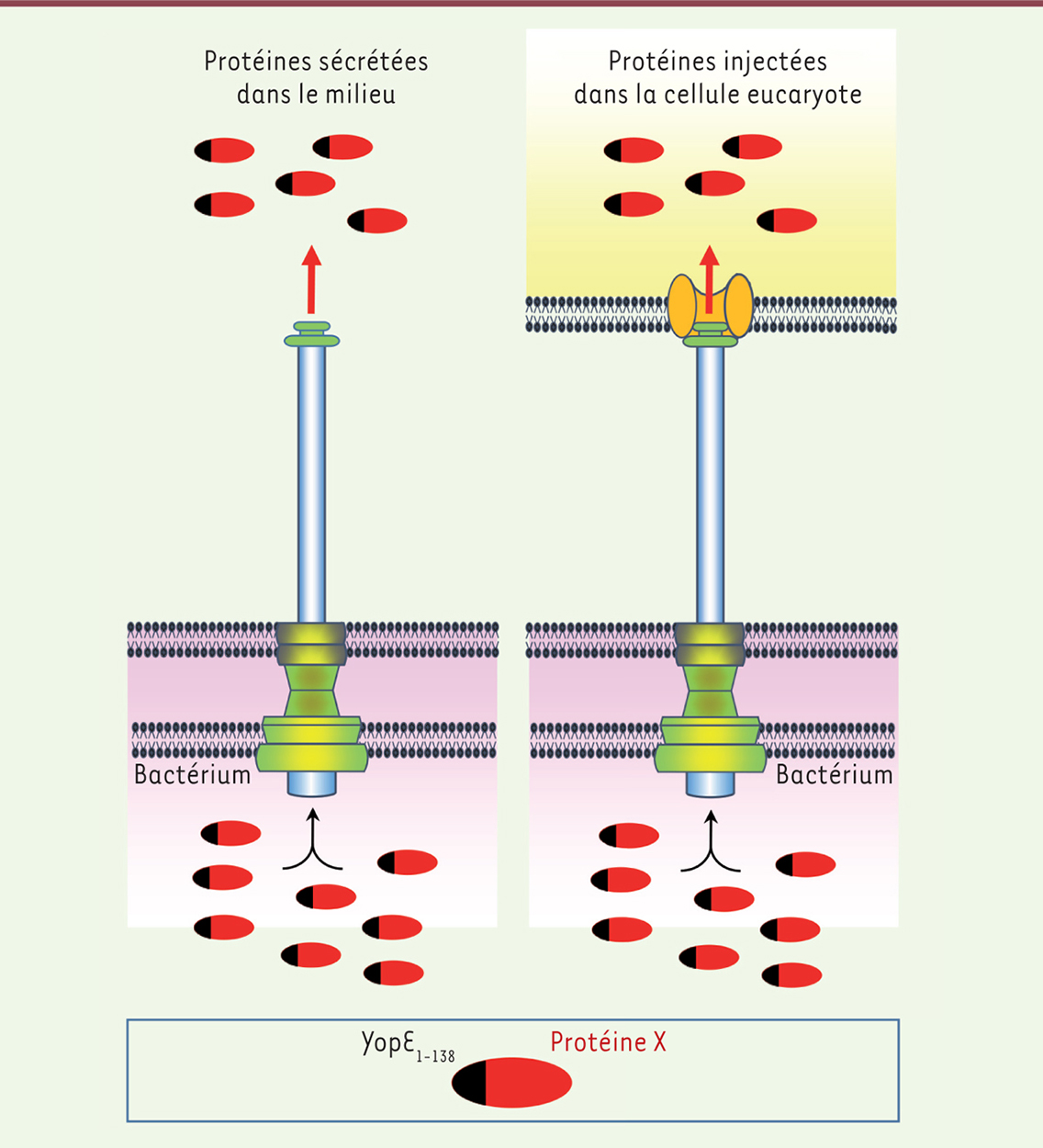
 | Figure 1.
Schéma illustrant l’outil de délivrance des protéines basé sur la ST3 des bactéries Y. enterocolitica. Les protéines d’intérêt sont produites dans les bactéries sous forme de protéines de fusion contenant le fragment YopE1-138. Elles peuvent être sécrétées dans le milieu extérieur (panneau de gauche) ou injectées dans des cellules cibles après contact avec celles-ci (panneau de droite). |
Cette seringue est constituée d’un corps basal inséré dans la paroi bactérienne et d’une structure ressemblant à une aiguille par laquelle transitent les protéines bactériennes (les effecteurs). Dans un processus infectieux, ces protéines perturbent certains des mécanismes de signalisation de la cellule hôte afin de détourner des activités cellulaires au profit de la bactérie. Dans un article publié dans le Journal of Cell Biology, nous avons décrit la façon dont nous avons détourné cette nanoseringue bactérienne pour en faire un nouvel outil de délivrance de protéines dans les cellules eucaryotes, et illustré plusieurs applications possibles de cette méthode en biologie cellulaire [2]. |
Sécrétion ou délivrance intracellulaire de protéines fusionnées au fragment YopE 1-138Cet outil est basé sur l’injectisome des bactéries Yersinia enterocolitica. La méthode utilise une souche déficiente pour tous les effecteurs connus de la bactérie, à savoir Yop (Yersinia outer protein) H, P, E, M et T (souche ∆HOPEMT). Le gène codant l’aspartate-β-semialdéhyde déshydrogénase a également été délété afin de rendre la bactérie incapable de se répliquer en l’absence d’acide meso-2,6-diaminopimelique exogène, ce qui minimise les risques liés à son utilisation. Enfin, la souche utilisée contient un plasmide de clonage dont le design a été optimisé pour faciliter la production de protéines de fusion. Ces protéines sont constituées des 138 premiers acides aminés de l’effecteur YopE (YopE1-138) et de la protéine d’intérêt. Le fragment YopE1-138, comprenant un peptide signal pour la ST3 et un site de liaison à la protéine SycE, chaperonne associée à YopE, avait en effet été préalablement décrit comme suffisant pour induire une efficacité élevée de translocation par l’injectisome [3]. La mise en œuvre de la méthode n’est pas réservée aux spécialistes des infections bactériennes. Elle peut être utilisée dans deux buts : la sécrétion d’une protéine d’intérêt dans le milieu extracellulaire, ou l’injection dans des cellules cibles après leur mise en contact avec les bactéries (Figure 1). Afin de valider le second objectif, nous avons montré que cette méthode permet d’injecter une protéine de fusion, étiquetée par l’épitope Myc2, (YopE1-138-Myc), en quelques minutes et de manière uniforme dans l’ensemble de la population cellulaire ciblée. Cette injection peut s’opérer dans différents types de cellules comme les fibroblastes, les cellules épithéliales et endothéliales ou les lymphocytes T. Ce système est également efficace pour délivrer des effecteurs de type III fonctionnels qui proviennent d’autres bactéries que Y. enterocolitica. Ainsi, nous avons pu observer la déphosphorylation de la MAPK (mitogen-activated protein kinase) p38 induite après stimulation par le TNFα, après l’injection d’OspF, un effecteur de la bactérie S. flexneri qui possède une activité phosphothréonine lyase3. Il peut également servir à injecter des substrats de la sécrétion de type IV, alternative à la ST3 utilisée par de nombreuses bactéries pour injecter des facteurs de virulence dans des cellules cibles. L’utilisation de l’outil de délivrance basé sur la ST3 n’est pas restreinte aux seules protéines bactériennes. En effet, de nombreuses protéines humaines de différentes tailles et structures peuvent également être sécrétées. L’efficacité dépend de la protéine testée. Elle peut être facilement mesurée, après déclenchement de la sécrétion par un choc thermique à 37 °C et une privation de calcium, en déterminant la quantité de protéines libérées par les bactéries dans le milieu extracellulaire. Les protéines humaines injectées dans des cellules eucaryotes sont biologiquement actives. Ce point a été plus particulièrement étudié pour le facteur pro-apoptotique BID (BH3-interacting domain death agonist). Au cours de l’apoptose, BID est clivé par la caspase-8 et un fragment tronqué (tBID) est libéré [4]. Celui-ci se relocalise au niveau de la mitochondrie déclenchant le relargage du cytochome C, l’activation de la caspase-3 et les processus terminaux de mort cellulaire par apoptose tels que la condensation de la chromatine et le détachement du substrat, pour les cellules adhérentes. Nous avons montré que l’injection de tBID par l’outil de ST3 induit l’apoptose de cellules en culture par un mécanisme qui met en jeu la caspase-3 et qui est similaire à celui décrit dans la littérature [4]. |
Les applications en biologie cellulaire La méthode que nous décrivons et qui permet de délivrer en quelques minutes des protéines fonctionnelles dans des cellules eucaryotes, a de nombreuses applications en biologie cellulaire. Au-delà de l’étude d’une protéine, elle permet d’analyser les interactions fonctionnelles entre plusieurs protéines après qu’elles aient été injectées simultanément lors d’expériences de co-infection. Ainsi, nous avons pu vérifier les activités antagonistes des protéines bactériennes SopE et Sptp. Ces protéines possèdent, respectivement, des activités GEF (guanine nucleotide exchange factor) et GAP (GTP-ase-activating protein) pour la protéine Cdc42 (cell division cycle 42)4 [5]. Alors que l’injection de SopE seule induit la formation de câbles d’actine polymérisée, l’injection concomitante de Sptp bloque l’effet de SopE sur le réseau d’actine. Cette méthode offre la possibilité d’un ciblage subcellulaire spécifique par l’utilisation de protéines fusionnées avec des anticorps à domaine unique (nanobodies). En effet, en utilisant des cellules exprimant de manière stable les protéines marquées par la GFP (green fluorescent protein), l’histone 2B-GFP et la Rab2a-GFP, nous avons montré qu’il était possible de cibler la localisation d’une protéine de fusion, contenant un nanobody spécifique de GFP [6], vers le noyau ou la région périnucléaire. Il est également possible d’injecter des protéines fluorescentes. La GFP est notoirement décrite pour être un très mauvais substrat de la ST3 [7]. Notre outil, basé sur la translocation de protéines fusionnées à YopE1-138, permet une efficacité de translocation de la GFP faible mais reproductible, et il a un bon rendement pour la dsRed, une autre protéine fluorescente. Dans les deux cas, l’addition d’une séquence de localisation nucléaire induit la localisation de la protéine dans le noyau. La présence du fragment YopE1-138 au sein de la séquence protéique, peut perturber la localisation de certaines protéines et donc interférer avec leurs fonctions. Afin de minimiser les effets liés à YopE1-138, nous avons optimisé notre outil pour libérer, si nécessaire, la protéine d’intérêt de ce fragment. Après translocation dans la cellule cible, celui-ci peut en effet être clivé par la protéase virale TEV (tobacco etch virus), elle-même injectée par co-infection avec des bactéries produisant YopE1-138-TEV. Une option alternative consiste à fusionner la protéine d’intérêt à l’ubiquitine, celle-ci étant ensuite clivée dans sa partie C-terminale par un groupe de protéases endogènes spécifiques. L’outil de délivrance basé sur la ST3 offre la possibilité de délivrer des protéines in vivo. Ce point a été confirmé en mesurant l’impact, dans des embryons du poisson zèbre, de la translocation de la protéine z-BIM, le facteur pro-apoptotique le mieux caractérisé chez le poisson zèbre [8]. Contrairement à l’injection de bactéries contrôles (YopE1-138), l’injection de bactéries exprimant la protéine YopE1-138-zBIM induit une réponse apoptotique forte, visualisée par une augmentation du clivage de la caspase-3 dans les tissus du poisson. Cette méthode peut, finalement, être combinée avec des études de type -omique afin de caractériser l’impact de la translocation rapide d’une protéine fonctionnelle sur un réseau cellulaire complexe. Nous avons plus particulièrement caractérisé les effets de tBID sur le réseau de signalisation, en couplant son injection par ST3 à la phosphoprotéomique. La translocation de tBID induit des changements de phosphorylation de centaines de protéines impliquées dans des fonctions cellulaires variées comme la traduction ou le contrôle du cytosquelette. Parmi celles-ci, nous avons identifié environ 70 substrats de la caspase-3 et montré qu’il existait un enrichissement de ces substrats dans le phosphoprotéome de tBID. Ainsi, l’injection de tBID par notre nouvel outil de délivrance combinée à la phosphoprotéomique nous a permis de mieux caractériser ses effets au cours de l’apoptose, et de confirmer des travaux récents suggérant qu’il existe une corrélation entre phosphorylation et clivage par la caspase-3 [9]. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Cornelis GR. The type III secretion injectisome . Nat Rev Microbiol. 2006; ; 4 : :811.–825. 2.
Ittig SJ, Schmutz C, Kasper CA, et al. A bacterial type III secretion-based protein delivery tool for broad applications in cell biology . J Cell Biol. 2015; ; 211 : :913.–931. 3.
Wolke S, Ackermann N, Heesemann J. The Yersinia enterocolitica type 3 secretion system (T3SS) as toolbox for studying the cell biological effects of bacterial Rho GTPase modulating T3SS effector proteins . Cell Microbiol. 2011; ; 13 : :1339.–1357. 4.
Li H, Zhu H, Xu CJ, Yuan J. Cleavage of BID by caspase 8 mediates the mitochondrial damage in the Fas pathway of apoptosis . Cell. 1998; ; 94 : :491.–501. 5.
Stebbins CE, Galan JE. Structural mimicry in bacterial virulence . Nature. 2001; ; 412 : :701.–705. 6.
Saerens D, Frederix F, Reekmans G, et al. Engineering camel single-domain antibodies and immobilization chemistry for human prostate-specific antigen sensing . Anal Chem. 2005; ; 77 : :7547.–7555. 7.
Jacobi CA, Roggenkamp A, Rakin A, et al. In vitro and in vivo expression studies of yopE from Yersinia enterocolitica using the gfp reporter gene . Mol Microbiol. 1998; ; 30 : :865.–882. 8.
Jette CA, Flanagan AM, Ryan J, et al. BIM and other BCL-2 family proteins exhibit cross-species conservation of function between zebrafish and mammals . Cell Death Differ. 2008; ; 15 : :1063.–1072. 9.
Dix MM, Simon GM, Wang C, et al. Functional interplay between caspase cleavage and phosphorylation sculpts the apoptotic proteome . Cell. 2012; ; 150 : :426.–440. 10.
Loquet A, Habenstein B, Demers JP, et al. Structure d’une nanomachine bactérienne. Med Sci (Paris). 2012; ; 28 : :926.–928. |