| |
| Med Sci (Paris). 32(4): 335–339. doi: 10.1051/medsci/20163204008.Lutter contre les infections bactériennes : le système immunitaire des plantes est aussi très efficace ! Justine Claverie,1* Lény Teyssier,1,2* Daphnée Brulé,1 Marie-Claire Héloir,1 Jean-Louis Connat,2 Olivier Lamotte,1b and Benoît Poinssot1a 1UMR 1347 Agroécologie, AgroSup Dijon, CNRS, Inra, Univ. Bourgogne Franche-Comté, F-21000Dijon, France 2UMR 866 Lipides Nutrition Cancer, Inserm, Univ. Bourgogne Franche-Comté, F-21000Dijon, France MeSH keywords: Alarmines, Animaux, Arabidopsis, Infections bactériennes, Humains, Système de signalisation des MAP kinases, Molécules contenant des motifs associés aux pathogènes, Maladies des plantes, Immunité des plantes, Transduction du signal, physiologie, génétique, immunologie, métabolisme, effets des médicaments et substances chimiques, pharmacologie, microbiologie |
Les micro-organismes ont conquis l’ensemble des niches écologiques de la biosphère et sont les êtres vivants les plus répandus à la surface de la terre. Les plantes et les animaux ont donc appris à vivre et à cohabiter avec eux, qu’ils leur soient bénéfiques ou nuisibles. Comme les animaux, les plantes possèdent une immunité innée leur permettant de percevoir les micro-organismes agressifs. Des avancées majeures ont récemment permis d’identifier, chez les plantes, des récepteurs de l’immunité qui reconnaissent les signaux de danger qui sont associés à une invasion microbienne [1]. Ces signaux sont de deux types : soit des motifs moléculaires directement associés aux agents pathogènes, les PAMP (pathogen-associated molecular pattern), soit des molécules libérées lors de lésions tissulaires de l’hôte, les DAMP (danger-associated molecular pattern). |
Systèmes moléculaires de perception des agressions microbiennes Chez les animaux comme chez les plantes, les DAMP et les PAMP sont reconnus par des récepteurs appelés PRR (pattern recognition receptor) [1, 2] (→).
(→) Voir la Synthèse de Y. Jamilloux et T. Henry, m/s n° 11, novembre 2013,page 975
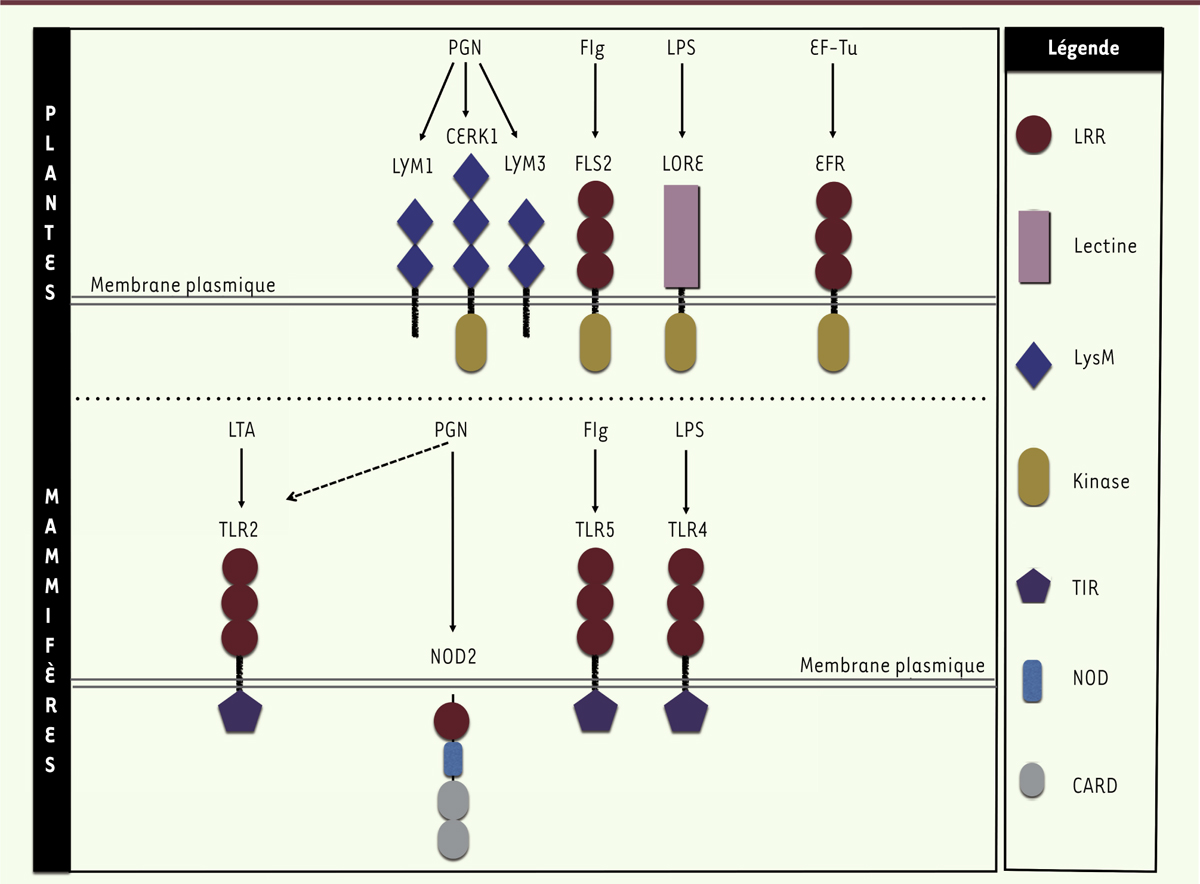
Certains PRR doivent s’associer en complexes moléculaires avec d’autres protéines afin de reconnaître leur ligand. Chez les animaux, les PRR ont été identifiés pour la plupart des PAMP connus. Parmi ces récepteurs, on trouve par exemple les TLR1,, les NLR et les CLR. Les orthologues2 de ces trois types de récepteurs ne semblent pas exister chez les plantes. Toutefois, des domaines protéiques de type LRR (leucine-rich repeat) sont conservés entre les PRR des deux règnes. Chez les plantes, les PRR reconnaissant des PAMP bactériens appartiennent à trois familles de protéines : les RLK, les RLP et les NB-LRR. Certains PAMP bactériens, tels que la flagelline (Flg), le lipopolysaccharide (LPS) et le peptidoglycane (PGN), sont reconnus à la fois chez les plantes et les animaux (Figure 1). Toutefois les épitopes reconnus peuvent être différents entre les deux règnes. Ainsi, pour la flagelline, l’épitope de 22 acides aminés (Flg22) localisé dans la partie NH2-terminale est reconnu par le RLK FLS2 de la plante Arabidopsis thaliana, alors que, chez les mammifères, d’autres acides aminés forment l’épitope peptidique perçu par le TLR5 [3]. Chez ces derniers, la flagelline peut aussi être perçue par le NLR cytoplasmique NAIP5. Pour le LPS, un PRR nommé LORE a récemment été identifié chez A. thaliana [4] alors que, pour les mammifères, le récepteur reconnaissant le LPS est le TLR4 qui forme un complexe multimérique avec ses protéines accessoires. Enfin, la reconnaissance du PGN est assurée par le RLK CERK1 et les RLP LYM1 et LYM3 chez A. thaliana [3] tandis que, chez les animaux, le récepteur cytosolique NOD2 et le récepteur membranaire TLR2 assurent cette reconnaissance.
 | Figure 1.
Différences et similitudes de perception des PAMP bactériens chez les plantes et les mammifères. Le système immunitaire emploie des classes variées de PRR qui permettent la détection rapide des bactéries au site de l’infection. Tandis que les PRR identifiés chez les plantes sont principalement membranaires, les récepteurs des mammifères peuvent être membranaires, cytoplasmiques ou localisés à la membrane des endosomes. Parmi les PAMP bactériens communément perçus par les plantes et les mammifères se trouvent le LPS, le PGN et la flagelline. Quelques couples PRR/PAMP identifiés chez les végétaux ou les mammifères sont représentés sur cette figure. Chez les plantes, le facteur d’élongation EF-Tu ( elongation factor Tu) d’ Escherichia coli est reconnu par le RLK EFR [ 3]. Son rôle chez les animaux n’est pas décrit. À l’inverse, l’acide lipotéichoïque (LTA) est reconnu par TLR2 chez les animaux mais sa perception n’est pas décrite chez les plantes. PGN : peptidoglycane ; Flg : flagelline ; LPS : lipopolysaccharide ; EF-Tu : elongation factor Tu ; EFR : elongation factor receptor ; LTA : acide lipotéichoïque ; TLR : toll like receptor ; FLS2 : flagellin sensing 2 ; NOD : nucleotide-binding oligomerization domain proteins ; LRR : leucin rich repeat ; LysM : lysin motif ; TIR : Toll/ interleukin 1-receptor domain ; CARD : caspase recruitment domain ; RLK : receptor-like kinase. |
|
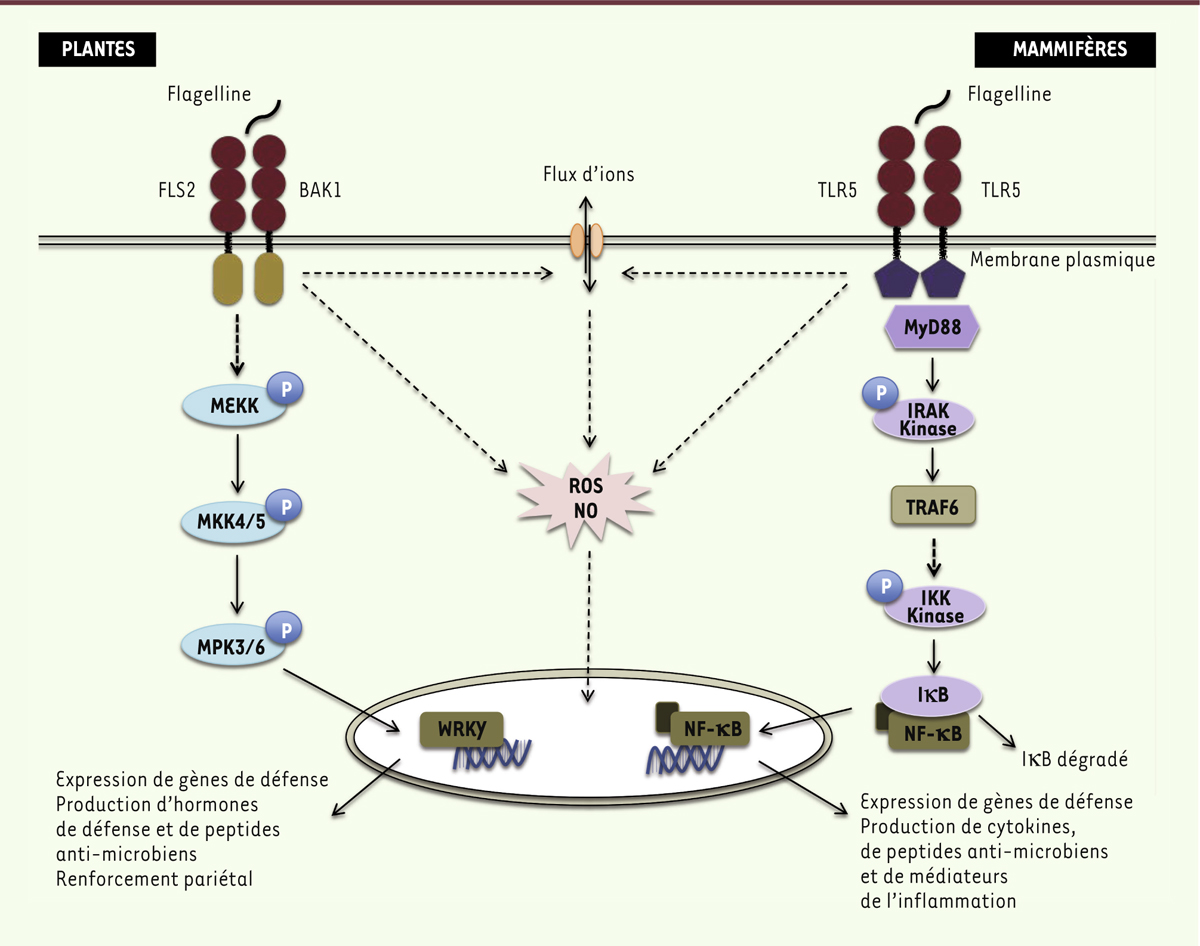
Signalisation et réponses immunitaires L’exemple de la perception de la flagelline par son PRR dans le règne animal ou végétal permet de rendre compte des similitudes dans la signalisation cellulaire qui mène à l’induction d’une réponse immunitaire (Figure 2).
 | Figure 2.
Principaux composants de la signalisation induite par la flagelline chez les plantes et les mammifères. La perception du PAMP par son PRR associé induit une cascade de signalisation conservée menant à l’activation de l’immunité innée chez les végétaux et les animaux. FLS2 : flagellin sensitive 2 ; BAK1 : BRI1 (brassinosteroid insensitive 1)-associated kinase 1 ; MEKK : MAP kinase kinase kinase ou MPK kinase kinase ou MAP3K ; MKK : MAP kinase kinase ou MPK kinase ou MAP2K ; MPK : mitogen-activated protein kinase ou MAPK ; WRKY : facteurs de transcription WRKY ; ROS : reactive oxygen species ; NO : nitric oxide ; TLR5 : Toll-like receptor 5 ; MyD88 : myeloid differentiation primary response gene 88 ; IRAK : interleukin-1 receptor-associated kinase ; TRAF6 : TNF (tumor necrosis factor) receptor associated factor 6 ; IKK : IκB kinase ; IκB : inhibitor of nuclear factor kappa-B ; NF-κB : nuclear factor-kappa B. |
Chez les plantes, la perception de Flg22 par le récepteur FLS2 entraîne son association avec BAK1, un co-récepteur de type LRR-RK. La formation de ce complexe fonctionnel entraîne, entre autre, l’activation d’une cascade de MAP kinases, impliquant notamment MEKK, MKK4/5 et MPK3/6, qui conduit à l’activation de facteurs de transcription comme ceux de la famille WRKY, et aboutit à la transcription de gènes de défense [5]. Ces gènes codent notamment des enzymes impliquées dans la synthèse de phytohormones (comme l’éthylène, l’acide jasmonique et l’acide salicylique), qui permettent d’amplifier la réponse immunitaire et d’avertir les cellules voisines. Ceci est à mettre en parallèle avec ce qui est observé dans les modèles animaux chez lesquels la reconnaissance de la flagelline par le TLR5 provoque le recrutement de protéines adaptatrices comme MyD88, entraînant l’autophosphorylation des kinases de la famille IRAK et leur association avec TRAF6. S’en suit l’induction d’une cascade de phosphorylation impliquant notamment les kinases du complexe IKK. Ce complexe permet la dégradation d’IκB et la translocation dans le noyau du facteur de transcription NF-κB, régulant ainsi l’expression de gènes codant des médiateurs de l’inflammation (comme les cytokines et les chémokines) qui vont permettre le recrutement de cellules de défense spécialisées. La transduction du signal chez les plantes et chez les mammifères implique aussi la modification de flux d’ions au travers des membranes, en particulier du Ca2+, ainsi que la production de ROS (reactive oxygen species) ou de NO (monoxyde d’azote). L’ensemble de ces seconds messagers contribue également à l’expression d’un transcriptome de défense. Chez les végétaux, la paroi cellulaire constitue une barrière physique de défense limitant la pénétration de l’agent pathogène. Une des premières réponses de la plante consiste à renforcer cette barrière structurale. Ainsi, l’accumulation de glycoprotéines riches en hydroxyproline et les réactions de pontage entre ces molécules augmentent sa résistance mécanique, comme cela est observé avec le collagène lors de la fibrose des tissus animaux. De plus, des appositions pariétales, structures constituées de composés phénoliques (lignine, subérine) ou de callose (polymère de β-1,3 glucanes), sont souvent observées aux sites d’infection et permettent de bloquer la pénétration des bactéries [6]. Les plantes et les animaux synthétisent des composés antimicrobiens dont certains sont communs aux deux règnes, comme ceux de la famille des défensines et des thionines [7]. Certaines molécules antimicrobiennes ne sont, en revanche, produites que par les cellules végétales comme les phytoalexines, des métabolites secondaires qui bloquent le développement des agresseurs. Dans certains cas, l’immunité des plantes peut conduire à une mort cellulaire programmée, appelée réponse hypersensible, qui permet de restreindre la propagation des micro-organismes. Elle se traduit par l’apparition de lésions nécrotiques localisées aux sites d’infection. Les mécanismes conduisant à cette mort cellulaire sont encore mal connus. Elle peut cependant être comparée, par certains aspects, à l’apoptose ou à la pyroptose3 que l’on observe chez les animaux. Plusieurs évènements de la mort cellulaire programmée sont en effet similaires entre les deux règnes, tels que le relargage du cytochrome c par la mitochondrie, l’activation d’enzymes de type caspases ou caspases-like, la condensation de la chromatine, la fragmentation de l’ADN et la rétraction du cytoplasme [8]. |
Contournement de l’immunité par les agents pathogènes Comme chez les animaux, les pathogènes qui infectent les plantes sont capables de manipuler les fonctions cellulaires de leur hôte par l’intermédiaire d’effecteurs (protéines, toxines, etc.), facilitant ainsi leur dissémination. C’est le cas pour la bactérie Pseudomonas syringae pv. tomato (Pst) qui injecte certains de ses effecteurs dans les cellules végétales par l’intermédiaire d’un système de sécrétion de type III4. Ces effecteurs ciblent largement les voies de signalisation cellulaire qui aboutissent à la réponse immunitaire mais également celles qui induisent l’ouverture des stomates [9]. Les stomates sont des pores naturels, localisés à la surface des feuilles, qui contrôlent les échanges gazeux. Délimités par deux cellules dites de garde, ces pores sont de véritables portes d’entrée pour les micro-organismes pathogènes. La reconnaissance des PAMP bactériens par les PRR de ces cellules conduit à leur fermeture. Parmi les effecteurs de la bactérie Pst, une toxine, la coronatine, provoque une réouverture des stomates facilitant ainsi la dissémination du pathogène [10]. Les microbes qui infectent les animaux utilisent des stratégies similaires pour infecter leur hôte [1]. Leur détection par les cellules épithéliales ou par les cellules du système immunitaire induit une réponse de type inflammatoire qui, lorsqu’elle n’est pas maîtrisée, peut entraîner des complications sévères (dommages tissulaires, etc.). Néanmoins, une voie dite inhibitrice, impliquant l’activation de récepteurs membranaires à motif ITIM (immunoreceptor tyrosine-based inhibitory motif), permet de contrôler cette réponse. Plusieurs bactéries pathogènes sont capables de manipuler cette voie inhibitrice via des effecteurs qui peuvent activer directement ces récepteurs. D’autres effecteurs bactériens possèdent un motif ITIM qui, mimant l’activation de ces récepteurs, atténuent les défenses de l’hôte. |
Ainsi, les plantes utilisent contre l’agression bactérienne des stratégies de défense très proches de celles des vertébrés. La connaissance des mécanismes de l’immunité innée des animaux pourrait donc guider les recherches visant à mieux comprendre les voies de défense induites par les PAMP et les DAMP chez les végétaux. À l’heure où les préoccupations agro-écologiques nous orientent vers une réduction des intrants5 en agriculture, ces connaissances permettront de développer de nouvelles stratégies de bio-contrôle basées sur la stimulation des défenses naturelles des plantes. Néanmoins, les similitudes des mécanismes de l’immunité entre végétaux et animaux mettent en exergue la nécessité de rester attentifs aux interférences qui pourraient advenir avec la santé des utilisateurs. En effet, l’utilisation de produits stimulant l’immunité des plantes pourrait également induire des réponses inflammatoires chez l’homme. |
Les auteurs déclarent n’avoir aucun conflit d’intérêt concernant les données publiées dans cet article.
|
Justine Claverie bénéficie d’une thèse co-financée par l’INRA et le Conseil Régional de Bourgogne, Leny Teyssier bénéficie d’une thèse financée par le Conseil Régional de Bourgogne, dans le cadre du projet FEDER « PHYTOSAFE ». Daphnée Brulé bénéficie d’un financement de l’ANR (projet PATRIC, ANR-13-KBBE-0001).
|
Glossaire
|
BAK1 :
|
BRI1 (brassinosteroid insensitive 1)-associated kinase 1
|
|
CARD :
|
caspase recruitment domain
|
|
CERK1 :
|
chitin elicitor receptor kinase 1
|
|
CLR :
|
C-type lectin receptor
|
|
DAMP :
|
danger-associated molecular pattern
|
|
Flg :
|
flagelline |
|
FLS2 :
|
flagellin sensing 2
|
|
IkB :
|
inhibitor of nuclear factor kappa-B
|
|
IKK :
|
IκB kinase
|
|
IRAK :
|
interleukin-1 receptor-associated kinase
|
|
ITIM :
|
immunoreceptor tyrosine-based inhibitory motif
|
|
LORE :
|
lipooligosaccharide-specific reduced elicitation
|
|
LPS :
|
lipopolysaccharide |
|
LRR :
|
leucine-rich repeat
|
|
LRR-RK :
|
leucine-rich repeat receptor kinase
|
|
LYM1/3 :
|
lysin motif domain-containing GPI(glycosylphosphatidylinositol)-anchored protein 1/3
|
|
MAP :
|
mitogen-activated protein
|
|
MEKK :
|
MAP kinase kinase kinase |
|
MKK4/5 :
|
MAP kinase kinase 4/5 |
|
MPK3/6 :
|
MAP kinase 3/6 |
|
MyD88 :
|
myeloid differentiation primary response gene 88
|
|
NAIP5 :
|
NLR family, apoptosis inhibitory protein 5
|
|
NB-LRR :
|
nucleotide-binding site-leucine-rich repeat
|
|
NF-kB :
|
nuclear factor-kappa B
|
|
NLR :
|
nucleotide-binding oligomerization domain receptor
|
|
NO :
|
monoxyde d’azote |
|
NOD2 :
|
nucleotide-binding oligomerization domain protein 2
|
|
PAMP :
|
pathogen-associated molecular pattern
|
|
PGN :
|
peptidoglycane |
|
PRR :
|
pattern recognition receptor
|
|
Pst :
|
Pseudomonas syringae pv. tomato
|
|
RLK :
|
receptor-like kinase
|
|
RLP :
|
receptor-like protein
|
|
ROS :
|
reactive oxygen species
|
|
TIR :
|
Toll/ interleukin 1-receptor domain
|
|
TLR :
|
toll-like receptor
|
|
TRAF6 :
|
TNF (tumor necrosis factor) receptor associated factor 6
|
|
WRKY :
|
facteurs de transcription WRKY |
|
Footnotes |
1.
Boller T, Felix G. A renaissance of elicitors : perception of microbe-associated molecular patterns and danger signals by pattern-recognition receptors . Annu Rev Plant Biol. 2009; ; 60 : :379.–406. 2.
Jamilloux Y, Henry T. Les inflammasomes : plates-formes de l’immunité innée . Med Sci (Paris). 2013; ; 29 : :975.–984. 3.
Zipfel C. Plant pattern-recognition receptors . Trends Immunol. 2014; ; 35 : :345.–351. 4.
Ranf S, Gisch N, Schäffer M, et al. A lectin S-domain receptor kinase mediates lipopolysaccharide sensing in Arabidopsis thaliana . Nat Immunol. 2015; ; 16 : :426.–433. 5.
Asai T, Tena G, Plotnikova J, et al. MAP kinase signalling cascade in Arabidopsis innate immunity . Nature. 2002; ; 415 : :977.–983. 6.
Underwood W. The plant cell wall : a dynamic barrier against pathogen invasion . Front Plant Sci. 2012; ; 3 : :85.. 7.
Van Loon LC, Rep M, Pieterse CM. Significance of inducible defense-related proteins in infected plants . Annu Rev Phytopathol. 2006; ; 44 : :135.–162. 8.
Fagundes D, Bohn B, Cabreira C, et al. Caspases in plants : metacaspase gene family in plant stress responses . Funct Integr Genomics. 2015; ; 15 : :639.–649. 9.
Kazan K, Lyons R. Intervention of phytohormone pathways by pathogen effectors . Plant Cell. 2014; ; 26 : :2285.–2309. 10.
Asai S, Shirasu K. Plant cells under siege : plant immune system versus pathogen effectors . Curr Opin Plant Biol. 2015; ; 28 : :1.–8. 11.
Van Avondt K, van Sorge NM, Meyaard L. Bacterial immune evasion through manipulation of host inhibitory immune signaling . PLoS Pathog. 2015; ; 11 : :e1004813.. |