| |
| Med Sci (Paris). 32(4): 323–326. doi: 10.1051/medsci/20163204004.Coopération entre mutation somatique et variant génétique de susceptibilité dans le sarcome d’Ewing Thomas G.P. Grünewald,1,2,3 Pascale Gilardi-Hebenstreit,4 Patrick Charnay,4 and Olivier Delattre2,3,5* 1Laboratory for Pediatric Sarcoma Biology, Institute of Pathology of the LMU Munich, Thalkirchnerstrasse 36, 80337Munich, Allemagne 2Institut Curie, PSL Research University, unité de Génétique et Biologie des Cancers, 26, rue d’Ulm, 75248Cedex 05 Paris, France 3Inserm U830, Institut Curie, Centre de recherches, 26, rue d’Ulm, 75248Paris, France 4École normale supérieure, PSL Research University, Inserm U1024, CNRS UMR8197, Institut de biologie de l’ENS (IBENS), 46, rue d’Ulm, 75005Paris, France 5Unité Génétique Somatique (UGS), Institut Curie, Centre hospitalier, 26, rue d’Ulm, 75248Paris, France MeSH keywords: Tumeurs osseuses, Facteur de transcription EGR-2, Épistasie, Régulation de l'expression des gènes tumoraux, Prédisposition génétique à une maladie, Humains, Mutation, Protéines de fusion oncogènes, Polymorphisme de nucléotide simple, Sarcome d'Ewing, génétique, anatomopathologie, physiologie |
L’analyse génétique des cancers humains comporte, aujourd’hui, deux voies d’études principales. La première concerne l’étude des prédispositions et susceptibilités individuelles au développement de la maladie. Elle repose sur l’investigation des formes familiales de cancer et, de plus en plus, sur des approches d’association entre variants génétiques et données médicales, fondées sur l’épidémiologie génétique (en particulier les genome wide association studies, ou GWAS). L’autre voie concerne l’étude des altérations somatiques acquises par les cellules tumorales au cours de leur développement. Elle repose en grande partie sur le séquençage du génome des cellules tumorales [1]. Ainsi, pour chaque tumeur, nous disposons, d’une part, de gènes impliqués dans la susceptibilité à cette tumeur et, d’autre part, de gènes, le plus souvent distincts des premiers, impliqués dans le développement tumoral lui-même. Une question majeure est donc de comprendre la façon dont ces deux ensembles de gènes interagissent pour aboutir au développement d’un cancer donné chez un individu donné. Certains cancers de l’enfant constituent des modèles intéressants du fait de susceptibilités individuelles assez fortes et du nombre relativement limité d’altérations somatiques. Nous avons récemment conduit une étude sur le sarcome d’Ewing et avons montré comment une mutation somatique est susceptible de coopérer avec un variant génétique constitutif pour déréguler un gène qui contribue au développement de cette tumeur [2]. |
Le sarcome d’Ewing : une tumeur d’origine cellulaire inconnue L’anatomo-pathologiste James Ewing identifia et décrivit, en 1921, cette tumeur éponyme comme un « endothéliome osseux ». L’hypothèse d’une origine endothéliale que sous-entendait cette description n’a cependant pas été confirmée et la cellule à l’origine de ce sarcome reste encore inconnue [3, 4] (→). (→) Voir la Nouvelle de F. Tirode et al., m/s n° 3, mars 2008, page 248 Cliniquement, le sarcome d’Ewing est un cancer agressif de l’enfant et du jeune adulte dont la localisation est le plus souvent osseuse. Au niveau génétique, le sarcome d’Ewing est caractérisé par des translocations chromosomiques spécifiques, exclusivement observées dans les cellules tumorales, qui conduisent à la fusion du gène EWSR1 (Ewing sarcoma breakpoint region 1) à différents facteurs de transcription de la famille ETS (E26 transformation-specific), le plus souvent FLI1 (Friend leukemia virus integration 1). Le gène de fusion résultant, EWSR1-FLI1, code un facteur de transcription aberrant qui contrôle la transcription en se fixant soit au niveau de sites consensus de la famille ETS soit, de façon plus originale, au niveau de motifs microsatellites de répétitions GGAA (guanine-guanine-adénine-adénine) [3]. Dans ce dernier cas, il transforme ces séquences répétitives en véritable amplificateurs transcriptionnels (enhancers) dont l’activité augmente avec le nombre de répétitions GGAA consécutives [5]. Par son action sur ces différents éléments, EWSR1-FLI1 dérégule l’expression de nombreux gènes qui deviennent alors les médiateurs de son activité oncogénique [5, 6]. Des études épidémiologiques ont établi que l’incidence du sarcome d’Ewing est très variable d’une population humaine à l’autre. Ainsi, cette tumeur est environ 10 fois plus fréquente dans les populations d’origine européenne que dans les populations africaine ou afro-américaine. Ce profil épidémiologique très particulier suggère que des polymorphismes génétiques spécifiques jouent un rôle important dans le développement de la maladie [7]. Pour étudier sa susceptibilité génétique, nous avons comparé les fréquences alléliques sur l’ensemble du génome de patients européens atteints de sarcome d’Ewing à celles d’individus témoins, non atteints, de même origine. Ce travail, publié en 2012, montre que trois régions du génome présentent des différences très significatives entre patients et témoins, identifiant ainsi des allèles de susceptibilité [7]. En règle générale, les allèles de susceptibilité identifiés par ces approches à haut débit ne sont pas eux-mêmes impliqués de façon causale dans la maladie, mais ils sont en déséquilibre de liaison avec une ou plusieurs variations génétiques de la même région qui sont, elles, directement impliquées. Pour identifier ces dernières, nous nous sommes concentrés sur la région du chromosome 10, qui contient deux gènes [7] : ADO (2-aminoethanethiol [cysteamine] dioxygenase), qui code une enzyme impliquée dans le métabolisme de la cystéamine [2, 7] et EGR2 (early growth response 2, aussi connu sous le nom de KROX20) [2] qui code un facteur de transcription impliqué dans le développement du système nerveux central et dans la différenciation de plusieurs types cellulaires, incluant les cellules de Schwann et les progéniteurs osseux [2, 7, 8]. Des mutations de EGR2 sont responsables de la maladie de Charcot-Marie-Tooth, une neuropathie périphérique [9]. Bien que la maladie de Charcot-Marie-Tooth et le sarcome d’Ewing soient très rares, quelques cas d’association chez un même patient ont été observés [10], suggérant une possible implication commune de EGR2. |
L’expression d’ EGR2 est sous la dépendance d’EWSR1-FLI1 et de polymorphismes constitutionnels Nous avons observé que le niveau d’expression d’EGR2 (early growth response 2) et, à un moindre degré, celui d’ADO (2-aminoethanethiol [cysteamine] dioxygenase), est très élevé dans les tumeurs d’Ewing en comparaison à des tissus normaux ou d’autres types tumoraux. De plus, une association significative existe entre la présence d’allèles de susceptibilité et un niveau d’expression élevé d’EGR2 et d’ADO, suggérant ainsi que certains allèles de la région soient responsables d’une expression accrue de ces gènes [7]. Cette corrélation est spécifique du sarcome d’Ewing car elle n’est retrouvée ni dans d’autres types de tumeurs, ni dans les tissus normaux. Enfin, nous avons montré que l’inhibition de l’expression d’EWSR1-FLI1 dans différentes lignées cellulaires de sarcome d’Ewing entraîne une baisse d’expression d’EGR2, mais pas d’ADO [2]. L’ensemble de ces données suggère que le niveau d’expression d’EGR2 et d’ADO dépend de polymorphismes constitutionnels, mais que seul EGR2 est régulé par EWSR1-FLI1. |
EGR2 contribue à la croissance des cellules d’Ewing Des expériences in vitro montrent que l’inhibition de l’expression d’EGR2 dans des cellules d’Ewing diminue très fortement leur croissance et leur capacité à former des clones [2]. De plus, l’inhibition d’EGR2 dans des xénogreffes de cellules d’Ewing, in vivo, induit leur régression complète démontrant ainsi un rôle majeur d’EGR2 dans la tumorigénicité de ces cellules [2]. Des études complémentaires sont encore nécessaires pour comprendre la fonction d’EGR2 dans la croissance de cellules d’Ewing mais des résultats préliminaires montrent qu’EGR2 agit en aval de la signalisation par le FGF (fibroblast growth factor) qui est capable d’induire à la fois l’expression d’EGR2 et la prolifération de cellules d’Ewing [2]. |
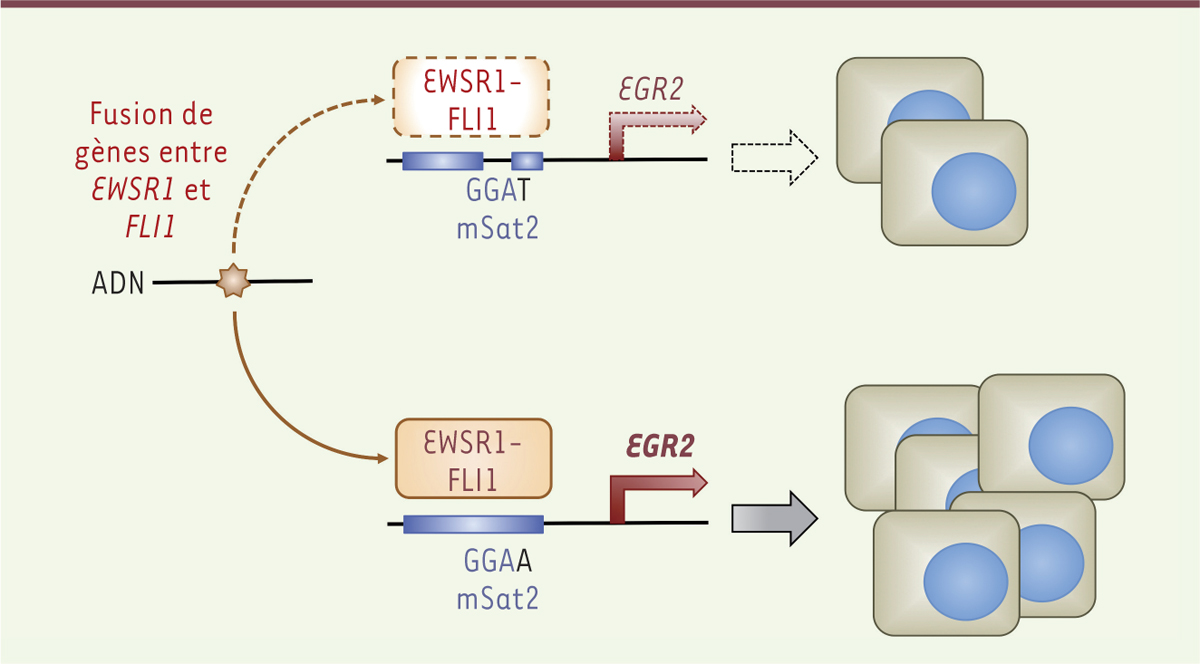
Une approche intégrée pour identifier les variants constitutionnels qui contrôlent EGR2Pour étudier en détail le locus de susceptibilité au niveau d’EGR2 et identifier les variants jouant un rôle direct dans la maladie, nous avons réalisé le séquençage complet de cette région dans l’ADN constitutif de 343 patients atteints de sarcome d’Ewing et 251 témoins. Cette approche nous a permis d’identifier 290 polymorphismes (SNP, single nucleotide polymorphisms) associés de façon significative au sarcome d’Ewing [2]. Sur la base du lien existant entre susceptibilité et niveau d’expression d’EGR2, nous avons restreint notre étude aux SNP associés aux marques épigénétiques liées à une activité transcriptionnelle et/ou à une forte liaison de la protéine EWSR1-FLI1 [6, 11]. Ces marques épigénétiques ne sont observées qu’au niveau de quatre zones dont deux correspondent à des éléments régulateurs connus d’EGR2 (actifs dans le tissu nerveux ou l’os) et deux à des microsatellites de type répétition GGAA (mSat1 et mSat2). Ces deux dernières séquences constituent, par ailleurs, des cibles de fixation de EWSR1-FLI1 [2], ce qui n’est pas le cas des deux autres éléments. L’élément mSat2 présente une forte activité transcriptionnelle amplificatrice (enhancer) et contient un SNP qui transforme l’un des motifs GGAA (allèle A) en GGAT (guanine-guanine-adénine-thymine, allèle T). L’allèle T interrompt ainsi la répétition GGAA, ce qui diminue l’activité amplificatrice par rapport à un mSat2 dont les répétitions GGAA sont ininterrompues. Par ailleurs, dans deux cohortes indépendantes, l’allèle A, a été significativement retrouvé plus fréquemment chez les patients atteints de sarcome d’Ewing que chez les témoins. D’où l’hypothèse que l’expression d’EGR2 est contrôlée par EWSR1-FLI1 via la séquence mSat2, et que l’allèle A, qui permet une répétition ininterrompue de GGAA, induit une expression plus élevée d’EGR2 que l’allèle T. En accord avec cette hypothèse, nous observons que EWSR1-FLI1 se lie, in vivo, plus fortement à l’allèle A qu’à l’allèle T, que le génotype A/A est associé à une expression accrue d’EGR2 par rapport aux génotypes A/T ou T/T et, enfin, qu’il existe un biais d’expression allélique chez les hétérozygotes A/T puisque l’allèle A est plus exprimé que l’allèle T. Enfin, nous montrons que l’allèle T est plus fréquent dans les populations africaines que dans les populations européennes, suggérant que ce polymorphisme pourrait jouer un rôle dans la différence d’incidence du sarcome d’Ewing dans ces différentes populations humaines. Globalement, nos résultats montrent qu’EGR2 est un gène de susceptibilité au sarcome d’Ewing dont la surexpression, au niveau tumoral, est contrôlée au moins en partie par l’interaction fonctionnelle de EWSR1-FLI1 avec une séquence régulatrice GGAA-microsatellite [2] (Figure 1).
 | Figure 1.
La coopération entre la fusion EWSR1-FLI1 et un polymorphisme du microsatellite mSat2 aboutit à une surexpression de EGR2 et à une prolifération accrue dans le sarcome d’Ewing. EGR2 : early growth response 2 ; EWSR1 : Ewing sarcoma breakpoint region 1 ; FLI1 : Friend leukemia virus integration 1 ; GGAA : motif microsatellite de répétition guanosine-guanosine-adénine-adénine ; GGAT : motif microsatellite de répétition guanosine-guanosine-adénine-thymine ; mSat2 : zone GGAA, cible de fixation de EWSR1-FLI1. |
|
Le sarcome d’Ewing comme modèle de coopération entre mutations somatiques et variants constitutionnels en oncogenèse Ce travail constitue l’un des premiers exemples pour lequel la coopération entre une altération somatique (EWSR1-FLI1) et un variant constitutif a pu être élucidée. Cette coopération interfère avec une voie de signalisation majeure (EGR2/FGF) qui contribue à la prolifération cellulaire. D’autres interactions de ce type seront certainement mises en évidence dans le sarcome d’Ewing, soit au niveau de la région EGR2 elle-même, soit au niveau des deux autres régions impliquées dans la susceptibilité à cette tumeur. Il est probable que les études en cours sur d’autres tumeurs, y compris certaines dont la génétique somatique est plus complexe, permettront dans les prochaines années de mieux comprendre comment les altérations génétiques somatiques, à l’origine de chaque tumeur, sont très fortement dépendantes du contexte génétique de l’individu dans lequel elles surviennent. |
Les auteurs déclarent n’avoir aucun conflit d’intérêt concernant les données publiées dans cet article.
|
Thomas G. P. Grünewald est soutenu par des subventions des fondations Daimler and Benz et Reinhard Frank, de la « LMU Munich’s Institutional Strategy LMU excellent within the framework of the German Excellence Initiative », de la Walter Schulz Foundation, de la Fritz Thyssen Foundation (FTF-40.15.0.030MN) et de la German Cancer Aid (DKH-111886). Le groupe Génétique et Biologie des Tumeurs Pédiatriques est labellisé par la Ligue Nationale Contre le Cancer et soutenu par le Cancéropôle Ile-de-France et l’INCa (subvention N° 2014-169). Le groupe Charnay est financé par l’Inserm, le CNRS, le Ministère de la recherche et de la technologie, le Cancéropôle Ile-de-France et l’Institut national du cancer (INCa, subvention N° 2014-169), et la Fondation pour la recherche médicale (FRM). Il a reçu un soutien dans le cadre du programme « Investissements d’Avenir » lancé par le gouvernement français et implémenté par l’Agence nationale de la recherche (ANR, ANR-10-LABX-54 MEMOLIFE et ANR-11-IDEX-0001-02 PSL * Research University).
|
1.
Lawrence MS, Stojanov P, Polak P, et al. Mutational heterogeneity in cancer and the search for new cancer-associated genes . Nature. 2013; ; 499 : :214.–218. 2.
Grünewald TGP, Bernard V, Gilardi-Hebenstreit P, et al. Chimeric EWSR1-FLI1 regulates the Ewing sarcoma susceptibility gene EGR2 via a GGAA microsatellite . Nat Genet. 2015; ; 47 : :1073.–1078. 3.
Lawlor ER, Sorensen PH. Twenty years on : what do we really know about Ewing sarcoma and what is the path forward? Crit Rev Oncog. 2015; ; 20 : :155.–171. 4.
Tirode F, Laud-Duval K, Delattre O. Origine cellulaire des tumeurs d’Ewing : un coin de voile est levé . Med Sci (Paris). 2008; ; 24 : :248.–251. 5.
Guillon N, Tirode F, Boeva V, et al. The oncogenic EWS-FLI1 protein binds in vivo GGAA microsatellite sequences with potential transcriptional activation function . PLoS One. 2009; ; 4 : :e4932.. 6.
Riggi N, Knoechel B, Gillespie SM, et al. EWS-FLI1 utilizes divergent chromatin remodeling mechanisms to directly activate or repress enhancer elements in Ewing sarcoma . Cancer Cell. 2014; ; 26 : :668.–681. 7.
Postel-Vinay S, Véron AS, Tirode F, et al. Common variants near TARDBP and EGR2 are associated with susceptibility to Ewing sarcoma . Nat Genet. 2012; ; 44 : :323.–327. 8.
Topilko P, Schneider-Maunoury S, Levi G, et al. Krox-20 controls myelination in the peripheral nervous system . Nature. 1994; ; 371 : :796.–799. 9.
Decker L, Desmarquet-Trin-Dinh C, Taillebourg E, et al. Peripheral myelin maintenance is a dynamic process requiring constant Krox20 expression . J Neurosci. 2006; ; 26 : :9771.–9779. 10.
Menjak IB, Gupta A, Grinman MN. Delayed diagnosis of metastatic Ewing sarcoma masked by Charcot-Marie-Tooth disease . J Adolesc Young Adult Oncol. 2013; ; 2 : :175.–178. 11.
Maurano MT, Humbert R, Rynes E, et al. Systematic localization of common disease-associated variation in regulatory DNA . Science. 2012; ; 337 : :1190.–1195. |