 |
| |||
Med Sci (Paris). 2015 December; 31(12): 1126–1132. Published online 2015 December 16. doi: 10.1051/medsci/20153112016.Construction de cellules synthétiques Du fondamental au pratique 1Department of Physics, Université du Minnesota, 115 Union Street SE, Minneapolis, 55455Minnesota, États-Unis Corresponding author. | ||||||
| ||||||
L’autoreproduction des cellules vivantes est une propriété unique de la matière. Ce mécanisme est très compliqué à comprendre car une cellule naît d’une autre cellule, comme l’a observé R. Virchow en 1859 : « omnis cellula e cellula » [1]. À ce jour, aucune cellule rudimentaire n’a été découverte qui puisse nous renseigner sur les mécanismes primitifs de la réplication de cellules programmées par l’ADN. Une façon de comprendre la logique de l’autoreproduction est d’essayer de construire une cellule synthétique à partir des molécules ou des machineries moléculaires de cellules simples comme les bactéries. Ce type de projet très ambitieux est devenu techniquement concevable ces deux dernières décennies avec, en particulier, les progrès de la biologie moléculaire (synthèse et séquençage de l’ADN) et de la physique de la matière molle (physique des amphiphiles1, mécanique de la cellule). Il est aujourd’hui envisageable d’assembler des molécules biologiques avec l’objectif de fabriquer une vésicule lipidique capable de se diviser et de grossir dans le temps [2, 3]. Ce domaine de recherche doit fournir des informations sur la logique de l’autoreproduction tant sur le point biologique que physique. C’est aussi une aire de recherche multidisciplinaire propice au développement de nouvelles technologies, en particulier pour les biotechnologies et la médecine. La construction de cellules synthétiques, par exemple, peut aider à la mise au point de nouveaux senseurs membranaires et servir de véhicule pour l’administration de médicaments. Plus généralement, la synthèse de processus biochimiques in vitro est une méthode utile à la compréhension des systèmes complexes codés par des réseaux biologiques. Ce type d’approche permet de saisir, par construction, le lien coopératif entre les machines moléculaires formant les cellules vivantes, et de caractériser le répertoire et les réseaux biochimiques trouvés en biologie. L’objectif est aussi de développer une connaissance quantitative du vivant et d’étendre les propriétés des systèmes naturels. La construction de réseaux biochimiques acellulaires à partir de protéines, d’ADN et d’autres cofacteurs biochimiques est un sujet de plus en plus populaire [4]. Dans cette ligne de recherche, l’un des projets les plus ambitieux est la fabrication d’une cellule à partir de molécules. On peut dégager trois démarches expérimentales pour assembler des cellules synthétiques en partant des composantes moléculaires, et ce malgré un vocabulaire souvent confus : cellule artificielle, cellule minimale, protocellule, semi-cellule, cellule réduite, coacervat (gouttelette contenant un colloïde riche en composés organiques, et entourée d’une membrane de molécules amphiphiles), cellular imitations, cellule partielle et autre jargons font ainsi partie des dénominations rencontrées [5–12]. L’approche protocellule explore l’origine de la vie à travers la construction de cellules faites de molécules prébiotiques. Les protocellules les plus simples ne contiennent pas d’information génétique et sont seulement fondées sur le métabolisme et l’auto-assemblage [13]. La membrane est composée d’acides gras qui, par exemple, ont été retrouvés dans des fragments de météorite [14]. Les protocellules plus sophistiquées, dépourvues de machines moléculaires complexes, incorporent des peptides ou de l’ARN comme information. L’objectif est d’établir des scénarios d’émergence de la vie cellulaire dans des conditions prébiotiques, de la formation de compartiments microscopiques à leur croissance et reproduction [15, 16]. La cellule artificielle consiste à mettre ensemble des molécules naturelles et synthétiques pour concevoir des compartiments microscopiques programmés pour des fonctions spécifiques. En utilisant des éléments artificiels, cette approche a aussi pour objectif d’étendre les propriétés des systèmes naturels [17]. Par exemple, la membrane des polymersomes (compartiment de taille cellulaire dont la membrane est faite de copolymères à blocs2) est beaucoup plus robuste que les bicouches lipidiques naturelles [18–21]. Ces compartiments sont utilisés comme transporteurs de molécules et servent aussi de microréacteurs. De nombreuses applications de ces polymersomes sont développées, notamment pour l’administration de médicaments [22]. Enfin, et c’est l’objet de cette revue, la construction de cellules dites minimales cherche à faire avancer notre connaissance de l’autoreproduction cellulaire par l’assemblage de compartiments microscopiques composés de molécules naturelles, le tout codé par un génome ADN le plus petit possible pour la reproduction du compartiment [11, 12]. L’approche par le bas cherche à révéler les liens coopératifs entre les trois blocs moléculaires nécessaires à l’autoreproduction [13] : l’information ADN, le métabolisme et l’auto-organisation (comme la membrane et le cytosquelette). Il va de soi qu’une cellule minimale est un système fragile construit en laboratoire dans des conditions idéales. Il est important de mentionner l’approche par le haut qui consiste à réduire le génome d’un organisme au minimum tout en restant autoreproductif. Dans ce domaine, c’est au Craig Venter Institute que l’on doit des avancées majeures. Cet institut a montré que le génome de la bactérie Mycoplasma genitalium, l’un des plus petits génomes connus, pouvait être entièrement reconstruit in vitro en utilisant des méthodes de clonage nouvelles, et ensuite être utilisé pour reformer une cellule capable de se reproduire [23, 24]. | ||||||
Comment la théorie peut-elle aider à construire une cellule minimale capable de se reproduire ? Devant la complexité des cellules vivantes, même des plus petites bactéries, on peut se demander comment procéder pour en construire une à partir des molécules prises séparément. Chose étonnante, la logique de l’autoreproduction des automates développée par Von Neumann aide à simplifier la vision du fonctionnement d’une cellule vivante [25]. Von Neumann a été le premier à imaginer qu’un automate capable de se reproduire, doit posséder une bande linéaire contenant les informations, autrement dit l’ADN génomique. Les trois autres parties de l’automate ont aussi des équivalents bien spécifiques dans les cellules vivantes : la transcription-traduction (le constructeur universel), la réplication de l’ADN (le copieur) et la régulation de ces fonctions (le contrôleur). Ce qui est remarquable, c’est que ce modèle théorique ait été développé avant la découverte de l’ADN et des autres machines moléculaires [26] (→). (→) Voir l’article d’Antoine Danchin, m/s n° 5, mai 2008, page 533 Bien qu’utile pour comprendre la logique de l’information trouvée dans les cellules vivantes, l’architecture des automates autoreproductifs ne saisit pas entièrement l’essence de la vie unicellulaire, loin de là. Une cellule ne se résume pas à un programme ADN. La construction de cellules synthétiques « par le bas » est confrontée à des questions fondamentales sur les processus non génétiques. Par exemple, d’un point de vue physique, l’assemblage de protéines en structures actives, l’entropie à travers l’encombrement moléculaire [27], les brisures de symétrie à des échelles microscopiques [28] et les différentes phases liquide-solide que les bicouches lipidiques peuvent adopter [29] sont des mécanismes essentiels aux cellules vivantes que la construction de cellules peut éclairer. De même, les questions et contraintes liées au métabolisme ne sont pas abordées dans le modèle de Von Neumann. Un apport continu de composants de base (nucléotides, acides aminés) est nécessaire aux fonctions les plus basiques des cellules, même avant de récapituler l’autoreproduction. À l’inverse, l’élimination de produits moléculaires dérivés de ce métabolisme est toute aussi importante. L’accumulation de produits issus de l’hydrolyse de l’ATP, par exemple, inhibe rapidement l’activité des enzymes utilisant cette source d’énergie chimique par le principe de charge énergétique en adénylates, définie comme l’énergie métabolique potentielle stockée par un organisme, ou en d’autres termes, comme la charge d’une batterie ([ATP + 0,5 ADP] / ATP) + ADP + AMP [30]. L’activité des enzymes qui utilisent l’ATP dépend exponentiellement de la charge énergétique. En présence d’ATP seulement, une réaction biochimique est chargée à 100 % (la charge énergétique est égale à 1). En l’absence d’ATP, la charge énergétique est zéro. La construction d’une cellule synthétique capable de se reproduire n’a pas encore été accomplie, mais c’est un problème difficile et stimulant dès les premières étapes qui nécessite aussi le développement d’un milieu extérieur adapté (nutriments, balance de la pression osmotique, etc.). | ||||||
La construction d’une cellule minimale consiste à incorporer, dans une vésicule lipidique, les machines moléculaires nécessaires à l’expression d’un génome ADN réduit, qui code pour les protéines nécessaires aux fonctions essentielles à l’autoreproduction de la cellule synthétique. Dans les premières étapes de construction, les substances nutritives de base (acides aminés, nucléotides) sont ajoutées directement dans la solution. L’approche adoptée par la plupart des groupes travaillant sur ce sujet consiste à intégrer trois blocs moléculaires (Figure 1) correspondant aux trois processus fondamentaux : l’information ADN (génome minimal), le métabolisme (substances nutritives de base, régénération de l’énergie chimique sous forme d’ATP et de GTP) et la compartimentation (membrane lipidique et auto-assemblage de structures actives nécessaires à l’autoreproduction) [3, 13]. Pris séparément, ces trois ensembles ont déjà fait l’objet de progrès importants tant sur le plan technique que dans la compréhension de leurs mécanismes. C’est l’intégration et la connexion de ces trois blocs pour construire un compartiment stable physiquement – la cellule minimale – et capable de se reproduire, qui représentent les obstacles principaux [31].
La compartimentation La limite physique du compartiment est une membrane composée de phospholipides. La physique de la matière molle et la recherche médicale ont fourni de nombreuses méthodes pour préparer des liposomes de taille cellulaire (1-10 µm de diamètre) [32]. Ces méthodes fonctionnent bien pour des solutions aqueuses diluées. La fabrication de liposomes stables contenant les machines moléculaires pour l’expression de gènes est beaucoup plus compliquée. En effet, ce processus requiert des réactions qui font intervenir des sels en concentrations physiologiques (100-200 mM) et des macromolécules (protéines, ribosomes, ADN) en concentrations élevées qui réduisent considérablement la stabilité et la production des liposomes. C’est un premier obstacle. Le second obstacle est le développement d’une membrane fonctionnelle contenant des protéines membranaires capables, par exemple, de filtrer les substances de base nécessaires à l’expression de gènes et d’équilibrer la pression osmotique. L’objectif est que ces fonctions soient « codées » dans un génome minimal. L’expression de protéines membranaires in vitro est encore limitée malgré des avancées récentes [33, 34]. Enfin, la division physique de la cellule minimale repose sur l’assemblage d’un cytosquelette actif à la membrane. Cette étape se heurte à la difficulté de reproduire, in vitro, l’encombrement moléculaire qui, par effet de volume, exclut, favorise ou accélère l’assemblage de telles structures dans les cellules vivantes [27]. À cela s’ajoute un problème de brisure de symétrie puisque les liposomes sont sphériques et que la symétrie devra être rompue pour que la division cellulaire s’accomplisse. Des instabilités doivent être produites à la membrane (par réduction de la tension pour obtenir une membrane souple) afin de rendre asymétrique l’assemblage du cytosquelette responsable de la division.Le métabolisme
In vivo, la synthèse des substances nutritives de base et le recyclage des produits dérivés sont accomplis par des voies métaboliques complexes, souvent couplées. Dans les premières étapes de la construction d’une cellule minimale, ces substrats de base sont directement ajoutés au milieu réactionnel encapsulé dans les liposomes, ainsi que dans la solution extérieure. La plus grande partie de l’énergie chimique (ATP, GTP) est consommée dans la traduction de protéines. Chaque acide aminé polymérisé nécessite deux ATP et deux GTP, ce qui est énorme. Par le principe de charge adénylate, correspondant au vrai paramètre d’énergie ATP, il est connu que le recyclage de l’ADP et AMP (GDP et GMP) est aussi important que le maintien de la concentration en ATP. Enfin, la dégradation des messagers et des protéines produites est essentielle pour le dynamisme de l’information génétique. La mise en œuvre et l’ajustement de l’inactivation et de la dégradation des messagers et des protéines in vitro est indispensable, ce qui est une tâche encore difficile à réaliser [35].Le génome minimal La plupart des difficultés mentionnées pour la membrane et le métabolisme peuvent être résolues en construisant un génome ADN minimal : celui-ci a pour objectif d’assurer la robustesse mécanique en codant pour une enveloppe cellulaire, de créer les voies essentielles du métabolisme en codant des enzymes capables d’exploiter des ressources de base (source carbone), de permettre la réplication de l’ADN et la synthèse de phospholipides, etc. Différents travaux ont estimé à entre 200 et 400 gènes la taille du génome minimal capable d’assurer l’autoreproduction d’un liposome [36–40]. Techniquement, un tel génome peut être assemblé, puisque la construction moléculaire de génomes synthétiques est possible [23]. Mais deux difficultés principales émergent : la conception de la régulation nécessaire au fonctionnement d’un tel programme ADN et son exécution initiale par des machines moléculaires. Faire fonctionner in vivo des circuits génétiques élémentaires synthétiques construits en laboratoire, même s’ils sont composés de séquences codantes bien caractérisées, est encore aujourd’hui un obstacle sérieux, quand bien même des progrès ont été réalisés récemment [41]. Le problème réside aussi dans le couplage de l’information digitale portée par l’ADN et de la dynamique des processus liés à l’autoreproduction qui sont essentiellement analogiques. Il n’est pas clair qu’en procédant comme des ingénieurs, nous puissions construire un génome synthétique codant pour l’autoreproduction. Une alternative est de créer un génome présentant assez de variabilité pour être modifié par l’évolution de façon à obtenir un génome stable codant pour l’autoreproduction. Pour cela, la création de cycles d’évolution en laboratoire, en réalisant des fusions-fissions sur des liposomes synthétiques, semble être un point de passage nécessaire, de même que la réplication de l’ADN avec un taux de mutations ajustable. Les techniques microfluidiques semblent être bien adaptées pour ce type d’expérience [42]. | ||||||
Les systèmes acellulaires d’expression comme plateforme expérimentale pour la construction de cellules minimales Les systèmes de production de protéines in vitro sont devenus récemment des plates-formes expérimentales utiles au développement de la biologie synthétique [43]. À l’origine, ces systèmes ont été développés pour déchiffrer le code génétique [44]. Dans les années 1980 et 1990, l’utilisation de kits de production de protéines in vitro s’est généralisée pour l’expression simple de protéines hors cellule. Au cours de ces dix dernières années, cette technologie est devenue une plateforme expérimentale pour la construction de systèmes biochimiques in vitro à des échelles très variées : de l’évolution de protéines à la construction de réseaux génétiques [4]. Ces systèmes sont aussi particulièrement bien adaptés à la construction de cellules minimales puisqu’ils contiennent les machines moléculaires nécessaires à la transcription et la traduction. Les kits commerciaux permettent de produire 1 mg/ml de protéines dans un tube à essai en quelques heures, ce qui correspond à une concentration de 30 µm de protéine comme l’eGFP (enhanced green fluorescent protein). La concentration moyenne de protéines cytoplasmiques chez E. coli est d’environ 0,5 µm [45]. Par conséquent, une cinquantaine de gènes peuvent être exprimés avec ces kits à des concentrations pertinentes biologiquement. La synthèse récente de phages fonctionnels comme T7 (40 kbp, 60 gènes) est une démonstration de ce niveau d’expression. Des auto-assemblages biologiques complexes programmés par l’ADN peuvent désormais être réalisés dans des tubes à essais (Figure 2A, B) [46]. Au regard de ce résultat, la construction de réseaux génétiques synthétiques in vitro reste limitée mais elle progresse rapidement (Figure 2C, D) [47, 48].
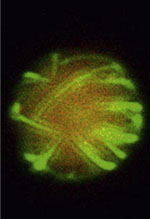
Le développement de systèmes d’expression encore plus puissants est nécessaire à la construction de cellules minimales. De tels travaux sont bénéfiques pour de nouvelles applications in vitro comme la production de molécules thérapeutiques telles que des vaccins [49, 50]. L’une des clés du développement de ce type de recherche réside dans la création de nouvelles voies métaboliques afin de stimuler et accroître l’expression in vitro. Les systèmes les plus récents utilisent des sources carbone (glucose, maltose) pour assurer la régénération de l’ATP, ce qui permet d’atteindre un taux de 2 mg/ml de protéines produites [51]. L’expression acellulaire de gènes dans des liposomes a été réalisée mais ces expériences ne concernent que quelques gènes rapporteurs. L’encapsulation de ces réactions complexes est compliquée. À cela s’ajoute l’hétérogénéité de l’encapsulation, et donc de l’expression, d’un liposome à l’autre, quelle que soit la méthode utilisée [52, 53]. Au-delà de l’encapsulation, la construction d’une membrane active est un défi. Des progrès importants ont été réalisés dans l’expression acellulaire de protéines membranaires, une aubaine pour la construction de cellules minimales [54]. Cependant, la création d’une membrane vraiment fonctionnelle, capable de filtrer les substances de base et de répondre à des contraintes physiques ou biochimiques, n’est pas encore une réalité. Des étapes importantes ont déjà été franchies, comme l’insertion de canaux dans les membranes et la polymérisation d’un cytosquelette (Figure 3) [55–57].
La réalisation de fonctions plus complexes comme la division ou la réplication de l’ADN semble encore lointaine. L’utilisation de compositions phospholipidiques plus variées que la seule phosphatidylcholine (PC) pour la fabrication de liposomes, doit mettre en lumière l’importance de la membrane dans la formation de phases lipidiques essentielles pour l’auto-organisation et la brisure de symétrie à des échelles de quelques micromètres. Le travail fait sur les liposomes et l’expression acellulaire favorise le développement de méthodes utiles pour les biotechnologies comme par exemple le liposome display (affichage sur liposome) qui permet de suivre l’évolution et la sélection de protéines membranaires [58]. | ||||||
La synthèse « par le bas » d’une cellule minimale est encore un objectif lointain mais des avancées significatives sont faites en ce qui concerne les trois composantes moléculaires qui forment ce puzzle complexe. Ce domaine de recherche favorise le développement d’approches quantitatives et de plates-formes expérimentales pour la biologie acellulaire. La construction d’une cellule minimale à partir de molécules est aussi un terrain de jeu formidable pour la recherche multidisciplinaire, de la physique molle à l’ingénierie métabolique, et de la génomique à la théorie des automates cellulaires. De nouvelles méthodes comme l’apprentissage par la machine, le haut débit et la microfluidique semblent nécessaires pour accélérer la construction et la compréhension de systèmes biochimiques complexes in vitro qui bénéficieront aux biotechnologies et à la médecine. | ||||||
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article. | ||||||
Je remercie Albert Libchaber pour ces commentaires sur le manuscrit et nos discussions sur les cellules synthétiques. Je remercie Marie-France Mercier, Claude Noireaux et Danielle Coustillet pour leur travail minutieux de relecture de l’article. | ||||||
1.
Virchow RLK. Die cellularpathologie in ihrer begründung auf physiologische und pathologische gewebelehre . Berlin: : A. Hirschwald; , 1858;; xvi : :440. p. 3.
Noireaux V, Maeda YT, Libchaber A. Development of an artificial cell, from self-organization to computation and self-reproduction . Proc Natl Acad Sci USA. 2011; ; 108 : :3473.–3480. 4.
Hockenberry AJ, Jewett MC. Synthetic in vitro circuits . Curr Opin Chem Biol. 2012; ; 16 : :253.–259. 5.
Caschera F, Stano P, Luisi PL. Reactivity and fusion between cationic vesicles and fatty acid anionic vesicles . J Colloid Interface Sci. 2010; ; 345 : :561.–565. 6.
Sokolova E, Spruijt E, Hansen MMK, et al. Enhanced transcription rates in membrane-free protocells formed by coacervation of cell lysate . Proc Natl Acad Sci USA. 2013; ; 110 : :11692.–11697. 7.
Mann S. Systems of creation: the emergence of life from nonliving matter . Accounts Chem Res. 2012; ; 45 : :2131.–2141. 8.
Forlin M, Lentini R, Mansy SS. Cellular imitations . Curr Opin Chem Biol. 2012; ; 16 : :586.–592. 9.
Ichihashi N, Matsuura T, Kita H, et al. Constructing partial models of cells . Cold spring Harb Perspect Biol. 2010; ; 2 : :a004945.. 10.
Loakes D, Holliger P. Darwinian chemistry: towards the synthesis of a simple cell . Mol Biosyst. 2009; ; 5 : :686.–694. 12.
Jewett MC, Forster AC. Update on designing and building minimal cells . Curr Opin Biotech. 2010; ; 21 : :697.–703. 13.
Sole RV. Evolution and self-assembly of protocells . Int J Biochem Cell Biol. 2009; ; 41 : :274.–284. 14.
Lawless JG, Yuen GU. Quantification of monocarboxylic acids in the Murchison carbonaceous meteorite . Nature. 1979; ; 282 : :396.–398. 15.
Mansy SS, Szostak JW. Reconstructing the emergence of cellular life through the synthesis of model protocells . Cold Spring Harb Symp Quant Biol. 2009; ; 74 : :47.–54. 16.
Hanczyc MM, Fujikawa SM, Szostak JW. Experimental models of primitive cellular compartments: encapsulation, growth, and division . Science. 2003; ; 302 : :618.–622. 17.
Rasmussen S, Chen LH, Nilsson M, Abe S. Bridging nonliving and living matter . Artif Life. 2003; ; 9 : :269.–316. 19.
Kamat NP, Katz JS, Hammer DA. Engineering polymersome protocells . J Phys Chem Lett. 2011; ; 2 : :1612.–1623. 20.
Marguet M, Bonduelle C, Lecommandoux S. Multicompartmentalized polymeric systems: towards biomimetic cellular structure and function . Chem Soc Rev. 2013; ; 42 : :512.–529. 21.
Peters RJ, Marguet M, Marais S, et al. Cascade reactions in multicompartmentalized polymersomes . Angew Chem Int Ed Engl. 2014; ; 53 : :146.–150. 22.
Lee JS, Feijen J. Polymersomes for drug delivery: design, formation and characterization . J Control Release. 2012; ; 161 : :473.–483. 23.
Gibson DG, Benders GA, Andrews-Pfannkoch C, et al. Complete chemical synthesis, assembly, and cloning of a Mycoplasma genitalium genome . Science. 2008; ; 319 : :1215.–1220. 24.
Gibson DG, Glass JI, Lartigue C, et al. Creation of a bacterial cell controlled by a chemically synthesized genome . Science. 2010; ; 329 : :52.–56. 25.
Von Neumann J. The general and logical theory of automata . In : Jeffress LA, ed. Cerebral mechanisms in behavior: the Hixon symposium . New York: : Wiley; , 1951. 26.
Danchin A. Saurons-nous construire une bactérie synthétique ? Med Sci (Paris). 2008; ; 24 : :533.–540. 27.
Minton AP. How can biochemical reactions within cells differ from those in test tubes? J Cell Sci. 2006; ; 119 : :2863.–2869. 28.
Li R, Bowerman B. Symmetry breaking in biology . Cold Spring Harb Perspect Biol. 2010; ; 2 : :a003475.. 29.
Baumgart T, Hess ST, Webb WW. Imaging coexisting fluid domains in biomembrane models coupling curvature and line tension . Nature. 2003; ; 425 : :821.–824. 30.
Atkinson DE. The energy charge of the adenylate pool as a regulatory parameter . Interaction with feedback modifiers. Biochemistry. 1968; ; 7 : :4030.–4034. 31.
Caschera F, Noireaux V. Integration of biological parts toward the synthesis of a minimal cell . Curr Opin Chem Biol. 2014; ; 22 : :85.–91. 32.
Walde P, Cosentino K, Engel H, Stano P. Giant vesicles: preparations and applications . Chembiochem. 2010; ; 11 : :848.–865. 33.
Soga H, Fujii S, Yomo T, et al. In vitro membrane protein synthesis inside cell-sized vesicles reveals the dependence of membrane protein integration on vesicle volume . ACS Synth Biol. 2014; ; 3 : :372.–379. 34.
Matsubayashi H, Kuruma Y, Ueda T. In vitro synthesis of the E. coli Sec translocon from DNA . Angew Chem Int Ed Engl. 2014; ; 53 : :7535.–7538. 35.
Shin J, Noireaux V. Study of messenger RNA inactivation and protein degradation in an Escherichia coli cell-free expression system . J Biol Eng. 2010; ; 4 : :9.. 36.
Koonin EV, Mushegian AR. Complete genome sequences of cellular life forms: glimpses of theoretical evolutionary genomics . Curr Opin Genet Dev. 1996; ; 6 : :757.–762. 37.
Kolisnychenko V, Plunkett G 3rd. , Herring CD, et al. Engineering a reduced Escherichia coli genome . Genome Res. (3rd) 2002; ; 12 : :640.–647. 38.
Gil R, Silva FJ, Pereto J, Moya A. Determination of the core of a minimal bacterial gene set . Microbiol Mol Biol Rev. 2004; ; 68 : :518.–537. 39.
Glass JI, Assad-Garcia N, Alperovich N, et al. Essential genes of a minimal bacterium . Proc Natl Acad Sci USA. 2006; ; 103 : :425.–430. 41.
Smanski MJ, Bhatia S, Zhao DH, et al. Functional optimization of gene clusters by combinatorial design and assembly . Nat Biotechnol. 2014; ; 32 : :1241.–1249. 42.
Fallah-Araghi A, Baret JC, Ryckelynck M, Griffiths AD. A completely in vitro ultrahigh-throughput droplet-based microfluidic screening system for protein engineering and directed evolution . Lab Chip. 2012; ; 12 : :882.–891. 43.
Hodgman CE, Jewett MC. Cell-free synthetic biology: thinking outside the cell . Metab Eng. 2012; ; 14 : :261.–269. 44.
Nirenberg M. Historical review: deciphering the genetic code: a personal account . Trends Biochem Sci. 2004; ; 29 : :46.–54. 45.
Ishihama Y, Schmidt T, Rappsilber J, et al. Protein abundance profiling of the Escherichia coli cytosol . BMC Genomics. 2008; ; 9 : :102.. 46.
Shin J, Jardine P, Noireaux V. Genome replication, synthesis, and assembly of the bacteriophage T7 in a single cell-free reaction . ACS Synth Biol. 2012; ; 1 : :408.–413. 47.
Shin J, Noireaux V. An E. coli cell-free expression toolbox: application to synthetic gene circuits and artificial cells. ACS Synth . Biol. 2012; ; 1 : :29.–41. 48.
Lapique N, Benenson Y. Vue de l’intérieur : des circuits génétiques pour l’analyse de profils moléculaires intracellulaires . Med Sci (Paris). 2015; ; 31 : :487.–491. 49.
Carlson ED, Gan R, Hodgman CE, Jewett MC. Cell-free protein synthesis: applications come of age . Biotechnol Adv. 2012; ; 30 : :1185.–1194. 50.
Ng PP, Jia M, Patel KG, et al. A vaccine directed to B cells and produced by cell-free protein synthesis generates potent antilymphoma immunity . Proc Natl Acad Sci USA. 2012; ; 109 : :14526.–14531. 51.
Caschera F, Noireaux V. Synthesis of 2,3 mg/ml of protein with an all Escherichia coli cell-free transcription-translation system . Biochimie. 2014; ; 99 : :162.–168. 52.
Nourian Z, Danelon C. Linking genotype and phenotype in protein synthesizing liposomes with external supply of resources . ACS Synth Biol. 2013; ; 2 : :186.–193. 53.
Hosoda K, Sunami T, Kazuta Y, et al. Quantitative study of the structure of multilamellar giant liposomes as a container of protein synthesis reaction . Langmuir. 2008; ; 24 : :13540.–13548. 54.
Katzen F, Peterson TC, Kudlicki W. Membrane protein expression: no cells required . Trends Biotechnol. 2009; ; 27 : :455.–460. 55.
Noireaux V, Libchaber A. A vesicle bioreactor as a step toward an artificial cell assembly . Proc Natl Acad Sci USA. 2004; ; 101 : :17669.–17674. 56.
Maeda YT, Nakadai T, Shin J, et al. Assembly of MreB filaments on liposome membranes: a synthetic biology approach . ACS Synth Biol. 2012; ; 1 : :53.–59. | ||||||