| |
| Med Sci (Paris). 2015 June; 31(6-7): 594–597. Published online 2015 July 7. doi: 10.1051/medsci/20153106007.NDP52, autophagie et pathogènes « Et le combat cessa faute de combattants » Pauline Verlhac,1,2,3,4,5 Christophe Viret,1,2,3,4,5 and Mathias Faure1,2,3,4,5* 1CIRI, centre international de recherche en infectiologie, université de Lyon, 69007Lyon, France 2Inserm U1111, 69007Lyon, France 3CNRS, UMR5308, 69007Lyon, France 4École normale supérieure de Lyon, 69007Lyon, France 5Université Lyon 1, Centre international de recherche en infectiologie, UCBL1, 21, avenue Tony Garnier, 69365Lyon Cedex 07, France MeSH keywords: Animaux, Autophagie, Bactéries, Infections bactériennes, Humains, Immunité innée, Protéines nucléaires, Phagosomes, génétique, immunologie, pathogénicité, physiologie, microbiologie |
Afin de lutter contre les microorganismes envahissant leur cytoplasme, les cellules sont dotées d’une arme de destruction sélective, l’autophagie. En effet, utilisée par la cellule pour éliminer et recycler ses propres constituants défectueux ou en fin d’activité biologique, l’autophagie cible également des pathogènes intracellulaires afin de les dégrader ; l’autophagie est ainsi indispensable non seulement pour éviter le développement de maladies infectieuses, mais également pour maintenir l’équilibre avec une flore commensale essentielle au développement et au fonctionnement de l’organisme. La compréhension moléculaire du contrôle des infections par l’autophagie progresse chaque jour, notamment avec l’identification de la protéine NDP52 (nuclear dot protein 52 kDa) qui contribue doublement à la lutte anti-microbienne : en acheminant sélectivement les bactéries vers l’autophagie et en promouvant leur destruction une fois piégées dans les autophagosomes.
Dans les années 1960, Christian de Duve observe que des composants propres à la cellule sont capturés dans des vésicules à double membrane pour y être à terme dégradés [1]. Il nomme ces vésicules autophagosomes et le processus autophagie (littéralement « se manger »). La dégradation des composants emprisonnés dans l’autophagosome est assurée lorsque celui-ci fusionne avec une autre vésicule spécialisée, un lysosome (organite également découvert par C. de Duve en 1955). Plusieurs travaux décrivent par la suite l’importance de l’autophagie pour l’homéostasie cellulaire, et, dans les années 1980, il est observé la formation d’autophagosomes lors de l’infection cellulaire par un micro-organisme, la bactérie Rickettsia [2]. La destruction par autophagie d’agents infectieux, ou xénophagie (« manger ce qui est étranger ») [3, 4], ne sera formellement démontrée qu’en 2004, à l’encontre de Mycobacterium tuberculosis et du streptocoque de groupe A, grâce à l’avènement d’outils moléculaires suffisamment précis pour l’étude fine de l’autophagie [5, 6]. |
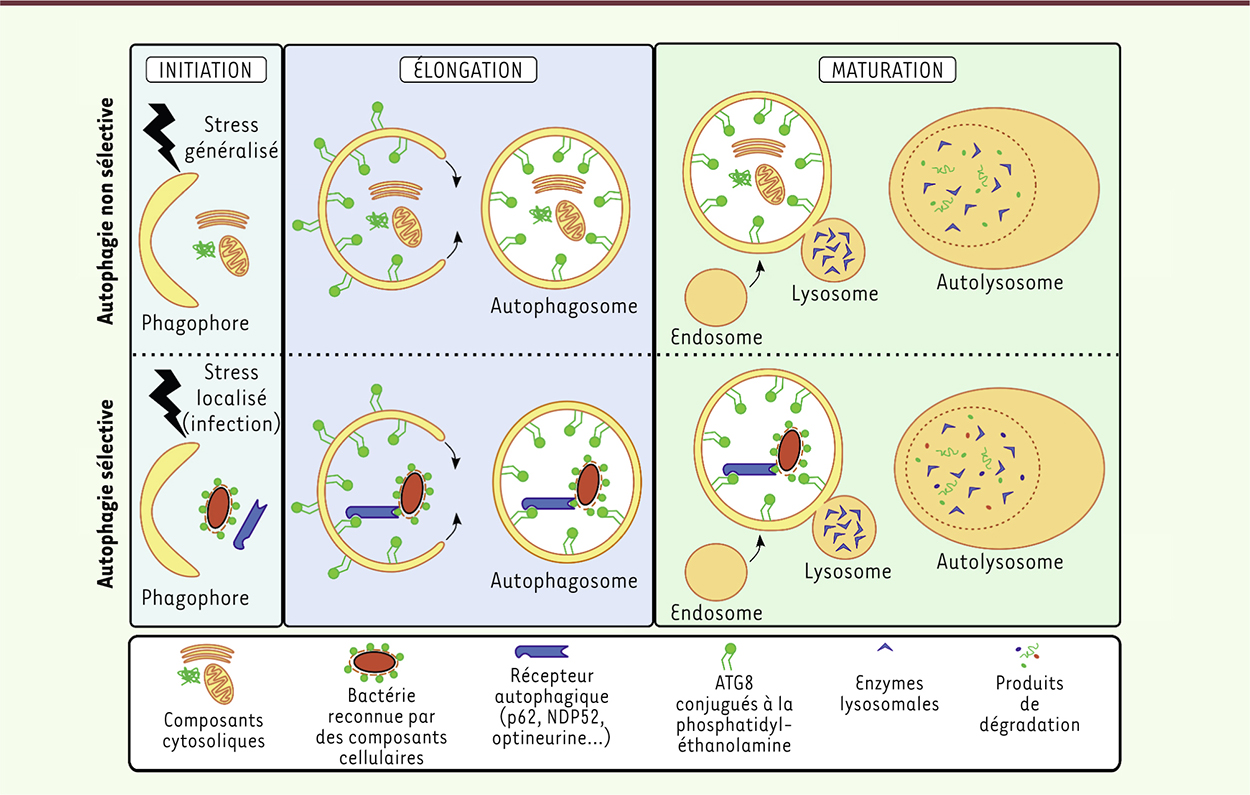
Les étapes du processus de macroautophagie : du phagophore à l’autolysosome La macroautophagie, appelée communément autophagie, s’articule en trois phases majeures [3]. La première étape génère une membrane incurvée, le phagophore. Les extrémités de ce dernier s’étendent ensuite par l’ajout de petites vésicules membranaires. Lors de cette étape d’élongation, la vésicule autophagique en formation va attraper des composants cytosoliques, appelés cargos ou substrats, destinés au recyclage (Figure 1). Dans les conditions d’un stress ressenti de manière généralisée par la cellule, comme une absence de nutriments dans l’environnement, les cargos peuvent être séquestrés de manière aléatoire par l’autophagie. Cependant, lors de stress localisés au sein de la cellule, comme celui que représente la présence d’agents infectieux, les substrats autophagiques sont sélectivement séquestrés. Cette autophagie sélective est assurée par des protéines cytosoliques appelées récepteurs autophagiques [7]. Ces derniers reconnaissent, d’une part, l’élément destiné à la dégradation, et, d’autre part, un membre de la famille des protéines ATG8 (grâce à un domaine appelé LIR, LC3-interacting region). Ces facteurs, qui incluent les protéines LC3 (light chain 3) et GABARAP (GABA-A receptor-associated protein), sont enchâssés dans la membrane du phagophore en croissance. Les récepteurs autophagiques servent donc de pont moléculaire entre le substrat et la vésicule autophagique en formation. La phase d’élongation s’achève par la fusion des deux extrémités du phagophore, structurant un autophagosome. Celui-ci isole ainsi son substrat du reste de la cellule, mais la lumière autophagique n’étant ni acide, ni riche en enzymes actives, l’autophagosome per se ne peut dégrader son contenu. Afin d’assurer cette fonction, le processus autophagique se poursuit (on parle de flux autophagique) par la fusion de l’autophagosome avec une vésicule de la voie endolysosomale. Cette étape de maturation mène à la formation d’un autolysosome (fusion autophagosome-lysosome), qui peut être précédée par la formation d’un amphisome (fusion autophagosome-endosome) ; la maturation est indispensable car elle seule permet de dégrader ce qu’emprisonne la vésicule autophagique (Figure 1).
 | Figure 1.
Séquestration et dégradation de composants cytoplasmiques par l’autophagie. Au-delà de son rôle d’élimination de composants obsolètes de la cellule, l’autophagie peut être induite en réponse à de nombreux stress perçus par la cellule. Trois étapes majeures orchestrent alors le flux autophagique : l’initiation, l’élongation et la maturation. Durant l’élongation, les cargos/substrats sont séquestrés dans l’autophagosome en formation : ces composants peuvent être séquestrés de manière aléatoire ou sélective (grâce à des récepteurs autophagiques), probablement selon l’origine, la nature et/ou l’intensité du stress. Durant la maturation, la fusion de l’autophagosome avec un lysosome peut être précédée d’une fusion avec un endosome. |
|
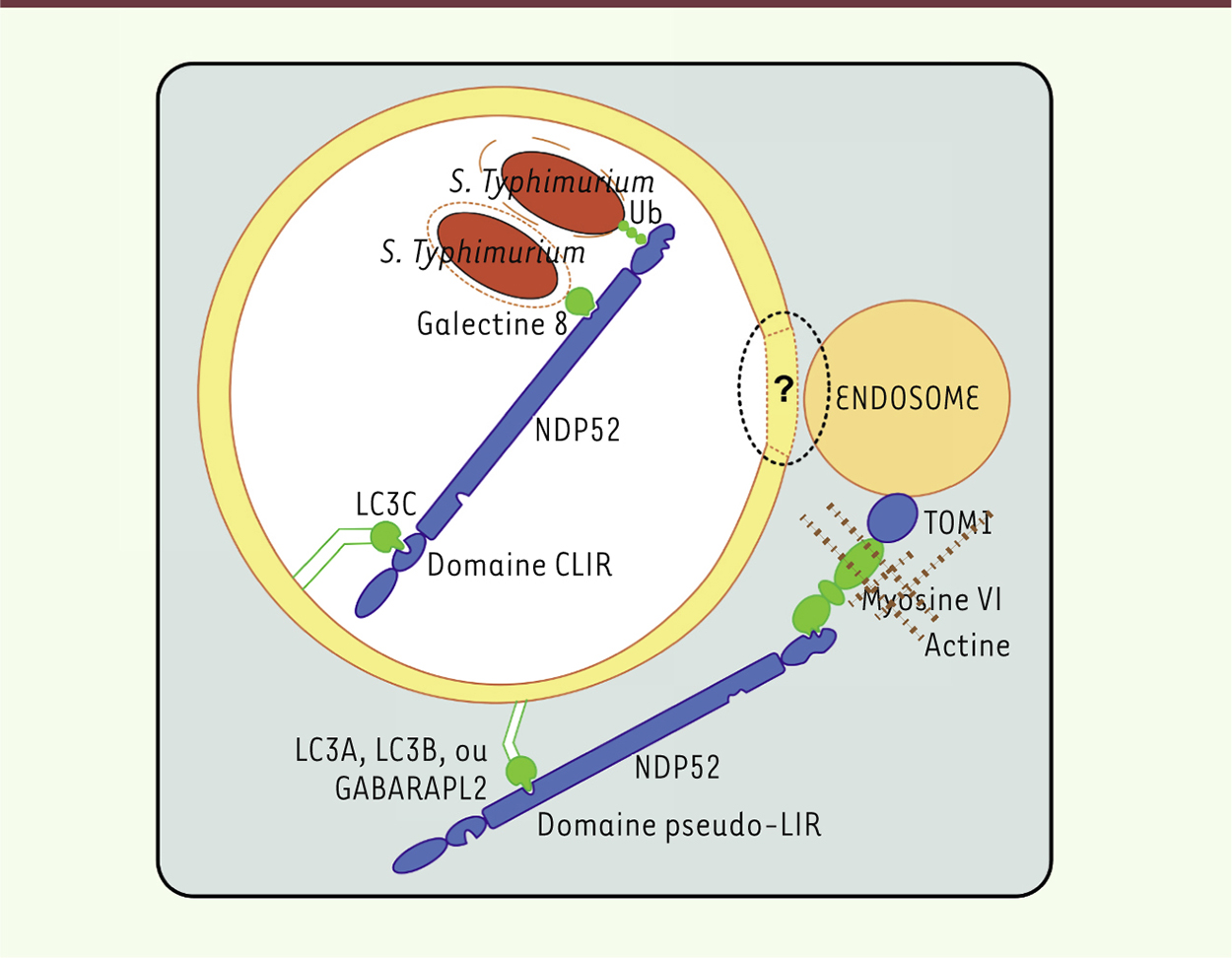
NDP52 guide les bactéries vers l’autophagosome NDP52 (nuclear dot protein 52) est l’un des récepteurs autophagiques qui permet de cibler des bactéries intracellulaires vers les autophagosomes en formation (Figure 2). Pour cela, NDP52 possède un domaine de liaison à la galectine 8, une protéine qui reconnaît des sites glysosylés de protéines. Ces sucres sont d’ordinaire uniquement exposés à l’extérieur des cellules, car portés par les glycoprotéines de la membrane plasmique. Lors de l’internalisation de bactéries comme Salmonella typhimurium, Listeria monocytogenes ou Shigella flexneri dans des vésicules d’endocytose, les sucres protéiques se retrouvent dans la lumière des endosomes. La tentative de sortie des bactéries de ces vésicules endommage la membrane endosomale, exposant alors les protéines cellulaires glycosylées à une reconnaissance par la galectine 8 [8]. Ce détecteur moléculaire d’une anormalité intracellulaire lie dès lors NDP52 qui, grâce à un domaine de liaison spécifique à la protéine LC3C (appelé domaine CLIR), concentre la vésicule endommagée avec la bactérie au sein d’un phagophore en croissance [9]. De plus, si les bactéries parviennent à s’échapper de l’endosome pour envahir le cytosol, leurs parois peuvent être rapidement recouvertes de petits peptides d’ubiquitine. NDP52, comme plusieurs autres récepteurs autophagiques, possède un domaine de liaison aux ubiquitines et peut ainsi à nouveau lier la bactérie et, en interagissant de manière concomitante avec LC3C, acheminer le microorganisme vers un autophagosome en formation [10].
 | Figure 2.
Modèle du double rôle joué par NDP52 dans la xénophagie. NDP52 lie S. Typhimurium indirectement, grâce à des domaines de liaison qui recrutent des intermédiaires moléculaires, la galectine 8 ou des ubiquitines, associés respectivement aux membranes endosomales endommagées par la bactérie internalisée ou aux bactéries s’étant échappées dans le cytosol. Via une interaction avec LC3C, NDP52 cible alors la bactérie vers l’autophagosome. De plus, NDP52 contribue à la fusion des autophagosomes avec la voie endo/lysosomale grâce à deux interactions supplémentaires, avec la myosine VI et un membre de la famille ATG8. L’ensemble de ces interactions concourt à l’efficacité de l’élimination de S. typhimurium par l’autophagie. Le mécanisme précis de fusion reste à être compris. |
|
NDP52, un facteur déterminant pour la dégradation de bactéries prisonnières d’autophagosomes Une fois séquestrés dans l’autophagosome, les pathogènes peuvent utiliser des subterfuges moléculaires pour, soit s’échapper de ce nouvel environnement, soit s’y adapter afin d’y proliférer. Cette adaptation peut passer par un ralentissement ou une inhibition de la maturation de l’autophagosome, afin d’éviter d’être la cible de l’activité lysosomale [3]. Il apparaît ainsi essentiel pour la cellule de rapidement et efficacement réguler la maturation de l’autophagosome contenant la bactérie, afin de la détruire. Nous avons montré que, indépendamment de sa fonction de récepteur autophagique, NDP52 a également la capacité de réguler la fusion entre l’autophagosome et un lysosome [11]. NDP52 possède deux domaines essentiels à la maturation des autophagosomes : un domaine de liaison à la myosine VI, et un autre capable de lier quelques membres particuliers de la famille ATG8 (LC3A, LC3B ou GABARAPL2, mais pas LC3C) grâce à un nouveau domaine identifié (appelé pseudo-LIR). En liant la myosine VI, la seule myosine motrice permettant un mouvement rétrograde des endosomes via son interaction avec la protéine endosomale TOM-1 (target of Mybl) [12], NDP52 est physiquement associée à la voie endo-lysosomale. En liant un membre des ATG8, NDP52 connecte alors l’endosome à l’autophagosome et permet leur fusion, promouvant ainsi la maturation autophagique [11]. Ainsi, pour que la xénophagie de S. typhimurium soit complète, il faut non seulement que la bactérie soit isolée au sein d’un autophagosome, mais également qu’elle soit dégradée. En intervenant successivement lors de ces deux étapes clés, NDP52 contribue à l’efficacité du contrôle d’une infection par l’autophagie. Il reste à déterminer avec précision comment NDP52 régule la fusion à proprement parler, et comment s’opère le lien fusionnel entre l’amphisome et le lysosome (Figure 2). Il est à noter que NDP52 régule également la maturation de l’autophagosome indépendamment d’infections bactériennes [11]. Dans la mesure où des dysfonctionnements de l’autophagie sont associés à de nombreuses pathologies [13], ces travaux, qui apportent des réponses sur les mécanismes moléculaires de l’autophagie, pourraient ouvrir de nouvelles perspectives pour la compréhension des processus pathologiques associés à des altérations de l’autophagie. |
Dans l’idée d’arriver, un jour prochain, au constat éclatant du vers célèbre de Don Rodrigue1 [14], il nous reste à comprendre si d’autres récepteurs autophagiques contribuent à l’efficacité de la xénophagie à la manière de NDP52 ; les (doubles ?) fonctions de ces récepteurs pourraient-elles être engagées de concert ou indépendamment ? Les autophagosomes en formation sont-ils prédestinés à accueillir de manière exclusive ou partagée des substrats apportés sélectivement par ces récepteurs ? Ces mécanismes sont-ils impliqués contre des microbes virulents et commensaux ? Mieux comprendre les liens étroits entre récepteurs autophagiques, pathogènes et maturation autophagique permettra de mieux lutter contre les agents infectieux, notamment ceux qui ciblent ces récepteurs pour en détourner la fonction. Avec ce qui pourrait apparaître comme un dilemme cornélien, assurer le ciblage des bactéries vers l’autophagie ou leur dégradation, nos travaux montrent l’efficience de NDP52 pour ces deux fonctions de la xénophagie, permettant à la cellule de remporter des batailles contre des envahisseurs pathogènes ; « Et le combat cessa faute de combattant » [14]. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Le travail de notre laboratoire sur l’autophagie est soutenu par l’Institut Universitaire de France (IUF), l’ANR et l’Association François Aupetit (AFA).
|
Footnotes |
1.
Codogno P. L’autophagie, dégrader plus pour vivre plus mais attention ! Med Sci (Paris). 2009; ; 25 : :323.–324. 2.
Rikihisa Y. Glycogen autophagosomes in polymorphonuclear leukocytes induced by rickettsiae . Anat Rec. 1984; ; 208 : :319.–327. 3.
Joubert PE, Pombo Gregoire I, Meiffren G, et al. Autophagie et pathogènes. Bon appétit Messieurs ! Med Sci (Paris). 2011; ; 27 : :41.–47. 4.
Levine B. Eating oneself and uninvited guests: autophagy-related pathways in cellular defense . Cell. 2005; ; 120 : :159.–162. 5.
Gutierrez MG, Master SS, Singh SB, et al. Autophagy is a defense mechanism inhibiting BCG and Mycobacterium tuberculosis survival in infected macrophages . Cell. 2004; ; 119 : :753.–766. 6.
Nakagawa I, Amano A, Mizushima N, et al. Autophagy defends cells against invading group A Streptococcus . Science. 2004; ; 306 : :1037.–1040. 7.
Deretic V. Autophagy as an innate immunity paradigm: expanding the scope and repertoire of pattern recognition receptors . Curr Opin Immunol. 2012; ; 24 : :21.–31. 8.
Thurston T, Wandel M, von Muhlinen N, et al. Galectin 8 targets damaged vesicles for autophagy to defend cells against bacterial invasion . Nature. 2012; ; 482 : :414.–418. 9.
Von Muhlinen N, Akutsu M, Ravenhill B, et al. LC3C, bound selectively by a noncanonical LIR motif in NDP52, is required for antibacterial autophagy . Mol Cell. 2012; ; 48 : :329.–342. 10.
Thurston TL, Ryzhakov G, Bloor S, et al. The TBK1 adaptor and autophagy receptor NDP52 restricts the proliferation of ubiquitin-coated bacteria . Nat Immunol. 2009; ; 10 : :1215.–1221. 11.
Verlhac P, Gregoire IP, Azocar O, et al. Autophagy receptor NDP52 regulates pathogen-containing autophagosome maturation . Cell Host Microbe. 2015; ; 17 : :515.–525. 12.
Tumbarello DA, Waxse BJ, Arden SD, et al. Autophagy receptors link myosin VI to autophagosomes to mediate Tom1-dependent autophagosome maturation and fusion with the lysosome . Nat Cell Biol. 2012; ; 14 : :1024.–1035. 13.
Choi AM, Ryter SW, Levine B. Autophagy in human health and disease . N Engl J Med. 2013; ; 368 : :1845.–1846. 14.
Corneille P. Le Cid . Paris: : Éditions J’ai Lu; , 2014 : :84..p. |