| |
| Med Sci (Paris). 2015 May; 31(5): 487–491. Published online 2015 June 9. doi: 10.1051/medsci/20153105009.Vue de l’intérieur Des circuits génétiques pour l’analyse de profils moléculaires intracellulaires Nicolas Lapique1* and Yaakov Benenson1 1Department of Biosystems Science and Engineering, École polytechinque fédérale de Zurich (ETHZ), Mattenstrasse 26, 4058Bâle, Suisse MeSH keywords: Animaux, Techniques de biocapteur, Compartimentation cellulaire, Lignée cellulaire, Conception d'appareillage, Analyse de profil d'expression de gènes, Régulation de l'expression des gènes tumoraux, Réseaux de régulation génique, Humains, Liquide intracellulaire, microARN, Modèles génétiques, Tumeurs, ARN tumoral, Protéines de répression, Biologie synthétique, Facteurs de transcription, Transcription génétique, génétique, composition chimique, métabolisme, anatomopathologie, biosynthèse, physiologie, méthodes, tendances |
Rencontre entre biologie naturelle et synthétique La capacité des organismes multicellulaires à coordonner les fonctions de cellules spécialisées est parfois menacée par la dérégulation d’une fonction particulière. Afin d’identifier un dysfonctionnement pouvant être d’origine endogène ou produit par un agent extérieur, l’organisme ne cesse de contrôler une myriade de paramètres pour se tenir prêt à opposer une réponse adaptée au problème. Il peut arriver que le corps produise une meilleure réponse avec l’aide d’un agent médicamenteux, mais il est très rare que les molécules thérapeutiques agissent avec autant de parcimonie et de précision que le système vivant. Il est évidemment souhaitable que les traitements chimiques soient toujours mieux ciblés, et on peut envisager produire des médicaments capables d’analyser leur environnement avant de décider de libérer leur(s) agent(s) actif(s). Dans Le voyage fantastique
1, un film de 1966, un traitement « intelligent » avait été imaginé à partir d’un sous-marin qui avait été miniaturisé avec un équipage de scientifiques à bord, le tout injecté chez une personne dans le coma. Avec quatre personnes aux commandes, le vaisseau-médicament pouvait intégrer une quantité innombrable d’informations et, ainsi, agir de manière très ciblée et efficace. Les machines fabriquées de nos jours ont une capacité d’analyse toujours plus grande, mais elles restent très limitées dans leur capacité de décryptage des réseaux d’informations des systèmes vivants. La construction, à partir des processus d’ingénierie classiques, de « nanomachines » capables de pénétrer dans les cellules et d’interagir avec des molécules présentes en quantités infinitésimales n’est pas une mince affaire. Un système créé à partir de composants biologiques se révèle être généralement bien mieux adapté pour fonctionner dans les cellules. La biologie synthétique est un nouveau domaine de recherche qui applique les principes de l’ingénierie, et crée, à partir des « briques » élémentaires du vivant, un dispositif susceptible d’accomplir une fonction désirée. Grâce à l’unicité du code de la vie, il est possible d’assembler et de connecter entre eux des éléments biologiques provenant du monde bactérien, végétal et animal, pour les faire fonctionner dans une grande variété de cellules appartenant à tous les règnes du vivant. Ces nouvelles « machines biologiques » ont été principalement développées à partir de circuits génétiques : ces circuits sont des ensembles de gènes sensibles à leur environnement et connectés entre eux par des mécanismes d’activation et de répression, dans le but de transmettre une réponse adaptée aux signaux reçus. Des systèmes biosynthétiques ont ainsi été conçus pour des applications très diverses, telles que l’utilisation de bactéries pour stocker de l’information dans leur ADN [1], de l’optogénétique pour stabiliser l’homéostasie du glucose [2, 9] (→) ou encore la reprogrammation de bactéries afin de limiter leur multiplication dans les tumeurs et détruire les cellules cancéreuses [3]. À partir du concept de calcul biomoléculaire [4], on peut concevoir des systèmes dont la puissance d’analyse est de plus en plus poussée. Dans cet article, nous détaillons les première et seconde générations de circuits génétiques capables d’intégrer le signal d’une demi-douzaine de molécules, afin d’identifier précisément différents types de cellules humaines en fonction des logiques implémentées [5, 6].
(→) Voir la Synthèse de G.P. Dugué et L. Tricoire, m/s n° 3, 2015, page 291
|
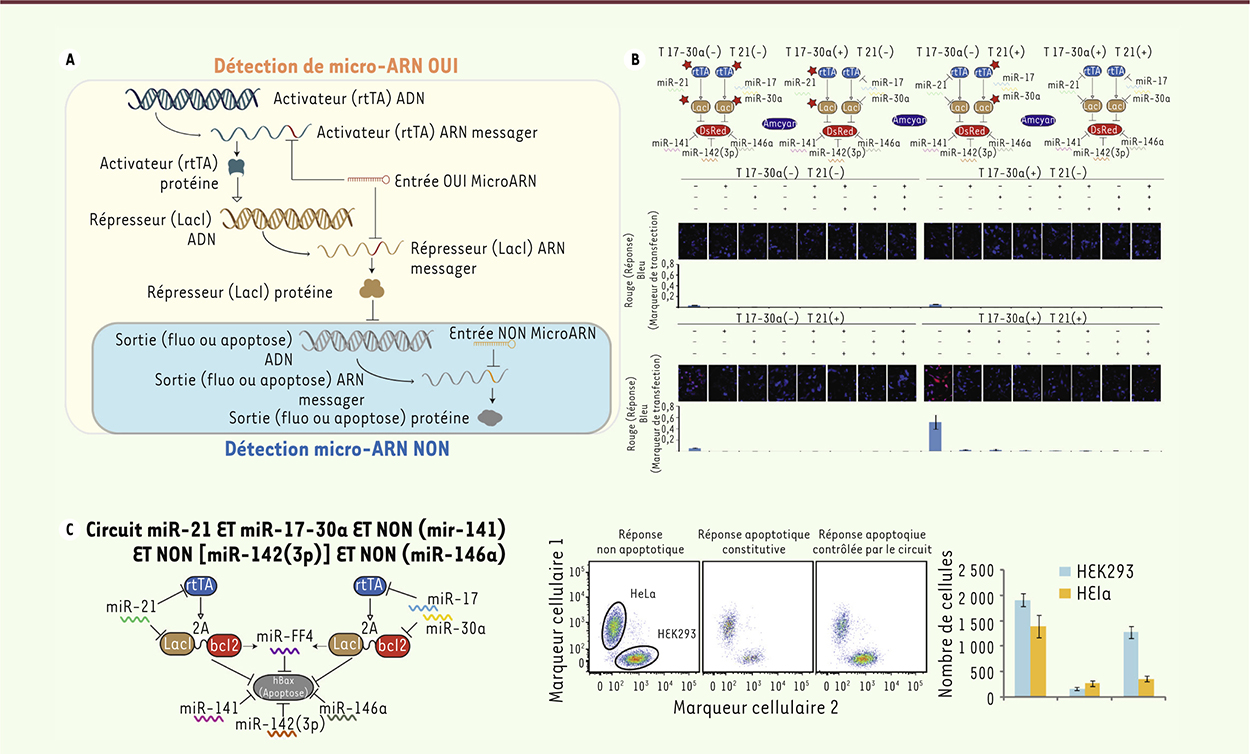
Des combinaisons de microARN pour identifier les cellules cancéreuses De nombreux critères morphologiques, métaboliques ou moléculaires caractérisent les cellules cancéreuses. Leur identification à partir de l’expression de facteurs endogènes tels que les récepteurs, les facteurs de transcription et les microARN est couramment utilisée, et certaines de ces molécules représentent des cibles thérapeutiques. Les microARN sont de courtes séquences d’ARN inhibitrices, qui s’hybrident à un ARN messager cible, et en régulent négativement la concentration et la traduction en protéine. Nous décrivons ici un circuit génétique que nous avons conçu pour analyser la signature microARN des cellules et agir sur celles-ci en fonction de leur profil moléculaire. Le circuit est programmé pour répondre à la détection d’une combinaison de microARN très précise, sous la forme de l’expression d’une molécule fluorescente ou d’une molécule toxique, selon que l’on souhaite respectivement identifier ou tuer les cellules exprimant un profil moléculaire prédéfini. Il existe des milliers de microARN différents, et chacune de ces molécules est relativement facile à détecter, car il suffit d’insérer la séquence complémentaire dans n’importe quel gène pour le réguler négativement. Avec les répresseurs tels que les microARN, la fonction logique la plus évidente à créer est l’opérateur NON, qui signifie qu’une sortie est produite uniquement en l’absence de la molécule d’entrée ; ainsi, en présence du répresseur, la protéine réponse du circuit est réprimée (Figure 1A, gauche). Il n’est pas plus difficile de produire la fonction logique ET avec deux opérateurs NON : dans ce cas, une sortie n’est produite qu’en l’absence de deux types de molécules, toutes deux contrôlant négativement la réponse (Figure 1A, centre). Enfin, la fonction OU de deux opérateurs NON produira une réponse dans tous les cas, excepté quand les deux entrées sont présentes (Figure 1A, droite). Pour les fonctions OUI, la sortie n’est pas directement contrôlée par l’entrée, un niveau intermédiaire constitué d’un système répresseur doit être intercalé. L’entrée microARN va réguler négativement un répresseur synthétique, qui, à son tour, va réprimer la production de la sortie. Ainsi, en présence de l’entrée, le répresseur sera réprimé et un signal sortie sera produit. Inversement, en l’absence d’entrée, le répresseur est présent, ce qui empêche la production de la sortie (Figure 1B, gauche). Comme pour l’opérateur NON, il est également possible d’implémenter des fonctions logiques ET et OU (Figure 1B).
 | Figure 1.
Fonction logique avec des molécules d’entrées répressives. Le(s) signal(aux) d’entrée du circuit est (sont) en noir, le signal de sortie est en rouge et le(s) élément(s) d’inversion du signal en brun(s). La couleur estompée indique l’absence de la molécule. Les connecteurs à extrémité droite indiquent une action de répression. Table de vérité (A) entrée(s) NON ; (B) entrée(s) OUI. |
|
Fonctionnement d’un profileur de microARN cellulaire L’inversion d’un signal répresseur en signal activateur nécessaire à l’implémentation de la logique OUI du microARN est évidemment la plus complexe, et compose la majeure partie du circuit (Figure 2A) [5]. Le répresseur synthétique est contrôlé négativement tant au niveau transcriptionnel que post-transcriptionnel par le microARN entrée. Pour le premier, le microARN va agir sur un activateur (rtTA) situé en amont qui va agir positivement sur la production du répresseur (LacI), ainsi que sur le répresseur lui-même (Figure 2A
encadrement jaune). Les microARN implémentés avec la fonction NON ciblent directement la sortie en l’empêchant de produire une protéine réponse (
Figure 2A
, encadrement bleu). Un profil moléculaire de six molécules a été établi à partir d’un atlas d’expression de microARN [7], qui reconnaît spécifiquement une lignée cellulaire dérivée d’un cancer du col de l’utérus (HeLa). Le profil cellulaire peut être décrit sous la forme d’une fonction booléenne, miR-21 ET miR-17-30a ET NON (miR-141) ET NON (miR-142[3p]) ET NON (miR-146a). Avec cinq entrées, il existe 32 configurations possibles, dont 31 doivent rester éteintes (Off) et une seule doit produire une réponse (On). Chacune des possibilité a été testée, et seule la configuration du profil désiré produit le signal voulu (On) (Figure 2B). Lorsque les trois entrées NON sont absentes, on peut observer que la réponse est légèrement produite. Afin d’éliminer au mieux le signal lorsque le profil moléculaire ne correspond pas (Off), un second répresseur a été ajouté sous la forme d’un microARN synthétique (miR-FF4) épissé à partir de l’ARN messager du premier répresseur (Figure 2C). La réponse de ce système prend la forme d’une protéine apoptotique (hBax), dont l’expression déclenche la mort cellulaire. L’action de hBax a été atténuée par une molécule antidote (Bcl-2), qui sera détruite par certains microARN lorsque la réponse est produite. La spécificité et la sensibilité du circuit génétique ont été testées sur des cellules non cancéreuses issues de rein embryonnaire (HEK-293), mélangées à des cellules cancéreuses HeLa (cancer du col de l’utérus). Dans l’enchevêtrement cellulaire in vitro, le circuit parvient à identifier et éliminer les cellules cancéreuses tout en préservant les autres (Figure 2C).
 | Figure 2.
Première génération de profileur de microARN. Dans les schémas, les flèches en arête indiquent la transcription (ADN → ARNm), les flèches à pointes noires indiquent la traduction (ARNm → protéine), les flèches à pointes blanches indiquent une activation et les connecteurs à extrémité droite indiquent la répression. A. Schéma de fonctionnement d’un circuit détectant un microARN OUI et un microARN NON. B. En haut, les schémas montrent les circuits utilisés pour tester les différents profils de microARN, les étoiles indiquent que le module est rendu insensible au microARN. En bas, sont montrés les résultats de la table de vérité avec les niveaux correspondants des signaux réponses. Sur les photographies d’analyse en microscopie, la réponse apparaît en rouge et le marqueur de transfection indiquant la présence du circuit dans la cellule apparaît en bleu. C. Circuit avec une réponse apoptotique pour éliminer les cellules HeLa. La figure centrale montre un graphique de cytométrie de flux utilisé pour identifier et compter les cellules. La réponse non apoptotique est une molécule fluorescente, la réponse apoptotique constitutive est la protéine hBax exprimée sans contrôle, la réponse apoptotique contrôlée par le circuit est la protéine hBax régulée par le circuit montré dans le schéma de gauche. Certaines figures ont été en partie reproduites à partir de [ 5] avec l’autorisation de l’éditeur. |
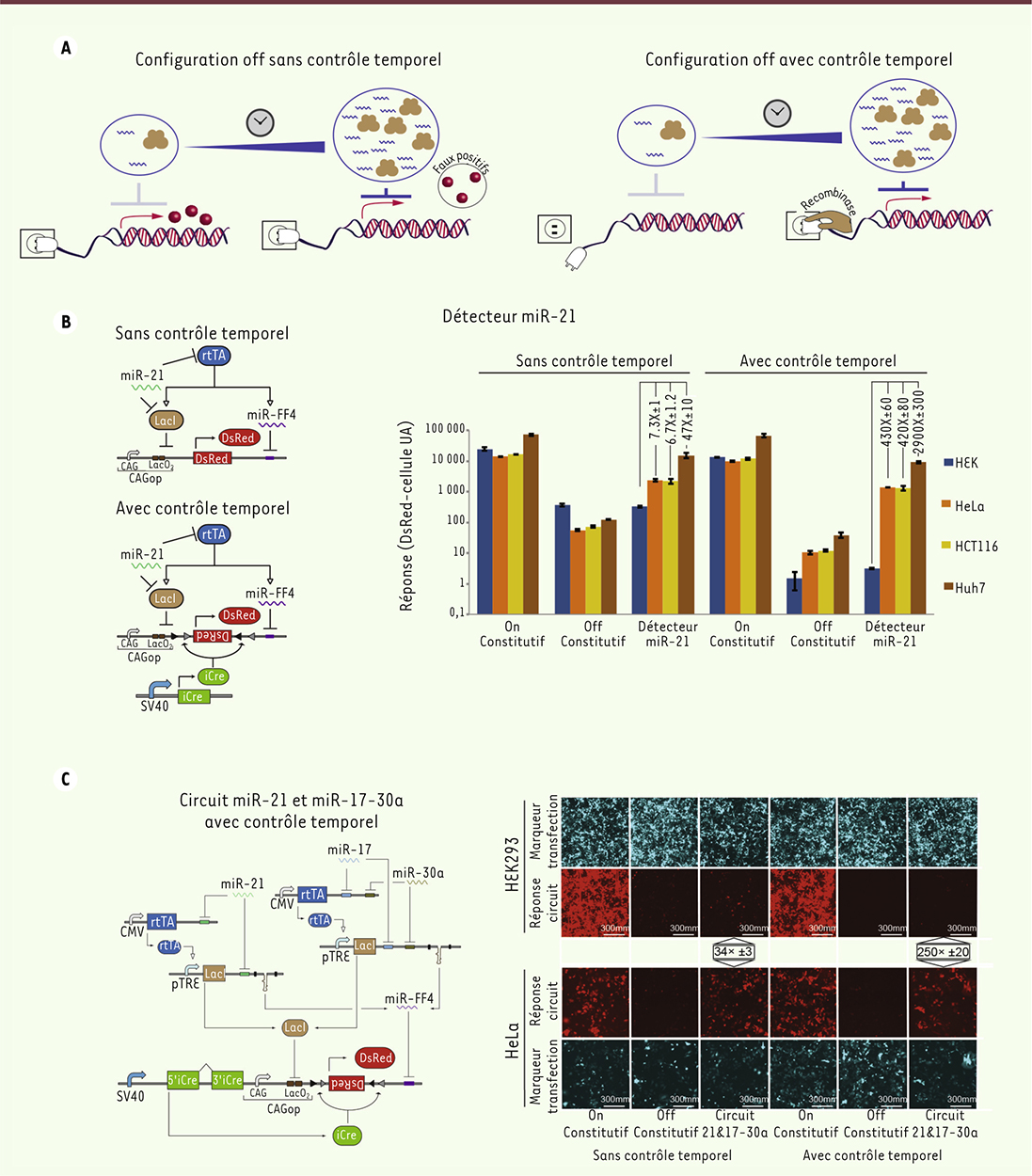
Pour dépasser la preuve de concept, il était important de comprendre pourquoi le circuit se déclenchait dans certaines cellules qui ne contiennent pas le bon profil moléculaire (~10 %), ce qui constituait un faux positif. En étudiant la dynamique de production du circuit grâce à la cytométrie de flux, nous avons observé que l’immense majorité des erreurs étaient produites lors de la phase d’assemblage du circuit [6] (Figure 3A, haut). Les composants régulateurs tels que rtTA, LacI, miR-FF4 mettent un certains temps (4-6 h) avant d’être pleinement opérationnels. Durant ce laps de quelques heures, le circuit n’est soumis à aucun contrôle. L’état par défaut de la réponse étant allumé, le circuit produit la molécule de sortie à pleine puissance avant d’être progressivement régulé par le module répresseur lorsque ce dernier atteint la concentration optimale. Un mécanisme retardant la production de la réponse a été introduit, de façon à ce que la cellule contienne suffisamment de molécules de contrôle lorsque la réponse commence à être produite (Figure 3A, bas). Ce retard est généré par l’intermédiaire d’une recombinaison génétique [8] : initialement la réponse est en position inversée, ce qui empêche sa synthèse, puis est finalement recombinée dans le sens de production (Figure 3B, bas). Cette opération – la production de l’enzyme (Cre recombinase) et la recombinaison – requiert un certain temps, ce qui permet d’éliminer presque intégralement les faux positifs. Les modules détectant les microARN ont été testés individuellement, et leur différence d’expression entre les cellules non cancéreuses dépourvues de miR-21 (HEK-293) et les cellules cancéreuses exprimant miR-21 (HeLa, HCT-116 et Huh-7) a été améliorée plus de 50 fois (Figure 3B). Le mécanisme de recombinaison a également été installé dans le circuit d’identification des cellules HeLa, ce qui améliore de près de 10 fois ses performances
(Figure 3C
). Cette nouvelle architecture permet une reconnaissance des cellules de manière digitale en éliminant pratiquement toutes les erreurs d’identification positive. Une configuration du système a également été testée dans laquelle la réponse s’exprime sous la forme d’une molécule extrêmement toxique (HSV-TK), et comme précédemment, le ciblage est sélectif, un seul type cellulaire est éliminé.
 | Figure 3.
Deuxième génération de profileur de microARN. A. Le schéma de gauche illustre le problème de « fuite » de la réponse, avant que les régulateurs soient pleinement opérationnels. Le schéma de droite illustre l’allumage tardif de la réponse pour permettre l’accumulation de modules régulateurs. B, C. Comparaison des performances du circuit avec et sans contrôle temporel. On constitutif indique la production de la réponse sans contrôle, Off constitutif indique la répression de la réponse sans contrôle, et détecteur miR-21 ou circuit 21/17-30a indiquent l’expression différentielle de la réponse en fonction du type cellulaire. B. À gauche, les schémas montrent les modules détecteur de miR-21 avec (bas) et sans (haut) contrôle temporel et leurs perfomances respectives (droite). C. À gauche, le schéma montre le circuit classificateur de cellule HeLa avec contrôle temporel, et les performances illustrées avec des prises de vues au microscope. Certaines figures ont été en partie reproduites à partir de [ 6] avec l’autorisation de l’éditeur. |
Les performances de la seconde génération de circuits permettront d’analyser des profils moléculaires de plus en plus complexes et, ainsi, d’agir sur des types de cellules extrêmement précis. Le circuit présenté ici a été spécifiquement développé à des fins thérapeutiques pour reconnaître les cellules cancéreuses. De nombreuses autres applications, en recherche comme dans une démarche diagnostique, requièrent une identification cellulaire fiable et robuste. La facilité avec laquelle le circuit peut être programmé pour reconnaître un profil moléculaire particulier permettra bientôt son utilisation dans de très nombreuses applications. |
Les auteurs déclarent que deux démarches de brevets ont été entreprises pour la première et la seconde génération de profileur moléculaire, dont une a abouti et est à présent enregistrée sous le numéro de publication WO2012012739 A3 et la seconde est en cours à l’European patent office sous le numéro d’enregistrement EP 14001960.5.
|
Footnotes |
1.
Farzadfard F, Lu TK. Synthetic biology. Genomically encoded analog memory with precise in vivo DNA writing in living cell populations . Science. 2014; ; 346 : :1256272.. 2.
Ye H, Daoud-El Baba M, Peng RW, Fussenegger M. A synthetic optogenetic transcription device enhances blood-glucose homeostasis in mice . Science. 2011; ; 332 : :1565.–1568. 3.
Anderson JC, Clarke EJ, Arkin AP, Voigt CA. Environmentally controlled invasion of cancer cells by engineered bacteria . J Mol Biol. 2006; ; 355 : :619.–627. 4.
Benenson Y. Biomolecular computing systems: principles, progress and potential . Nat Rev Genet. 2012; ; 13 : :455.–468. 5. Xie Z1, Wroblewska L, Prochazka L, et al. Multi-input RNAi-based logic circuit for identification of specific cancer cells . Science. 2011; ; 333 : :1307.–1311. 6.
Lapique N, Benenson Y. Digital switching in a biosensor circuit via programmable timing of gene availability . Nat Chem Biol. 2014; ; 10 : :1020.–1027. 7.
Landgraf P, Rusu M, Sheridan R, et al. A mammalian microRNA expression atlas based on small RNA library sequencing . Cell. 2007; ; 129 : :1401.–1414. 8. Schnütgen F, Doerflinger N, Calléja C, et al. A directional strategy for monitoring Cre-mediated recombination at the cellular level in the mouse . Nat Biotechnol. 2003;; 21 : :562.–565. 9. Dugué GP, Tricoire L. Principes et applications de l’optogénétique en neuroscience . Med Sci (Paris). 2015; ; 31 : :291.–303. |