| |
| Med Sci (Paris). 2015 May; 31(5): 469–472. Published online 2015 June 9. doi: 10.1051/medsci/20153105003.De la mouche à l’homme RACK1, un acteur essentiel de la traduction virale dépendante de l’IRES Mohamed Lamine Hafirassou,1,2* Carine Meignin,2,3 Thomas Baumert,1,2,4 and Catherine Schuster1,2** 1Inserm UMR1110, Institut de recherche sur les maladies virales et hépatiques, 3, rue Koeberlé, 67000Strasbourg, France 2Université de Strasbourg, 67000Strasbourg, France 3CNRS UPR9022, Institut de biologie moléculaire et cellulaire, 15, rue Descartes, 67000Strasbourg, France 4Institut hospitalo-universitaire (IHU), pôle hépato-digestif, hôpitaux universitaires de Strasbourg, 67000Strasbourg, France MeSH keywords: Animaux, Antiviraux, Protéines de Drosophila, Conception de médicament, Cellules eucaryotes, Protéines G, Humains, Thérapie moléculaire ciblée, Protéines tumorales, Biosynthèse des protéines, Coiffes des ARN, Virus à ARN, ARN viral, Récepteurs de surface cellulaire, Récepteurs cytoplasmiques et nucléaires, Séquences régulatrices de l'acide ribonucléique, Ribosomes, Réplication virale, pharmacologie, usage thérapeutique, antagonistes et inhibiteurs, physiologie, métabolisme, effets des médicaments et substances chimiques, génétique, ultrastructure |
Le ribosome support de la traduction de l’information génétique Le ribosome est une superstructure biologique universelle chargée de traduire l’information génétique portée par l’ARN messager (ARNm) en protéine(s). Il est formé de deux sous-unités (SU) caractérisées dans les années 1970 par leur constante de sédimentation (SU40S et SU60S chez les eucaryotes). Le ribosome eucaryote est composé de l’ARNr 18S constituant la SU40S, des ARNr 5S, 28S et 5,8S constituant la SU60S, et de plusieurs dizaines de protéines ribosomales associées spécifiquement à l’une ou l’autre sous-unité. Les ARNr jouent un rôle essentiel dans le mécanisme de traduction (reconnaissance de l’ARNm et catalyse de la formation de la liaison peptidique) [17]. Les progrès récents de la cristallographie, et surtout de la cryo-électromicroscopie, ont permis de mieux comprendre la structure et la fonction des composants du ribosome. Schématiquement, la SU40S prend en charge l’ARNm à traduire et, une fois les SU40S et SU60S assemblées pour former le ribosome, le ribosome débute et poursuit la synthèse protéique [1, 2]. Le ribosome a longtemps été perçu comme une structure ribonucléoprotéique figée dans sa mission de traduction de l’ARNm, la régulation de l’expression génique étant l’apanage des étapes transcriptionnelles et post-transcriptionnelles [3–5]. Plusieurs études récentes ont toutefois infirmé ce dogme en démontrant que les ribosomes ne constituaient pas une population homogène. Ainsi, en réponse à un stress par exemple, une hétérogénéité des protéines ou de l’ARNr peut être observée, ce qui entraînera une modification quantitative ou qualitative des ARNm traduits (« translatome ») [6]. |
Mécanismes de traduction dépendant de la coiffe versus dépendant d’un IRES La traduction chez les eucaryotes est majoritairement assurée par un mécanisme dépendant de la coiffe. En effet, simultanément à la transcription de l’ARNm, celui-ci acquiert sa maturation par l’ajout d’une coiffe en 5’, épissage, dépôt du complexe EJC (exon junction complex), pour se terminer par la polyadénylation en 3’. La coiffe en 5’ correspond à l’ajout d’une guanosine méthylée en position N7 (m7G), liée de façon covalente au nucléotide suivant par une liaison 5’-5’ triphosphate (m7G(5’)-PPP-(5’)N). Après transfert de l’ARNm mature du noyau dans le cytoplasme, la coiffe en 5’ est reconnue par le facteur d’initiation de la traduction eIF4E, permettant la mise en place du complexe d’initiation de la traduction et le recrutement de la SU40S (Figure 1A). La SU40S arrimée à l’ARNm va alors glisser le long de celui-ci (scanning) jusqu’à rencontrer le codon initiateur de la traduction, un codon AUG. Ce mécanisme est largement prédominant pour les ARNm cellulaires. À l’inverse, les virus à ARN effectuent généralement leur cycle biologique dans le cytoplasme, ce qui empêche l’ajout conventionnel d’une coiffe en 5’. Afin de protéger l’extrémité 5’ de leur génome, les virus ont développé différentes stratégies, comme l’ajout d’une protéine virale (par exemple VPg) ou la synthèse d’une coiffe par une enzyme virale [2]. Certains virus, dont le génome est un ARN de polarité positive de type ARNm, possèdent en 5’ de leur ARN une séquence formant une structure secondaire et tertiaire, appelée internal ribosome entry site ou IRES. L’IRES permet le recrutement du ribosome indépendamment de la coiffe en 5’ (Figure 1B). Ce mécanisme de traduction, qui nécessite moins de facteurs d’initiation de la traduction, constitue pour ces virus une manière astucieuse de s’affranchir de la traduction dépendante de la coiffe, mécanisme en général fortement inhibé lors d’une infection virale [7].
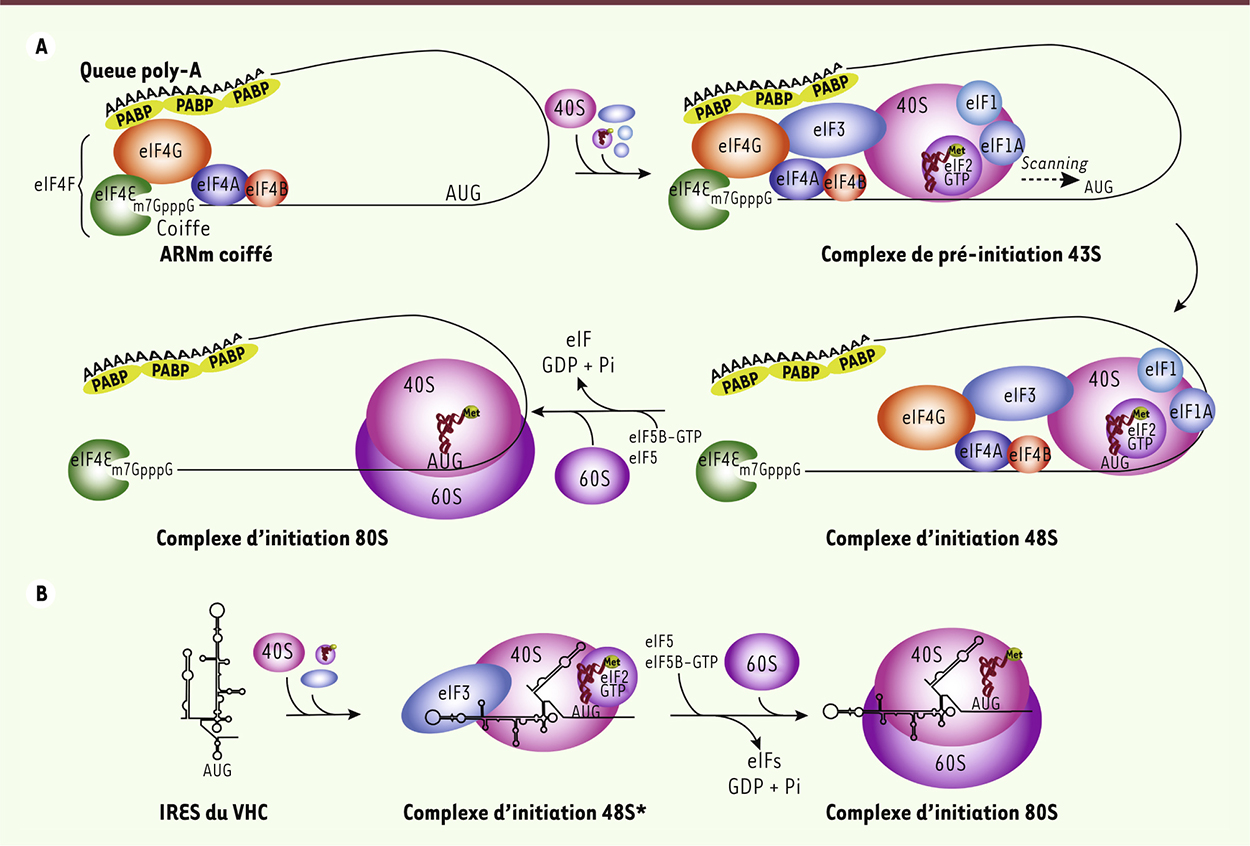
 | Figure 1.
Initiation de la traduction chez les eucaryotes. A. L’initiation de la traduction dépendante de la coiffe chez les eucaryotes. L’initiation de la traduction par scanning ou balayage implique la formation de trois complexes intermédiaires : le complexe de pré-initiation 43S, le complexe d’initiation 48S et le complexe d’initiation 80S. La majorité des ARNm cellulaires possèdent deux particularités à leurs extrémités : une coiffe en 5’ et une queue poly-A en 3’. Alors que la queue poly-A est reconnue par la protéine PABP (Poly-A binding protein), la coiffe (m7G-PPP-G) est reconnue par la protéine eIF4E (eukaryotic initiation factor 4E) appartenant au complexe d’initiation de la traduction eIF4F. Ce dernier permet le recrutement de la SU40S du ribosome, en association avec les facteurs eIF3, eIF1, eIF1A et le complexe ternaire eIF2-GTP-Met-tRNAiMet, pour former le complexe de pré-initiation 43S. La formation du complexe d’initiation 48S a lieu lorsque le complexe 43S atteint par scanning le codon d’initiation AUG et que les interactions codon-anticodon sont établies. Finalement, la formation du complexe d’initiation 80S a lieu lorsque la SU60S rejoint le complexe 48S à l’aide des facteurs eIF5 et eIF5B-GTP. Grâce à son activité GTPase, eIF5B hydrolyse le GTP associé, conduisant à la libération de eIF5B-GDP et des facteurs d’initiation de traduction, permettant à la traduction d’entrer en phase d’élongation. B. Initiation de la traduction IRES-dépendante du virus de l’hépatite C (VHC). L’IRES du VHC recrute la SU40S du ribosome en association avec eIF3 et le complexe ternaire eIF2-GTP-Met-tRNAiMet au niveau du codon initiateur AUG (sans scanning) pour former le complexe d’initiation 48S* (différent du complexe de pré-initiation 48S canonique). La formation ultérieure du complexe 80S dépend également de l’hydrolyse du GTP par eIF5B. |
|
Lutte antivirale, les facteurs de l’hôte en ligne de mire Classiquement, les thérapies antivirales ciblent le virus en proposant des « antiviraux à action directe ou DAA » (il s’agit d’analogues nucléosidiques, d’inhibiteurs de protéases, d’intégrase ou de polymérase) [8]. Ces molécules antivirales sont très efficaces chez la majorité des patients traités, mais peuvent malheureusement induire la sélection de virus mutants résistants qui requièrent la mise au point d’approches complémentaires [9]. Ceci est dû à la dynamique de réplication de ces virus et à la fidélité limitée des ARN polymérases virales lors de la réplication du génome viral. Une alternative thérapeutique consiste à mettre au point des molécules visant des composants cellulaires (host targeting agents) nécessaires au virus pour se multiplier, comme les récepteurs membranaires utilisés par le virus pour entrer dans la cellule ou des facteurs cellulaires requis dans la réplication, l’assemblage ou l’export viraux [10]. |
RACK1, une protéine ribosomale nécessaire à la propagation virale dépendante de l’IRES Les virus, parasites stricts par essence, ne possèdent ni système de traduction, ni système de production d’énergie. Au regard de la spécialisation fonctionnelle des ribosomes évoquée précédemment, nos récents travaux ont porté sur le rôle des protéines ribosomales de la SU40S dans la réplication virale [11]. RACK1 (receptor for activated C-kinase) est une protéine associée à la SU40S du ribosome. Elle appartient à la famille des WD repeat proteins qui, par leur fonction de protéines d’échafaudage, sont impliquées dans les voies de signalisation cellulaires [12]. Contrairement à celle de la plupart des protéines ribosomales, l’inhibition de l’expression de RACK1 n’affecte pas la survie des cellules de drosophile ou des cellules humaines. Chez la drosophile, l’absence de RACK1 induit toutefois une très forte baisse de la réplication des virus DCV (drosophila C virus) et CrPV (cricket paralysis virus), alors que la réplication d’autres virus, tels que VSV (vesicular stomatitis virus) ou FHV (flock house virus) reste inchangée. Une différence notoire entre ces virus est la présence en 5’ de leur génome ARN d’un IRES dans le cas de DCV et CrPV, et d’une coiffe dans le cas de VSV ou de FHV. RACK1 apparaît donc comme une protéine ribosomale discriminant les traductions dépendantes d’un IRES et celles dépendantes de la coiffe. L’importance de RACK1 dans la traduction dépendante d’un IRES a été confirmée chez l’homme en utilisant le virus de l’hépatite C (VHC) comme modèle de virus humain dépendant d’un IRES [13]. L’analyse des différentes étapes de l’infection virale a montré que RACK1 agit au niveau de l’étape de traduction des protéines virales, chez la drosophile comme chez l’homme. L’étude du mécanisme moléculaire a montré (1) que l’association de RACK1 au ribosome est essentielle dans ce mécanisme, (2) que dans le cas du VHC, RACK1 agit sur la traduction virale indépendamment de miR-122, un microARN hépatique impliqué dans la traduction de ce virus, et (3) que la sous-unité « j » du facteur d’initiation de la traduction eIF3 (eIF3j) est impliquée dans ce mécanisme de traduction particulier. RACK1 apparaît donc comme un facteur cellulaire nécessaire à la traduction des ARN viraux dépendants de l’IRES par les ribosomes (Figure 2).
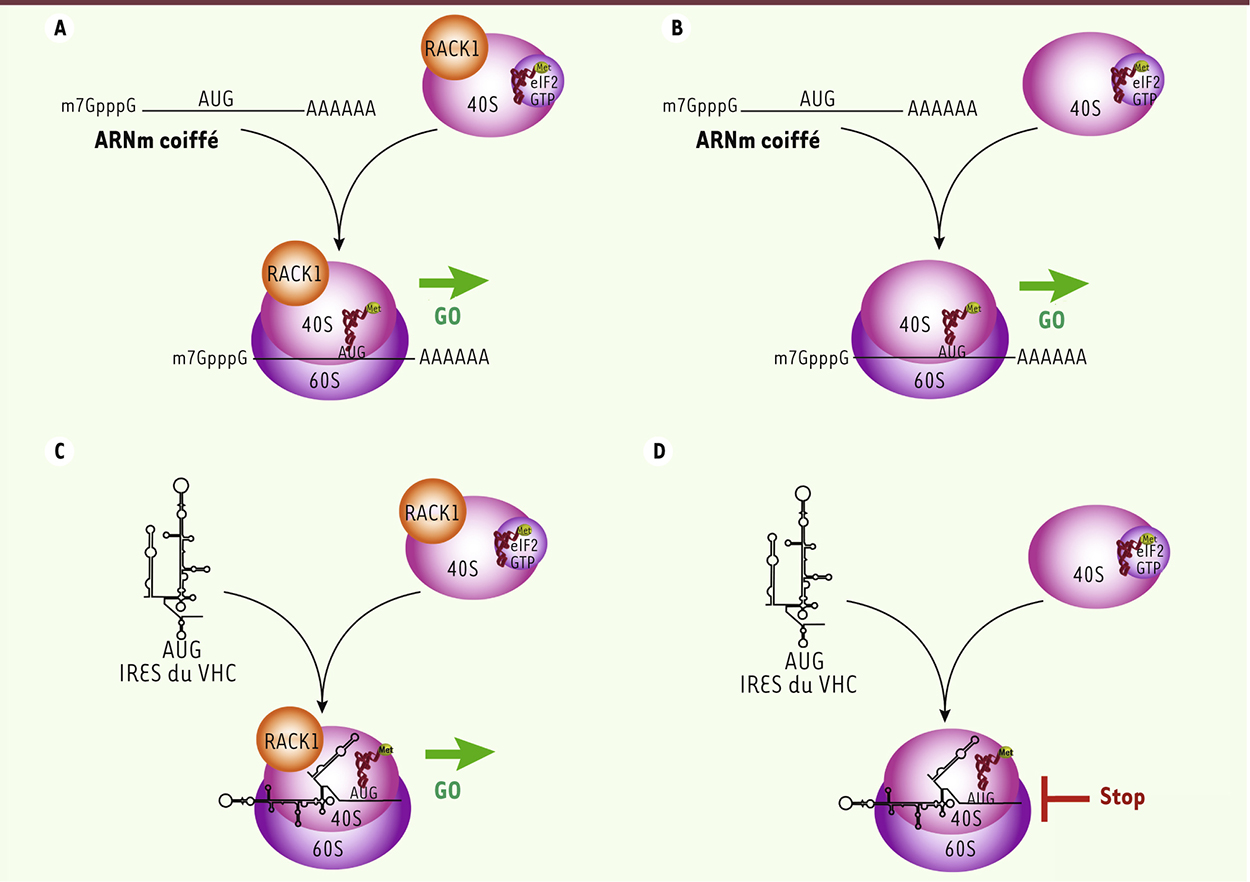
 | Figure 2.
Rôle de RACK1 dans la traduction dépendante de l’IRES. A, B. La traduction conventionnelle dépendante de la coiffe est indépendante de la présence (A) ou de l’absence (B) de RACK1. C, D. La traduction dépendante de l’IRES de virus tels que le virus de l’hépatite C (VHC) requiert la présence de RACK1 (C) et est inhibée lorsque que les ribosomes sont dépourvus de la protéine RACK1 (D). |
|
RACK1 : un facteur cellulaire de choix pour la lutte antivirale ? Notre découverte suggère un rôle de RACK1 dans la propagation d’autres virus contenant un IRES et ayant développé la même stratégie de piratage de la traduction cellulaire (par exemple virus de la poliomyélite, de la fièvre aphteuse, entérovirus 71, virus coxsackie B3). Des traitements à large spectre d’action ciblant RACK1 pour lutter contre les infections virales animales ou humaines sont donc envisageables. Décrypter les conditions dans lesquelles RACK1 est nécessaire à la cellule est toutefois un prérequis avant d’ouvrir de nouvelles perspectives thérapeutiques basées sur le blocage spécifique du site de reconnaissance des ARN viraux dépendants de l’IRES sur le ribosome, mais sans perturber le fonctionnement de la cellule hôte. RACK1 est conservée de la drosophile à l’homme, un homologue existe également chez la levure. L’invalidation génétique de RACK1 chez les drosophiles et les souris est létale [14, 15], suggérant un rôle crucial de RACK1 dans le développement. Cependant, lorsque l’invalidation de RACK1 est faite de façon conditionnelle chez des drosophiles adultes, celles-ci sont viables, et, d’autre part, les souris Rack1-/- meurent à l’étape de gastrulation [15], ce qui signifie que RACK1 n’est requis que pour la traduction d’une sous-population d’ARNm. Un rôle de RACK1 dans la traduction d’ARNm cellulaires dépendants de l’IRES est donc à considérer, car certains ARNm cellulaires peuvent être traduits à l’aide d’un IRES de manière exclusive (c’est le cas de gènes du développement, d’oncogènes) ou mixte. Dans ce dernier cas, il est proposé que les ARNm utilisent la traduction dépendante de la coiffe en conditions physiologiques et la traduction dépendante de l’IRES en situation d’urgence, la structure IRES ne se dévoilant qu’en réponse à des situations particulières comme la réponse à un stress [16]. La traduction dépendante de l’IRES permet, dans ce cas, une synthèse protéique rapide et efficace en réponse à une situation mettant en jeu la survie de la cellule. Au niveau fondamental, nos résultats démontrent une fois encore que des études menées sur les virus permettent d’identifier des mécanismes cellulaires fondamentaux impliqués dans la traduction des ARNm, et confirment la complexité du mécanisme de traduction. Nos travaux offrent de nouvelles clés pour comprendre « le code du ribosome » qui, selon la composition en protéines du ribosome, traduirait sélectivement certains ARNm, d’autres non. |
1.
Ramakrishnan, V. The ribosome emerges from a black box . Cell. 2014; ; 159 : :979.–984. 2.
Decroly E, Ferron F, Lescar J, Canard B. Conventional and unconventional mechanisms for capping viral mRNA . Nat Rev Microbiol. 2012; ; 10 : :51.–65. 3.
Kugel JF, Goodrich JA. Non-coding RNAs: key regulators of mammalian transcription . Trends Bioch Sci. 2012; ; 37 : :144.–151. 4.
Schubeler D. Function and information content of DNA methylation . Nature. 2015; ; 517 : :321.–326. 5.
Rouault TA. The role of iron regulatory proteins in mammalian iron homeostasis and disease . Nat Chem Biol. 2006; ; 2 : :406.–414. 6.
Xue S, Barna M. Specialized ribosomes: a new frontier in gene regulation and organismal biology . Nat Rev Mol Cell Biol. 2012; ; 13 : :355.–369. 7.
Balvay L, Soto Rifo R, Ricci EP, et al. Structural and functional diversity of viral IRESes . Biochim Biophys Acta. 2009; ; 1789 : :542.–557. 8.
Lou Z, Sun Y, Rao Z. Current progress in antiviral strategies . Trends Pharmacol Sci. 2014; ; 35 : :86.–102. 9.
Chung RT, Baumert TF. Curing chronic hepatitis C: the arc of a medical triumph . N Engl J Med. 2014; ; 370 : :1576.–1578. 10.
Zeisel MB, Lupberger J, Fofana I, Baumert TF. Host-targeting agents for prevention and treatment of chronic hepatitis C: perspectives and challenges . J Hepatol. 2013; ; 58 : :375.–384. 11.
Majzoub K, Hafirassou ML, Meignin C, et al. RACK1 controls IRES-mediated translation of viruses . Cell. 2014; ; 159 : :1086.–1095. 12.
Nilsson J, Sengupta J, Frank J, Nissen P. Regulation of eukaryotic translation by the RACK1 protein: a platform for signalling molecules on the ribosome . EMBO Rep. 2004; ; 5 : :1137.–1141. 13.
Niepmann M. Hepatitis C virus RNA translation . Curr Top Microbiol Immunol. 2013; ; 369 : :143.–166. 14.
Kadrmas JL, Smith MA, Pronovost SM, Beckerle MC. Characterization of RACK1 function in Drosophila development . Dev Dyn. 2007; ; 236 : :2207.–2215. 15.
Volta V, Beugnet A, Gallo S, et al. RACK1 depletion in a mouse model causes lethality, pigmentation deficits and reduction in protein synthesis efficiency . Cell Mol Life Sci. 2013; ; 70 : :1439.–1450. 16.
Fitzgerald KD, Semler BL. Bridging IRES elements in mRNAs to the eukaryotic translation apparatus . Biochim Biophys Acta. 2009; ; 1789 : :518.–528. 17.
Mace K, Giudice E, Gillet R. La synthèse des protéines par le ribosome . Med Sci (Paris). 2015; ; 31 : :282.–290. |