| |
| Med Sci (Paris). 2015 April; 31(4): 423–431. Published online 2015 May 8. doi: 10.1051/medsci/20153104017.Force et spécificité du criblage pour des molécules bioactives au CMBA-Grenoble
Une plate-forme dédiée à la découverte et à l’analyse de molécules bioactives et candidats médicaments Caroline Barette,1** Emmanuelle Soleilhac,1 Céline Charavay,1 Claude Cochet,2 and Marie-Odile Fauvarque1* 1Université Grenoble Alpes ; CEA-Direction des sciences du vivant, Institut de recherches en technologies et sciences pour le vivant, iRTSV-BGE-CMBA, CEA-Grenoble; Inserm UMRS_1038, 17, rue des Martyrs, 38054Grenoble Cedex 9, France 2Université Grenoble Alpes ; CEA-Direction des sciences du vivant, Institut de recherches en technologies et sciences pour le vivant, BCI ; Inserm UMRS_1036, iRTSV-BCI-KIN, CEA-Grenoble, 17 rue des Martyrs, 38054 Grenoble Cedex 9, France |
En tant que composés chimiques interférant avec le vivant, les molécules bioactives représentent des enjeux majeurs pour la santé et l’environnement. Isolées à partir d’extraits naturels de divers organismes vivants (plantes, bactéries, champignons, organismes marins, etc.), les molécules bioactives peuvent ensuite être purifiées ou synthétisées au bénéfice de notre pharmacopée moderne (par ex. aspirine, paclitaxel, antibiotiques, etc.). Une autre stratégie de découverte consiste à sélectionner des molécules bioactives à partir de collections de molécules fabriquées par synthèse chimique (par exemple Glivec1). Dans la plupart des cas, la découverte de molécules bioactives repose sur la capacité à cribler de manière automatisée plusieurs milliers à centaines de milliers de composés stockés en microplaques - les chimiothèques - dans des tests biologiques adaptés à ce format et utilisant des protéines purifiées ou des cellules mises en culture. En raison des risques élevés d’échecs et des coûts toujours croissants de la valorisation clinique de nouvelles molécules thérapeutiques, il semble légitime de se poser la question des chances de réussite des laboratoires de recherche académiques dans la découverte de molécules bioactives ou de nouveaux médicaments [1]. La réponse réside au sein même de ces laboratoires, où les meilleurs experts internationaux - forts de nombreuses années de recherche dans leur domaine de spécialité - ont élaboré une panoplie de tests biologiques ingénieux et pertinents. Or, le développement d’un test biologique pertinent est la clé de la réussite des cribles automatisés à haut débit (high throughput screening ou HTS) ou par imagerie automatisée (high content screening ou HCS). À défaut d’interroger un très grand nombre de molécules, les plates-formes académiques mettent en place des stratégies de criblage moléculaire (sur protéines purifiées) ou phénotypiques (sur cellules vivantes) de collections représentatives d’un espace chimique diversifié ou, au contraire, de collections focalisées en fonction de l’effet recherché. Cette tendance se retrouve également dans les industries pharmaceutiques : ces dernières années ont vu une réduction du criblage à très grande échelle (> 100 000 molécules) au profit d’une stratégie qui fait appel à des chimiothèques plus réduites, avec un ensemble de composés parfois sélectionnés in silico sur des critères prédictifs (comme la biocompatibilité, la capacité à cibler des interfaces protéine-protéine, ou encore la capacité à s’insérer dans un site catalytique dont la structure est connue). Les criblages phénotypiques qui s’effectuent sur cellules vivantes, voire sur des organismes vivants uni- ou pluricellulaires (algues, amibes, vers nématodes, poissons zèbre, mouches drosophiles) offrent quant à eux une pertinence accrue vis-à-vis de la maladie ciblée et/ou de la physiologie du système biologique étudié, puisqu’ils permettent l’intégration d’un phénotype à l’échelle d’un organisme entier [2, 37] (→).
(→) Voir la Synthèse de C. Voisset et M. Blondel, m/s n° 12, décembre 2014, page 1161
Utilisés quotidiennement en laboratoire, les tests et le savoir-faire des chercheurs constituent une ressource largement sous exploitée pour le criblage et la découverte de molécules bioactives. Le champ d’application de ces molécules ne se limite pas au secteur du médicament : les molécules chimiques découvertes au centre de criblage pour des molécules bioactives du CEA-Grenoble (CMBA-Grenoble) se révèlent des outils prometteurs tant en recherche fondamentale [3–9] que pour des applications dans les domaines de la santé2,, de l’environnement ou des bioénergies3. |
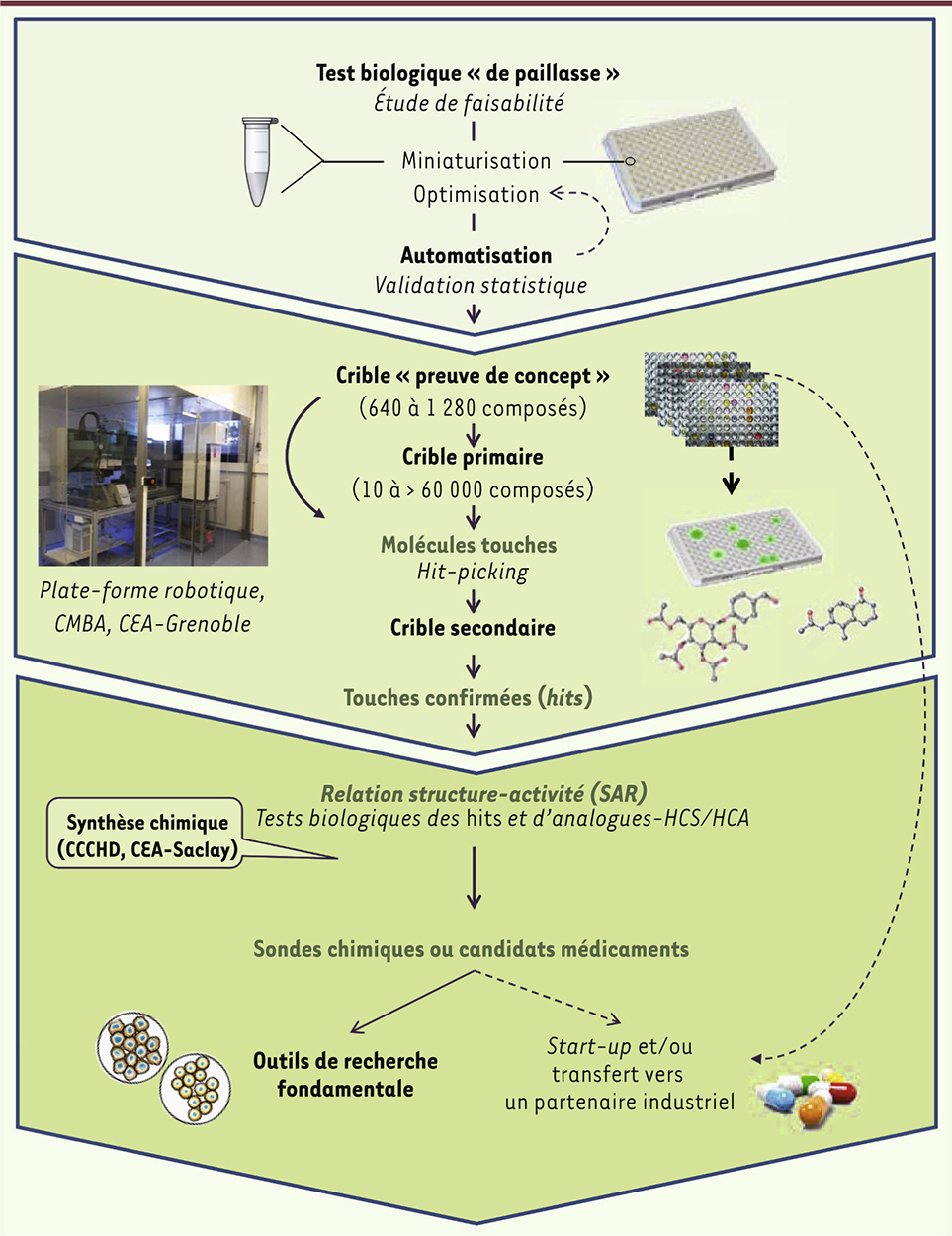
Stratégie de criblage du CMBA Les plates-formes de criblage académiques ouvertes à la communauté scientifique, telles que le groupe de chimie combinatoire, criblage cellulaire et chémogénomique (G5C, labélisé IBiSA, http://www.ibisa.net/), qui regroupe le CMBA-Grenoble et le CCHD (chimie combinatoire et criblages à haut débit du CEA-Saclay), jouent un rôle essentiel pour inciter et aider les chercheurs à utiliser « l’or » qu’ils ont entre les mains, en d’autres termes, pour transposer un savoir-faire et des essais expérimentaux imaginés par les chercheurs, en tests automatisables adaptés au criblage robotisé. Concrètement, les membres du CMBA-Grenoble accompagnent les scientifiques dans l’évaluation de la faisabilité de leur projet de criblage, et les assistent dans les étapes de miniaturisation et d’optimisation d’un test biologique « de paillasse » en microplaques (96 ou 384 puits) (Figure 1). Ils procèdent ensuite à l’automatisation du test avant sa mise en œuvre dans un crible pilote d’une banque de molécules chimiques de petite ou moyenne envergure, typiquement la collection Prestwick Chemical Library® - constituée de 1 280 molécules majoritairement approuvées pour une utilisation en clinique -, ou la CNE (Chimiothèque nationale essentielle) qui comprend 640 molécules de structures variées représentatives de près de 35 000 molécules synthétisées et collectées par les laboratoires de chimie français au sein de la Chimiothèque nationale (CN, http://www.chimiotheque-nationale.org/) [10, 38] (→). Le crible pilote apporte la preuve de concept de la pertinence biologique du test, de sa robustesse statistique et de sa portabilité sur une plate-forme automatisée. Le CMBA-Grenoble est ensuite en mesure de réaliser un crible primaire de tout ou partie de ses collections commerciales ou académiques totalisant plus de 60 000 entités moléculaires [11] (Figure 1). Les molécules candidates issues du crible primaire d’une ou plusieurs chimiothèques sont testées à nouveau dans un crible de validation pour sélectionner des « touches » (hits en anglais) confirmées. Grâce à l’accès à des bases de données de molécules disponibles sur le marché, le porteur de projet sera aidé par les ingénieurs de la plate-forme pour le choix d’une série d’analogues à tester afin de déterminer les fonctions chimiques strictement nécessaires à l’activité biologique de chaque molécule touche (SAR pour structure activity relationship), ainsi que pour la mise en œuvre de tests biologiques complémentaires qui conduiront à la sélection d’une « sonde chimique » ou « tête de série » (lead). Cette sonde a toutes les chances de constituer un outil puissant et original pour la poursuite d’une recherche de pointe dans le domaine étudié par le scientifique qui dirige le projet [3, 12–14], et, selon l’objectif visé, elle entrera, ou non, dans un processus d’optimisation et de valorisation pharmacologique ou industrielle (Figure 1).
(→) Voir la Synthèse de Florence Mahuteau-Betzer, page 417 de ce numéro
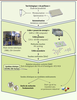 | Figure 1.
Stratégies de criblage et de valorisation. À partir d’un test biologique « de paillasse » imaginé par un chercheur, les ingénieurs du CMBA définissent des contrôles pour la validation statistique du test et élaborent les conditions optimales d’automatisation en termes de débit et de pertinence pour l’identification des molécules bioactives recherchées. Une fois validé, ce test est mis en œuvre lors d’un crible « preuve de concept » sur des banques de molécules de taille restreinte, avant le crible d’une plus grande collection de molécules. Les molécules candidates issues du crible primaire ( hits primaires) sont repiquées parmi les stocks ( hit-picking) et regroupées en microplaques pour être testées à nouveau lors d’un crible de validation. À l’issue du crible secondaire, les molécules touches validées (ou confirmed hits) sont optimisées par étude de la relation structure-activité (SAR) à partir d’analogues structuraux présentant des fonctions chimiques modifiées. Au cours de la SAR, toute une batterie de tests biologiques sont proposés, dont des tests phénotypiques par imagerie à haut contenu (voir
Figure 3
), afin de caractériser l’activité biologique des composés (profilage pharmaceutique). Au terme de ce processus, les molécules présentant une bioactivité maximale et une bonne sélectivité sont sélectionnées comme sondes pour des stratégies de génétique chimique en recherche fondamentale. Les molécules présentant un potentiel thérapeutique sont valorisées via un dépôt d’invention, la création de start-up et/ou le transfert à un partenaire industriel. La méthodologie de criblage est l’étape souvent la plus longue et la plus délicate du processus de criblage à haut débit. Une fois validée par un crible « preuve de concept », elle représente un savoir-faire à haute valeur ajoutée qui peut être transféré directement à un partenaire industriel. |
|
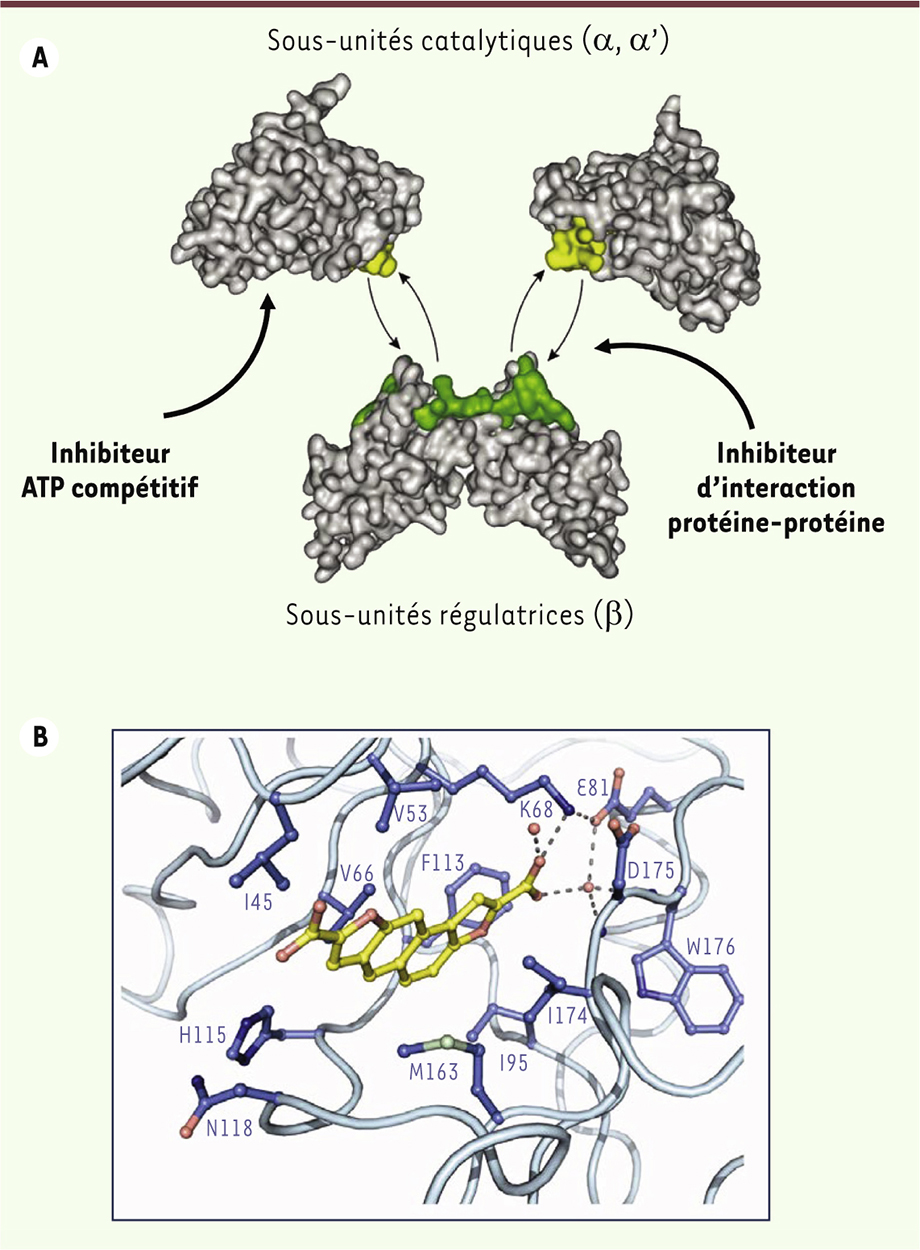
Des molécules pour modifier la fonction d’une cible thérapeutique : l’exemple de la protéine kinase CK2 Jusqu’à ces dernières années, les efforts de la communauté scientifique, aussi bien dans le monde académique qu’industriel, se sont essentiellement concentrés sur la recherche de molécules capables d’interférer avec des protéines, autrement dit, des cibles thérapeutiques, dans l’espoir de contrer leur pouvoir pathogénique (on parle d’approche target-based) [15]. Le CMBA-Grenoble dispose des méthodologies nécessaires à la mise en œuvre de tests réalisés in vitro sur des protéines d’intérêt en biologie. En recherche, les molécules sélectionnées par ce type d’approche sont en tout premier lieu utilisées pour bloquer tout ou partie des fonctions d’une protéine, à l’image des mutations en génétique [16, 17]. Les sondes chimiques utilisées dans ces essais permettent cependant de s’affranchir d’éventuels problèmes de létalité causés par la perte d’une protéine induite par les mutations. En outre, elles évitent des problèmes de redondance fonctionnelle en ciblant l’ensemble des protéines similaires. Les sondes chimiques représentent aussi des outils d’investigation irremplaçables pour aborder des questions sous un angle dynamique : l’expérimentateur peut évaluer les effets biologiques d’une molécule dans les minutes qui suivent son addition au milieu de culture cellulaire, afin de comprendre le rôle de sa cible biologique dans des processus d’adaptation ou de réponse cellulaire à des stimulus extérieurs (sans interférer avec l’état physiologique de cette cellule préalablement à l’expérience, ce qui n’est pas possible dans le cas d’une mutation permanente de la protéine). Finalement, les sondes chimiques peuvent interférer de façon partielle avec la fonction d’une protéine. Ainsi, un inhibiteur enzymatique permet de différencier la fonction catalytique d’une enzyme de son rôle structurel dans la formation de complexes multi-protéiques. A contrario, un inhibiteur d’interaction protéine-protéine empêchera spécifiquement l’interaction d’une protéine avec un de ses partenaires tout en laissant libres d’autres interfaces fonctionnelles. Finalement, la détermination de la structure d’une protéine peut être facilitée par sa co-cristallisation avec une sonde chimique déterminée dans ces essais, qui favorise sa stabilisation dans un état actif ou inactif [18]. La démonstration du potentiel oncogénique de la protéine kinase CK2 et sa validation comme biomarqueur dans différentes pathologies cancéreuses font de cette kinase une cible attractive pour la recherche de petites molécules d’intérêt thérapeutique [19, 20]. Cette sérine/thréonine kinase est une holoenzyme formée de l’association réversible de deux sous-unités catalytiques α ou α’ avec un dimère de deux sous-unités régulatrices β (Figure 2A). La multiplicité de ses cibles et de ses fonctions biologiques nécessite des outils d’investigation dynamique impliquant de nouvelles sondes chimiques [21]. La mise au point d’un test enzymatique simple et robuste a permis d’identifier des inhibiteurs présentant des propriétés anticancéreuses prometteuses (Tableau I). Ainsi, des dérivés des pyridocarbazoles et des benzopyridoindoles ont été caractérisés comme des inhibiteurs de CK2, agissant comme compétiteurs de l’ATP et présentant des propriétés antiprolifératives dans un modèle de xénogreffe de glioblastome humain [5]. Trois dérivés de l’acide 2,8-difurandicarboxylique sont également de puissants inhibiteurs compétitifs de l’ATP, à la fois pour CK2 et la kinase PIM, une autre sérine/thréonine kinase oncogénique. Les structures cristallographiques des complexes réalisés avec CK2 (ou PIM) montrent comment ces inhibiteurs chimiques se logent dans le site catalytique en prenant la place du substrat de l’enzyme (Figure 2B). CK2 et PIM sont des protéine kinases constitutivement actives, dont la fonction est altérée conjointement dans les leucémies et le cancer de la prostate. La faculté de bloquer simultanément CK2 et PIM dans ce type de cancers est donc particulièrement intéressante [6]. Des dérivés des azonaphatalènes, qui induisent un changement de conformation de type allostérique après leur fixation sur CK2, se montrent efficaces dans un modèle de xénogreffes de glioblastome [4]. Les polyoxométalates (POM), quant à eux, sont des agrégats nanométriques anioniques de métaux de transition (vanadium, molybdène, tungstène). Ces molécules ciblent un segment régulateur de CK2a avec une forte affinité, et l’inhibent par un mécanisme de type allostérique, en bloquant l’interaction entre le segment d’activation et la région amino-terminale de la kinase [7, 8]. Bien que n’ayant pas les propriétés requises pour une utilisation sur des cellules vivantes, ces molécules représentent néanmoins des sondes moléculaires intéressantes pour la détermination d’un site d’inhibition allostérique et la compréhension du mode de régulation de l’enzyme.
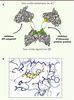 | Figure 2.
Structure de l’holoenzyme CK2. A. CK2 est un complexe moléculaire formé par l’association réversible de deux sous-unités catalytiques a/a’ avec un dimère de sous-unités régulatrices b. Le criblage de diverses chimiothèques a permis d’identifier plusieurs familles d’inhibiteurs ciblant le site catalytique sur a/a’ (ATP compétitifs) ou l’interface entre les 2 sous-unités (voir
Tableau I
). Les deux types d’inhibiteurs devraient avoir des effets différents sur l’activité de CK2 : les inhibiteurs ATP compétitifs affectent la phosphorylation de tous les substrats de la kinase, alors que les antagonistes de l’interaction entre ses sous-unités perturbent spécifiquement les substrats dont la phosphorylation dépend de cette interaction protéine-protéine. B. Structure du site catalytique de CK2a en présence d’un inhibiteur ATP compétitif (l’acide 2,8-difurandicarboxylique). La structure plane et le groupement carboxyle de cette molécule lui permettent de s’insérer dans le site de liaison de l’ATP pour y former une liaison forte avec la lysine catalytique (K68). |
Tableau I.
| Molécule |
Cible |
IC50 |
Structure chimique |
Mécanisme d’action |
Champ d’application |
| Ellipticines et analogues (pyridocarbazoles et benzopyridoindoles) |
CK2 |
300-500 nM |
|
ATP compétitif, ciblent le site catalytique |
Glioblastomes (tests précliniques sur xénogreffes de glioblastomes) [5] |
|
| Acide 2,8-difurandicarboxylique (et dérivés) |
CK2
PIM |
50-100 nM |
|
ATP compétitif, ciblent le site catalytique |
Leucémies, cancer de la prostate [6] |
|
| Azonaphtalènes (Quinobène) |
CK2 |
400 nM |
|
Inhibiteurs allostériques (induisent un changement de conformation du site catalytique) |
Propriétés antitumorales (sur cellules cancéreuses en culture et régression tumorale [xénogreffes de glioblastomes]) [4] |
|
| Composés inorganiques de la famille des Polyoxométalates (POM)* |
CK2 |
1-5 nM |
|
Non ATP compétitifs, Ciblent un segment régulateur de CK2a [8] |
Mécanisme d’inhibition de l’activité kinase
Actifs in vitro uniquement |
Inhibiteurs de CK2 : mécanismes d’action et champs d’application. *Les POM sont des complexes de métaux de transition et de ligands oxo de formule : MO4n- où M = molybdate, vanadate, ou tungstène. Leur structure repose sur la connexion de polyhèdres de métaux de transition (M) avec des atomes d’oxygène (O). Leur composition et leur structure sont donc fondamentalement différentes de celles des inhibiteurs organiques qui, eux, ont un squelette en carbone. Les trois familles principales de POM sont les structures de type : Anderson (XM6O24n-), Keggin (XM12O40n-), et Dawson (X2M18O62n-). La structure représentée ici est de type Dawson. |
L’association réversible des sous-unités catalytiques et régulatrices de CK2 est un paramètre essentiel à la régulation de l’activité cellulaire de cette kinase [22]. Une approche peptidique ainsi qu’une stratégie de criblage in silico ont permis d’identifier les premières molécules qui, en ciblant cette interface, inhibent cette interaction protéine-protéine de haute affinité [23]. Ces molécules originales sont à l’origine d’une stratégie non conventionnelle destinée à manipuler l’activité catalytique de CK2 dans les cellules normales ou cancéreuses [9, 24]. |
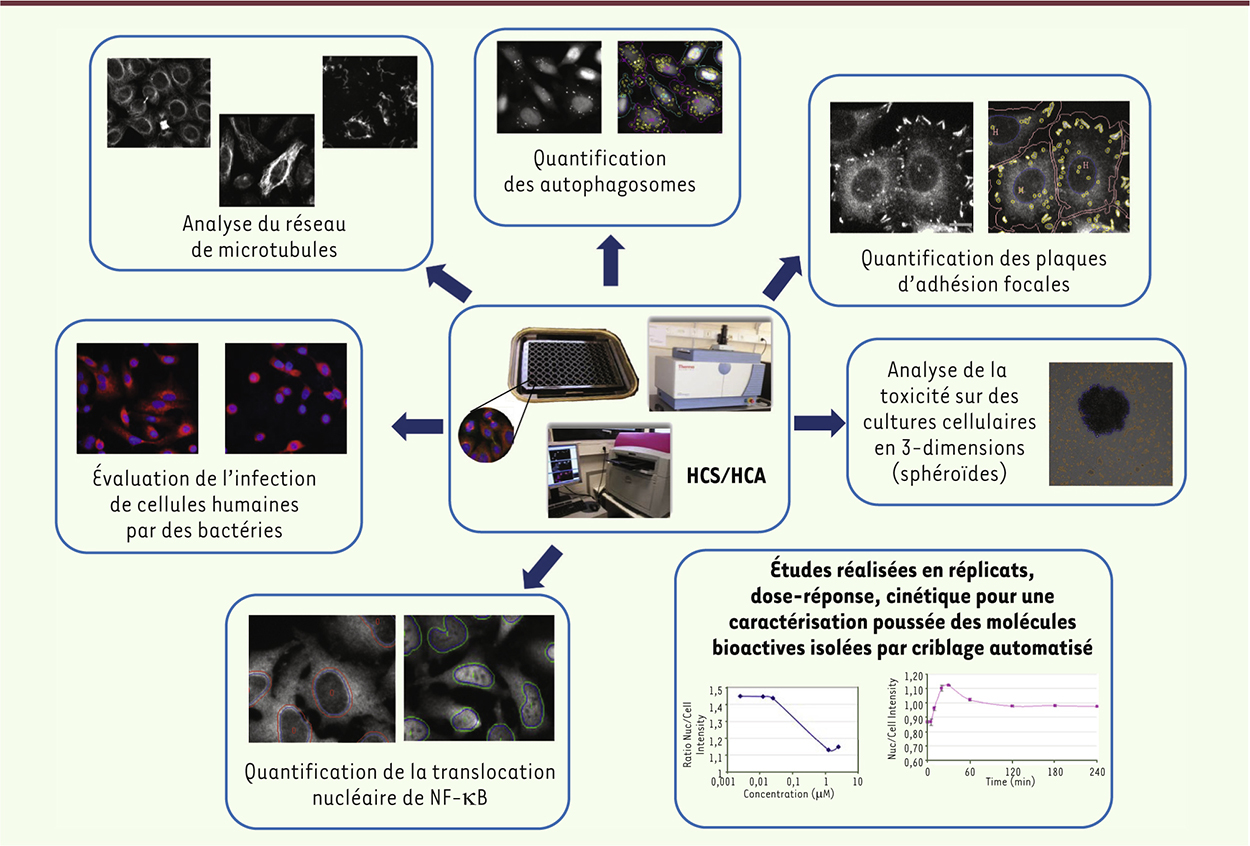
Les promesses du criblage phénotypique et du criblage à haut contenu en information : des stratégies à haut potentiel développées au CMBA La possibilité d’interférer chimiquement avec une cible biologique purifiée a largement incité la recherche de nouvelles cibles pertinentes par des approches génétiques, génomiques ou protéomiques et vice versa. Cependant, l’inhibition d’une seule cible ne suffit pas toujours à enrayer des maladies d’origine multifactorielle ; ce constat est particulièrement vrai pour le cancer, où l’apparition de cellules résistantes à la chimiothérapie provoque rechutes et métastases. D’où parfois un sentiment mitigé de réussite des approches pharmacologiques de criblage target-based au cours de ces deux dernières décennies. Le criblage phénotypique aborde différemment la question de l’efficacité d’une molécule : il s’agit de trouver un composé chimique capable d’altérer une fonction cellulaire sans avoir d’indication préalable sur la nature moléculaire de sa (ou de ses) cible(s) biologique(s) (approche phenotype-based) [25]. Ainsi, un test phénotypique permet, par exemple, de sélectionner des molécules qui exercent une toxicité différentielle vis-à-vis de cellules cancéreuses et de cellules saines. Dans cette approche, on pourra également rechercher la perte, ou l’apparition, d’un biomarqueur cellulaire caractéristique du cancer, de l’inflammation, d’une infection virale ou bactérienne, ou de tout autre processus biologique d’intérêt, notamment grâce aux techniques de marquage en fluorescence [11, 26, 27]. Dans le cadre de travaux de recherche fondamentale sur le vivant, le processus d’identification de la (ou des) cible(s) des composés sélectionnés sur des modèles cellulaires sera motivé par les informations précieuses qu’il peut apporter, puisqu’à partir d’un phénotype altéré, le chercheur identifiera la protéine responsable du phénotype normal, à l’image du généticien qui enrichit les connaissances sur le génome en identifiant le gène associé à un phénotype mutant ou à une maladie génétique. Ainsi, un crible réalisé au CMBA-Grenoble sur plus de 30 000 molécules (Chimiothèque nationale, ChemBridge) par le Dr Laurence Lafanechère a conduit à la sélection de molécules capables de bloquer la dynamique du réseau de microtubules. Certaines des molécules ainsi caractérisées sont susceptibles d’être développées comme agents thérapeutiques en raison de leur capacité à bloquer la prolifération in vivo de lignées cellulaires cancéreuses. En outre, l’identification de la LIM kinase comme cible directe d’une de ces molécules (nommée Liminib) a permis la mise en évidence du rôle de cette enzyme dans la régulation du réseau de microtubules via la phosphorylation de la cofiline [3, 11]. Ces dernières années, le criblage phénotypique connaît une expansion considérable avec l’apparition de microscopes entièrement automatisés. Ceux-ci offrent la possibilité de visualiser et de quantifier des phénotypes cellulaires complexes à partir de l’acquisition et de l’analyse automatiques de multiples images de cellules traitées ou non par des molécules chimiques (méthodologie de high content screening (HCS)/high content analysis (HCA)) [26, 28–30, 39] (→). Le CMBA-Grenoble poursuit ainsi un effort de recherche et développement dans la mise au point d’une panoplie de tests phénotypiques qui peuvent être directement utilisés, soit pour la phase de criblage primaire de petites collections de molécules, soit pour la caractérisation du profil d’activité biologique de molécules d’intérêt [31]. Les essais disponibles, ou en cours de développement, au CMBA-Grenoble incluent notamment des tests de mesure de la viabilité cellulaire, de la dynamique du cytosquelette microtubulaire, de l’autophagie, de quantification de plaques d’adhésion focales, de localisation subcellulaire de facteurs de transcription, etc. (Figure 3). L’ouverture de cette activité à des équipes de recherche donne lieu à des collaborations dans divers domaines de recherches fondamentale et appliquée. Par exemple, le Dr E. Faudry (iRTSV, CEA-Grenoble) a développé un test en microplaques permettant l’évaluation de l’infection de cellules humaines par différentes souches de bactéries (Figure 3). Outre les microscopes et les logiciels d’analyse, toujours plus performants, les techniques de culture cellulaire évoluent vers des systèmes en 3-dimensions reconstituant autant que possible le microenvironnement cellulaire existant dans un organe, en reproduisant partiellement les contraintes mécaniques. Dans cette optique, le CMBA-Grenoble développe, en collaboration avec le Dr O. Filhol (iRTSV, CEA-Grenoble), l’acquisition et l’analyse automatisées de sphéroïdes par HCS/HCA pour l’évaluation de la toxicité de composés bioactifs (Figure 3).
(→) Voir la Synthèse de P.O. Vidalain et al., m/s n° 1, janvier 2015, page 98
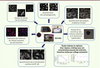 | Figure 3.
Développement des tests phénotypiques adaptés au HCS/HCA. Différents tests phénotypiques en plaque multipuits ont été développés pour analyser le réseau microtubulaire et les plaques d’adhésion focales, quantifier le nombre d’autophagosomes ou la translocation nucléaire de facteurs de transcription. Pour chaque test, des protocoles spécifiques d’acquisition et d’analyse automatiques d’images sont mis en place en vue d’une utilisation en routine. Certains de ces essais sont développés en collaboration avec des chercheurs de l’iRTSV (Dr E. Faudry pour le test d’infection de cellules humaines par des bactéries ; Dr O. Filhol pour le test de toxicité sur sphéroïdes, etc). Ces tests peuvent être utilisés dans le cadre de criblage de petites collections de molécules ou pour accélérer et approfondir la caractérisation des molécules touches (hits) préalablement sélectionnées par criblage robotisé. Dans ce dernier cas, la puissance de cette technologie réside dans la possibilité d’étudier les molécules bioactives en plusieurs réplicats, dose-réponse, cinétique ou encore sur cellules vivantes, dans un délai relativement court (quelques jours à quelques semaines). |
|
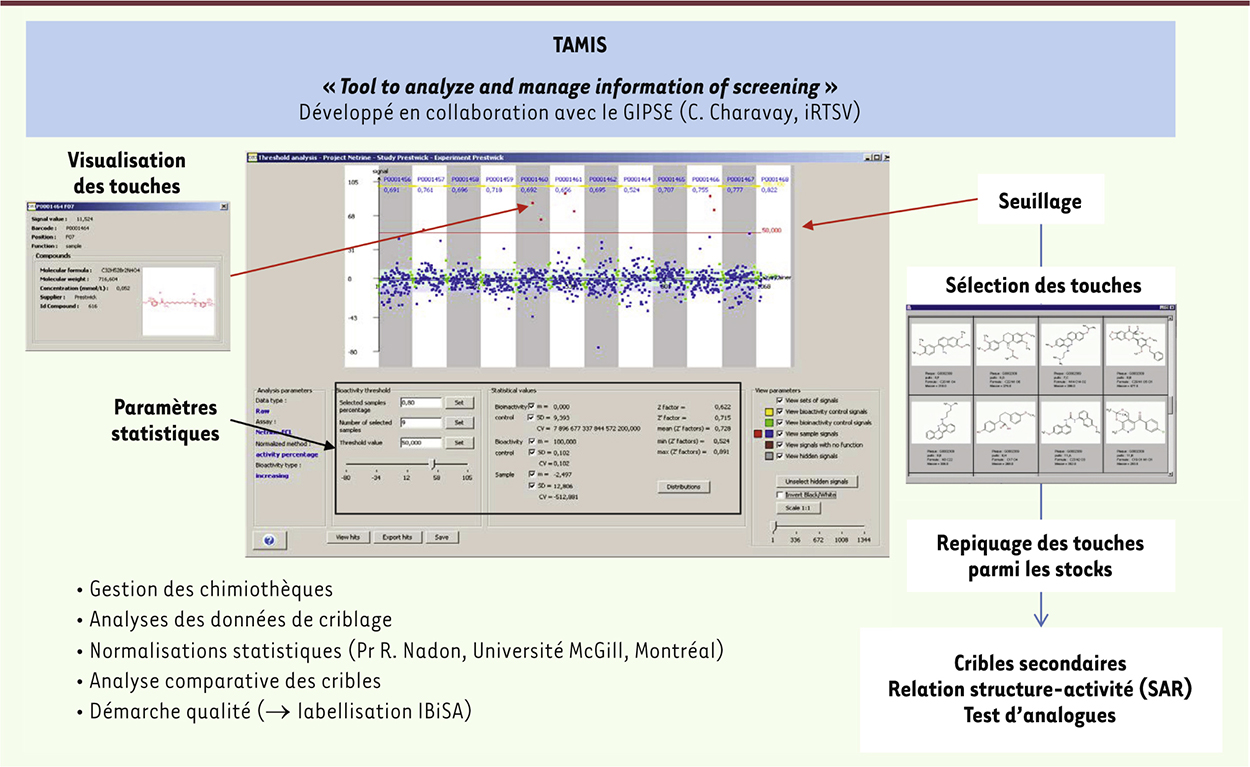
Gestion et analyse des données de criblage : le logiciel TAMIS La notion de haut débit, et a fortiori de haut contenu, suggère la production de grandes quantités de données qu’il faut pouvoir conserver et analyser avec toute la traçabilité nécessaire à l’identification fiable de nouvelles molécules bioactives. Dans cette optique, et afin de s’inscrire dans une démarche qualité, le logiciel TAMIS (Tool to analyse and manage information of screening) a été développé en étroite collaboration avec des informaticiens du GIPSE4 (iRTSV, CEA-Grenoble). Ce logiciel assure une gestion optimale des collections de molécules ainsi que l’analyse statistique des résultats de criblage (Figure 4). TAMIS permet d’une part de gérer les collections de molécules selon des concepts à la fois chimiques (structure, formule, etc.) et pratiques (généalogie des solutions de molécules) et, d’autre part, de sécuriser les données brutes issues des criblages dans une base de données à partir de laquelle sera effectuée l’analyse statistique permettant de sélectionner les molécules bioactives d’intérêt. TAMIS évolue continuellement, notamment pour l’intégration de méthodes d’analyses statistiques innovantes dédiées à l’analyse des résultats de HTS et HCS ; ces méthodes ont été développées en collaboration avec le Pr R. Nadon de l’Université de Montréal [32–35].
 | Figure 4.
Le logiciel TAMIS. Le logiciel TAMIS a été développé dès la mise en place de la plate-forme CMBA pour gérer les collections de molécules, analyser les données issues des criblages et sécuriser le lien entre bioactivité détectée et identité de la molécule. Ce logiciel intègre également différentes méthodes de normalisation statistique et assure un contrôle qualité des données (facteurs Z/Z’, détection de singularité, stacking function, etc.). |
|
Le criblage moléculaire, une stratégie plus que jamais pertinente dans le monde académique Les résultats de cette dernière décennie montrent que les activités de criblage moléculaire dans le monde académique offrent un véritable potentiel pour la recherche fondamentale, la recherche translationnelle et le rayonnement scientifique des laboratoires. Cette activité est en plein essor dans les universités américaines [28, 36]. En recherche, les stratégies de génétique chimique constituent un formidable potentiel pour l’étude de toutes sortes de processus physiologiques ou pathologiques avec des contraintes de biodisponibilité et de toxicité bien moindres - et largement surmontables - quand il est question de traiter des cellules en culture avec des sondes chimiques, que lorsqu’il s’agit d’administrer des médicaments à des patients. La valorisation de molécules bioactives en médicaments relève aussi en grande partie de la puissance publique puisqu’ils sont destinés à combattre des fléaux sociétaux ou environnementaux, dans un contexte où certains de ces secteurs sont délaissés par le monde industriel, soumis à une contrainte de rentabilité immédiate. Le véritable défi actuel pour le développement de molécules à visée thérapeutique est de réduire le taux d’attrition observé lors de la transition préclinique-clinique. Celui-ci est essentiellement dû à la toxicité ou à la médiocre biodisponibilité des molécules caractérisées. On peut espérer que le développement de tests phénotypiques sur des cellules vivantes permette d’anticiper la toxicité et l’efficacité des molécules dès le statut de touche, et ceci tout au long des programmes de développement conduisant à des molécules thérapeutiques. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Arrowsmith J. A decade of change . Nat Rev Drug Discov. 2012; ; 11 : :17.–18. 2.
Zheng W, Thorne N, McKew JC. Phenotypic screens as a renewed approach for drug discovery . Drug Discov Today. 2013; ; 18 : :1067.–1073. 3.
Prudent R, Vassal-Stermann E, Nguyen CH, et al. Pharmacological inhibition of LIM kinase stabilizes microtubules and inhibits neoplastic growth . Cancer Res. 2012; ; 72 : :4429.–4439. 4.
Moucadel V, Prudent R, Sautel CF, et al. Antitumoral activity of allosteric inhibitors of protein kinase CK2 . Oncotarget. 2011; ; 2 : :997.–1010. 5.
Prudent R, Moucadel V, Nguyen CH, et al. Antitumor activity of pyridocarbazole and benzopyridoindole derivatives that inhibit protein kinase CK2 . Cancer Res. 2010; ; 70 : :9865.–9874. 6.
Lopez-Ramos M, Prudent R, Moucadel V, et al. New potent dual inhibitors of CK2 and Pim kinases: discovery and structural insights . FASEB J. 2010; ; 24 : :3171.–3185. 7.
Prudent R, Hasenknopf B, Cochet C Les polyoxométallates : des inhibiteurs inorganiques de la protéine kinase CK2 . Med Sci (Paris). 2008; ; 24 : :1012.–1014. 8.
Prudent R, Moucadel V, Laudet B, et al. Identification of polyoxometalates as nanomolar noncompetitive inhibitors of protein kinase CK2 . Chem Biol. 2008; ; 15 : :683.–692. 9.
Prudent R, Cochet C. New protein kinase CK2 inhibitors: jumping out of the catalytic box . Chem Biol. 2009; ; 16 : :112.–120. 10.
Hibert MF. French/European academic compound library initiative . Drug Discov Today. 2009; ; 14 : :723.–725. 11.
Prudent R, Soleilhac E, Barette C, et al. Les criblages phénotypiques ou comment faire d’une pierre deux coups . Med Sci (Paris). 2013; ; 29 : :897.–905. 12.
Hoang TMN, Favier B, Valette A, et al. Benzo[e]pyridoindoles, novel inhibitors of the aurora kinases . Cell Cycle. 2009; ; 8 : :765.–772. 13.
Camara D, Bisanz C, Barette C, et al. Inhibition of p-aminobenzoate and folate syntheses in plants and apicomplexan parasites by natural product rubreserine . J Biol Chem. 2012; ; 287 : :22367.–22376. 14.
Boussouar A, Barette C, Nadon R, et al. Acacetin and chrysin, two polyphenolic compounds, alleviate telomeric position effect in human cells . Mol Ther Nucleic Acids. 2013; ; 2 : :e116.. 15.
Deininger M, Buchdunger E, Druker BJ. The development of imatinib as a therapeutic agent for chronic myeloid leukemia . Blood. 2005; ; 105 : :2640.–2653. 16.
Kawasumi M, Nghiem P. Chemical genetics: elucidating biological systems with small-molecule compounds . J Invest Dermatol. 2007; ; 127 : :1577.–1584. 17.
O’Connor CJ, Laraia L, Spring DR. Chemical genetics . Chem Soc Rev. 2011; ; 40 : :4332.–4345. 18.
McPherson A, Nguyen C, Cudney R, et al. The role of small molecule additives and chemical modification in protein crystallization . Cryst Growth Des. 2011; ; 11 : :1469.–1474. 19.
Laramas M, Pasquier D, Filhol O, et al. Nuclear localization of protein kinase CK2 catalytic subunit (CK2alpha) is associated with poor prognostic factors in human prostate cancer . Eur J Cancer. 2007; ; 43 : :928.–934. 20.
Giusiano S, Cochet C, Filhol O, et al. Protein kinase CK2alpha subunit over-expression correlates with metastatic risk in breast carcinomas: quantitative immunohistochemistry in tissue microarrays . Eur J Cancer. 2011; ; 47 : :792.–801. 21.
Buchou T, Cochet C. La protéine kinase CK2, une enzyme qui cultive la différence . Med Sci (Paris). 2003; ; 19 : :709.–716. 22.
Filhol O, Martiel JL, Cochet C. Protein kinase CK2: a new view of an old molecular complex . EMBO Rep. 2004; ; 5 : :351.–355. 23.
Laudet B, Barette C, Dulery V, et al. Structure-based design of small peptide inhibitors of protein kinase CK2 subunit interaction . Biochem J. 2007; ; 408 : :363.–373. 24.
Laudet B, Prudent R, Filhol O, et al. Des agents thérapeutiques ciblant des interactions protéine-protéine . Med Sci (Paris). 2007; ; 23 : :273.–278. 25.
Moffat JG, Rudolph J, Bailey D. Phenotypic screening in cancer drug discovery-past, present and future . Nat Rev Drug Discov. 2014; ; 13 : :588.–602. 26.
Brodin P, Del Nery, Elaine, Soleilhac E. Criblage phénotypique à haut contenu pour la chémobiologie et ses enjeux . Med Sci (Paris). 2015; ; 31 : :187.–196. 27.
Vassal E, Barette C, Fonrose X, et al. Miniaturization and validation of a sensitive multiparametric cell-based assay for the concomitant detection of microtubule-destabilizing and microtubule-stabilizing agents . J Biomol Screen. 2006; ; 11 : :377.–389. 28.
Eggert US, Mitchison TJ. Small molecule screening by imaging . Curr Opin Chem Biol. 2006; ; 10 : :232.–237. 29.
Korn K, Krausz E. Cell-based high-content screening of small-molecule libraries . Curr Opin Chem Biol. 2007; ; 11 : :503.–510. 30.
Soleilhac E, Nadon R, Lafanechere L. High-content screening for the discovery of pharmacological compounds: advantages, challenges and potential benefits of recent technological developments . Expert Opin Drug Discov. 2010; ; 5 : :135.–144. 31.
Martinez A, Soleilhac E, Barette C, et al. Novel synthetic pharmacophores inducing a stabilization of cellular microtubules . Curr Cancer Drug Targets. 2014; ; 15 : :2.–13. 32.
Lo E, Soleilhac E, Martinez A, et al. Intensity quantile estimation and mapping: a novel algorithm for the correction of image non-uniformity bias in HCS data . Bioinforma Oxf Engl. 2012; ; 28 : :2632.–2639. 33.
Murie C, Barette C, Lafanechere L, et al. Single assay-wide variance experimental (SAVE) design for high-throughput screening . Bioinforma Oxf Engl. 2013; ; 29 : :3067.–3072. 34.
Murie C, Barette C, Lafanechère L, et al. Control-plate regression (CPR) normalization for high-throughput screens with many active features . J Biomol Screen. 2014; ; 19 : :661.–671. 35.
Murie C, Barette C, Button J, et al. Improving detection of rare biological events in high-throughput screens . J Biomol Screen. 2014; ; 20 : :230.–241. 36.
Thomas CJ, Auld DS, Huang R, et al. The pilot phase of the NIH chemical genomics center . Curr Top Med Chem. 2009; ; 9 : :1181.–1193. 37.
Voisset C, Blondel M. Chémobiologie à l’happy hour : la levure comme modèle de criblage pharmacologique . Med Sci (Paris). 2014; ; 30 : :1161.–1168. 38.
Mahuteau-Betzer F. Chimiothèque Nationale : avancées et perspectives . Med Sci (Paris). 2015; ; 31 : :417.–422. 39.
Vidalain PO, Lucas-Hourani M, Helynck O, Tangy F, Munier-Lehmann H. Activation de la réponse innée antivirale par des inhibiteurs de la biosynthèse des pyrimidines : les surprises d’un criblage phénotypique . Med Sci (Paris). 2015; ; 31 : :98.–104. |