| |
| Med Sci (Paris). 2015 March; 31(3): 304–311. Published online 2015 April 8. doi: 10.1051/medsci/20153103016.Le génome intime… et en trois dimensions David Umlauf1* 1Université de Toulouse, université Paul Sabatier, laboratoire de biologie moléculaire des eucaryotes, CNRS, 118 route de Narbonne, 31000Toulouse, France |

Le noyau des cellules eucaryotes en interphase est une structure hautement compartimentalisée dans laquelle les chromosomes ne sont pas répartis au hasard, mais occupent chacun un volume délimité appelé territoire chromosomique (Figure 1A).
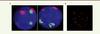 | Figure 1.
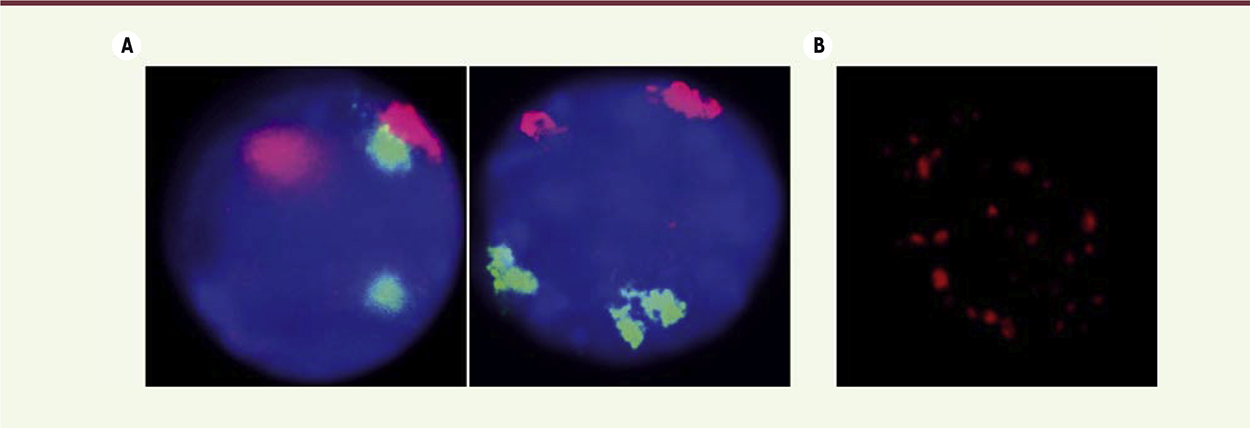
Le noyau des cellules eucaryotes en interphase est compartimentalisé. A. Noyaux de cellules érythroïdes (panneau de gauche) et de rein fœtal (panneau de droite) de souris colorés au DAPI (4’,6’-diamidino-2-phénylindole) (bleu), dans lesquels les chromosomes 7 (rouge) et 11 (vert) sont détectés avec un ensemble de sondes ADN-FISH. Les images ont été obtenues par microscopie à fluorescence standard (avec l’aimable autorisation de Lyubomira Chakalova et Peter Fraser, Babraham Institute, Cambridge, Royaume-Uni). Chaque chromosome occupe dans le noyau un espace défini appelé territoire chromosomique et, pour les cellules érythroïdes, on observe dans 45 % des cas une association préférentielle entre les chromosomes 7 et 11, qui n’existe que dans 12 % des cellules de rein fœtal (communication personnelle, Peter Fraser). B. Noyau de cellule HeLa dans lequel la protéine SC35 (serine/arginine-rich splicing factor 2) est détectée par immunofluorescence indirecte en microscopie confocale. La distribution de cette protéine impliquée dans l’épissage des ARN pré-messagers n’est pas diffuse, mais concentrée dans des sites appelés corps nucléaires. |
Il existe une corrélation entre la densité en gènes d’un chromosome et sa position radiale dans le noyau. Plus cette densité est élevée, plus le chromosome est localisé vers l’intérieur du noyau. De façon générale, cette tendance a perduré au cours de l’évolution. D’autre part, une association non aléatoire et préférentielle de certains chromosomes a été montrée, chez l’homme et la souris, dans un nombre restreint de lignées cellulaires (Figure 1A). Cependant, il ne semble pas que de tels profils puissent être généralisables à l’ensemble des chromosomes [1]. Parallèlement aux territoires chromosomiques, le noyau se structure en sous-domaines nucléaires fonctionnels, comme le nucléole ou les régions de la chromatine associées à la lamine. L’état de condensation de la chromatine dans ces environnements, ainsi que sa dynamique, varient et reflètent l’activité transcriptionnelle qui y prend place [2]. Enfin, de nombreuses régions regroupées sous le terme de corps nucléaires sont enrichies en facteurs impliqués dans les grandes fonctions du noyau (Figure 1B) [3]. Les travaux pionniers du groupe de Peter Cook ont montré, par exemple, que dans certains types cellulaires mammifères, la transcription des gènes codant pour les protéines n’est pas diffuse, mais prend place au sein d’un nombre restreint « d’usines de transcription » qui sont enrichies en ARN polymérases II actives [4]. Une conséquence fonctionnelle de ce type de structure est la formation d’associations préférentielles de gènes corégulés au sein d’usines contenant des facteurs de transcription communs. Ces associations peuvent être inter- ou intrachromosomiques, et créent des réseaux d’interactions transcriptionnelles [5, 6]. L’architecture nucléaire a été principalement étudiée grâce à l’utilisation de techniques de microscopie sur cellules fixées, comme l’hybridation in situ fluorescente (FISH) qui permet la visualisation d’un nombre limité de cibles ADN et ARN. Les progrès de l’imagerie sur cellules vivantes ont permis de mieux caractériser la dynamique des chromosomes ou l’activité transcriptionnelle de gènes rapporteurs, grâce notamment à l’utilisation de séquences bactériennes répétées et de protéines de fusion fluorescentes [7]. Cependant, ces approches ne renseignent nullement sur la structure précise des locus étudiés et présentent des limites inhérentes à leur nature, comme la limite de résolution de la lumière ou le seuil de fluorescence en dessous duquel l’observation n’est plus possible. La composition de la fibre chromatinienne et la caractérisation des protéines qui interagissent avec elle peuvent être déterminées par immunoprécipitation de chromatine (ChIP), une technique qui détecte les interactions directes ou indirectes d’une protéine avec l’ADN [8]. Couplée au séquençage à haut débit, l’immunoprécipitation de chromatine a permis d’établir, à l’échelle du génome, la distribution d’un grand nombre de protéines et de séquences régulatrices importantes pour l’expression des gènes (www.encodeproject.org/ENCODE/). Cette approche fournit des données d’une résolution supérieure à celle de l’imagerie, mais dans une seule dimension et sans qu’il soit possible d’inférer une structure tridimensionnelle (3D) pour une région ou un chromosome donnés. Au début des années 2000, l’émergence d’une nouvelle technique appelée capture de la conformation des chromosomes (3C) va permettre de franchir une nouvelle étape dans la caractérisation des interactions chromatiniennes [9]. Une décennie plus tard, les techniques dérivées du 3C ont dévoilé les principes d’organisation du génome à un niveau sans précédent et ont ouvert une nouvelle ère dans la compréhension de l’architecture nucléaire. Dans cette revue, nous décrirons les apports de la 3C et de ses principales variantes, et discuterons les différents niveaux d’organisation du génome observés chez les métazoaires. |
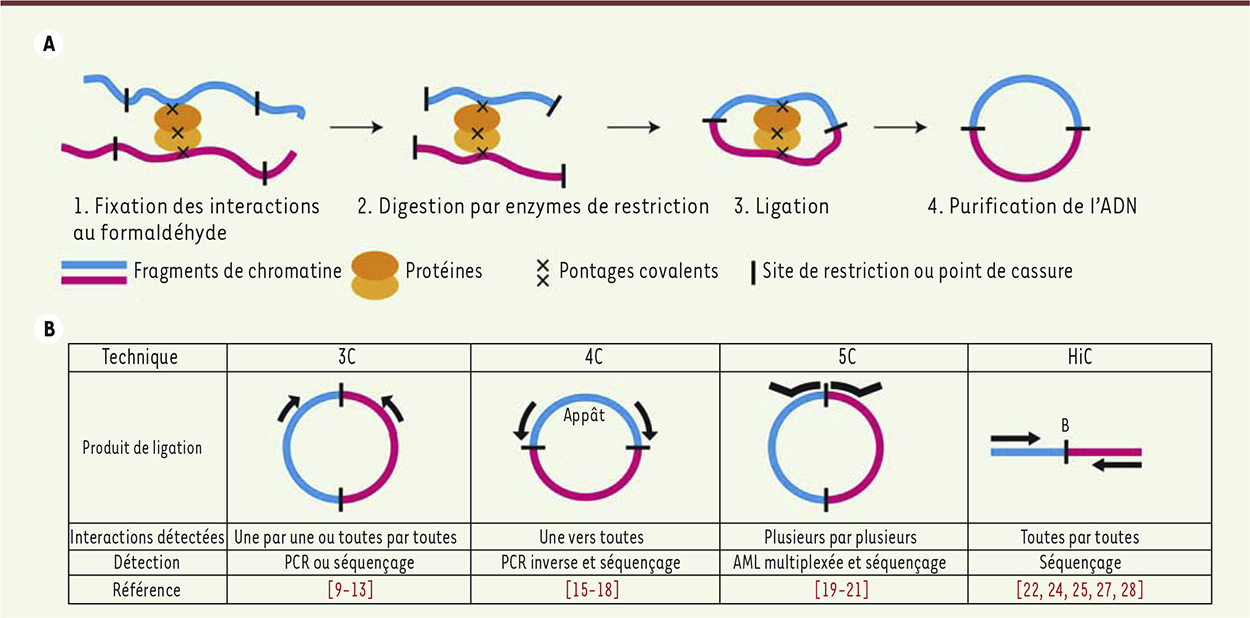
Capture de la conformation des chromosomes : molecular biology is your friendLa 3C permet de détecter la proximité spatiale de deux fragments de chromatine. Ces deux fragments peuvent être à des distances plus ou moins grandes sur le même chromosome, ou appartenir à des chromosomes différents. La 3C se divise en deux étapes principales : la génération des produits de ligation et leur détection (Figure 2A). Les cellules sont fixées au formaldéhyde, les noyaux isolés et la chromatine digérée par une enzyme de restriction, dont le choix dépend du locus à analyser. Les noyaux sont lysés et on procède à la ligation des fragments libérés en conditions diluées. L’ADN est ensuite purifié avant analyse.
 | Figure 2.
Capture de la conformation des chromosomes (3C) et ses dérivés. A. Génération des produits de ligation. Les cellules sont traitées au formaldéhyde de façon à fixer les interactions entre fragments de chromatine spatialement proches. Les noyaux sont isolés, et la chromatine est digérée par une enzyme de restriction avant ligation en conditions diluées, et purifiée. B. Principaux variants du 3C. Pour la 3C, l’analyse des produits de ligation s’effectue par PCR classique ou, comme décrit plus récemment, par séquençage à haut débit après sonication et sélection de la taille des produits [ 24]. L’analyse des interactions entre une région d’intérêt (appât) et le reste du génome par la technique de la 4C se fait par PCR inverse en utilisant des amorces spécifiques de l’appât. Pour la 5C, la librairie produite par la 3C est analysée par une amplification impliquant la ligation (AML). Un très grand nombre d’amorces peuvent être utilisées simultanément. Pour la HiC, un résidu biotinylé est introduit avant ligation afin de purifier sélectivement les fragments. L’ensemble des produits est séquencé à haut débit. D’après [ 14]. |
Connaissant la région génomique à analyser ainsi que la distribution des sites de restriction utilisés, il est possible de tester par PCR (polymerase chain reaction) ou PCR quantitative un évènement de ligation particulier entre deux régions (c’est-à-dire leur proximité spatiale) en utilisant des amorces situées de part et d’autre de la jonction de ligation [9, 10]. La résolution de la 3C, qui dépend du site de restriction choisi, est de l’ordre d’une centaine à quelques milliers de paires de bases. Les résultats obtenus rendent compte de la fréquence d’interaction des fragments testés. Sachant qu’il existe de nombreux contacts aléatoires, la spécificité des interactions détectées doit être contrôlée rigoureusement [11]. Initialement utilisée pour l’étude de la structure du chromosome III de levure, la 3C sera appliquée avec succès à de nombreux locus de mammifères [9, 12]. Pour la première fois, l’existence de boucles chromatiniennes spécifique du tissu, qui permettent de rapprocher physiquement gènes et éléments régulateurs distaux (comme les enhancers), est démontrée [12]. Le potentiel et la puissance de cette technique seront illustrés par la démonstration d’interactions interchromosomiques, expliquant la régulation coordonnée de certains gènes de cytokines [13]. Notons que les interactions détectées par la 3C peuvent être validées par microscopie en utilisant des sondes ADN-FISH, ce qui en fait deux techniques complémentaires, voire indissociables l’une de l’autre. |
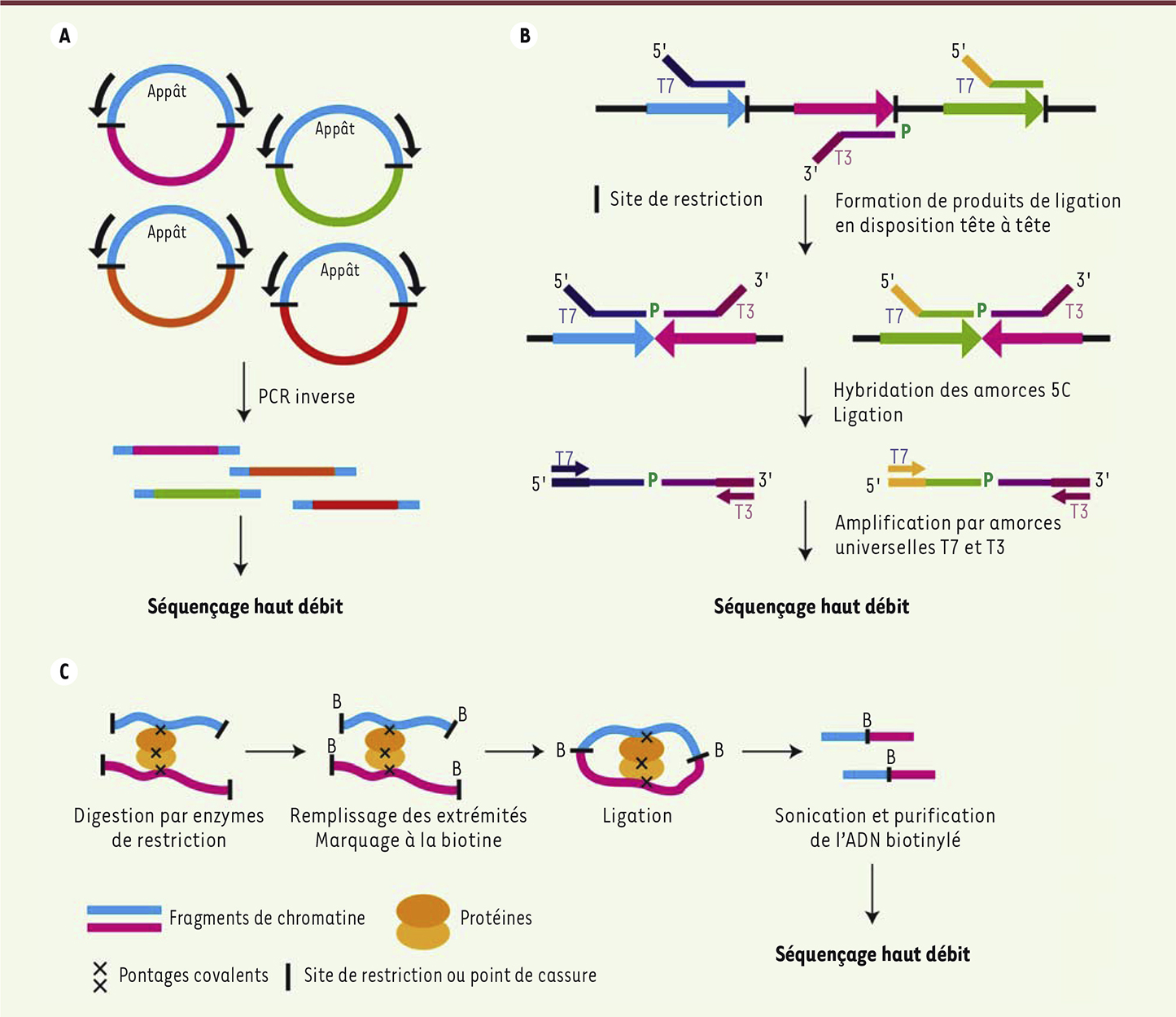
Les techniques dérivées de la 3C : zooming in and out of the genomeLa 3C s’avère être une technique de choix pour l’étude de la régulation de l’expression génique, puisqu’elle permet une analyse des interactions à grande distance opérant entre les gènes et l’ensemble de leurs séquences régulatrices. Cependant, elle peut vite s’avérer fastidieuse pour l’étude d’un grand nombre de régions. Récemment, de nombreuses techniques dérivées ont vu le jour et ont permis de systématiser la détection et l’analyse des produits de ligation (Figure 2B) [14]. Chromosome conformation capture on chip (4C) La chromosome conformation capture on chip (4C) permet de caractériser l’ensemble des interactions entre une séquence connue (ou séquence appât) et le reste du génome [ 15, 16]. Les cellules sont traitées de façon identique que pour la 3C. Cependant, la détection des produits de ligation se fait par PCR inverse en utilisant un couple d’amorces s’hybridant sur la séquence appât. La banque de produits PCR ainsi générée est soit hybridée sur puce ADN, soit séquencée à haut débit (Figure 3A). Plusieurs techniques 4C sont disponibles, chacune présentant de légères variations (voir pour exemples [ 17, 18]). Il est clair que pour une cellule donnée, la séquence appât n’interagit pas avec l’ensemble des régions détectées. Cependant, les résultats obtenus permettent de déterminer, à l’échelle d’une population de cellules, les régions qui interagissent préférentiellement ensemble. On a caractérisé par la 4C [ 6, 15, 16] des réseaux d’interactions fonctionnelles impliquant les gènes du locus de la β-globine, certains locus soumis à l’empreinte génomique, ainsi que des gènes corégulés par les mêmes facteurs de transcription.
 | Figure 3.
Détail des techniques 4C, 5C et HiC. A. Technique de la 4C. Lorsque la librairie 3C est générée, il y a formation d’un grand nombre de produits de ligation entre la séquence d’intérêt et le reste du génome. En utilisant des amorces spécifiques de l’appât, il est possible d’amplifier simultanément l’ensemble des interactions par PCR inverse. B. Amplification médiée par la ligation (AML). Dans la technique 5C, ce sont les produits de ligation tête à tête qui sont testés. L’amorce sens comporte une séquence unique qui correspond au brin sens de l’extrémité 3’ du site de restriction. Réciproquement, l’amorce antisens possède une séquence unique correspondant au brin antisens de l’extrémité 3’ du site de restriction. Elle est, de plus, la seule à être phosphorylée sur son extrémité 5’ de sorte que la ligation ne se fait qu’entre une amorce sens et antisens. Celles-ci possèdent respectivement sur leurs extrémités 5’ et 3’ un promoteur T7 et T3, ce qui permet l’amplification simultanée de toutes les interactions. C. Génération de produits de ligation biotinylés pour la HiC. Après digestion par une enzyme de restriction, il est nécessaire de réparer les extrémités cohésives générées et d’introduire un nucléotide biotinylé. Après ligation, les produits sont fragmentés par sonication et purifiés. |
Chromosome conformation capture carbon copy (5C) La 3C génère une librairie complexe de produits de ligation. La chromosome conformation capture carbon copy (5C) utilise cette librairie afin de déterminer simultanément un grand nombre d’interactions au sein de larges régions génomiques [ 19]. La technologie 5C repose sur une amplification médiée par ligation (AML) qui met en jeu des amorces s’hybridant sur le même brin ADN. Afin d’éviter la détection des fragments issus de digestions partielles ou d’un même fragment circularisé sur lui-même, seuls les produits tête à tête sont analysés avec des amorces alternées (Figure 3B). Les amorces sont liguées au niveau de la jonction de ligation et amplifiées en utilisant des amorces universelles T7 et T3. L’analyse par la 5C de régions couvrant 1 % du génome humain a permis de dégager certaines caractéristiques, jusqu’alors inconnues, des interactions entre sites d’initiation de la transcription et éléments régulateurs [ 20]. Parmi les observations les plus intéressantes, les éléments régulateurs établissent très rarement des boucles d’interaction avec les gènes qui sont à proximité, et leur distribution est asymétrique puisqu’ils se situent en moyenne à 120 kb en amont des sites d’initiation de la transcription. Chez la souris, la 5C a dévoilé, pour la première fois, l’organisation du centre d’inactivation du chromosome X en domaines s’associant topologiquement (voir plus loin) [ 21]. High throughput 3C : les principes de l’organisation du génome dévoilés La technique de l’ high throughput 3C (HiC) permet, de façon non biaisée, la détection de toutes les interactions qui ont lieu à l’échelle du génome [ 22]. La complexité de leur analyse bio-informatique est un véritable défi et leur modélisation fait appel à la physique des polymères [ 23]. Malgré quelques variations, la technique employée reste proche de la 3C (Figure 3C). Les premières expériences de HiC ont permis d’approcher l’organisation du génome humain à une échelle sans précédent. L’analyse de l’ensemble des matrices d’interactions confirme l’organisation des chromosomes en territoires et la disposition radiale en fonction de la densité en gènes décrite plus haut. Chaque chromosome peut être décomposé en deux types de blocs principaux qui sont, soit enrichis, soit appauvris en interactions (Figure 4B). Cela permet de définir un ensemble de compartiments avec une résolution d’environ 1 Mb, nommés arbitrairement A et B. De façon intéressante, les régions appartenant au même compartiment interagissent plus fréquemment ensemble, alors que des régions de type différent ont une probabilité d’interaction moindre. Cette observation est valable au niveau intra- et interchromosomique. Fait remarquable, ces compartiments peuvent être associés à des caractéristiques chromatiniennes qui sont reproductibles. Le compartiment A est corrélé à des régions riches en gènes, à une chromatine sensible à la DNase I et à des modifications post-traductionnelles des histones caractéristiques d’une chromatine active ; le compartiment B correspond à des régions de chromatine plus condensée. L’appartenance d’une région à l’un ou l’autre des compartiments semble être spécifique du tissu [22].
 | Figure 4.
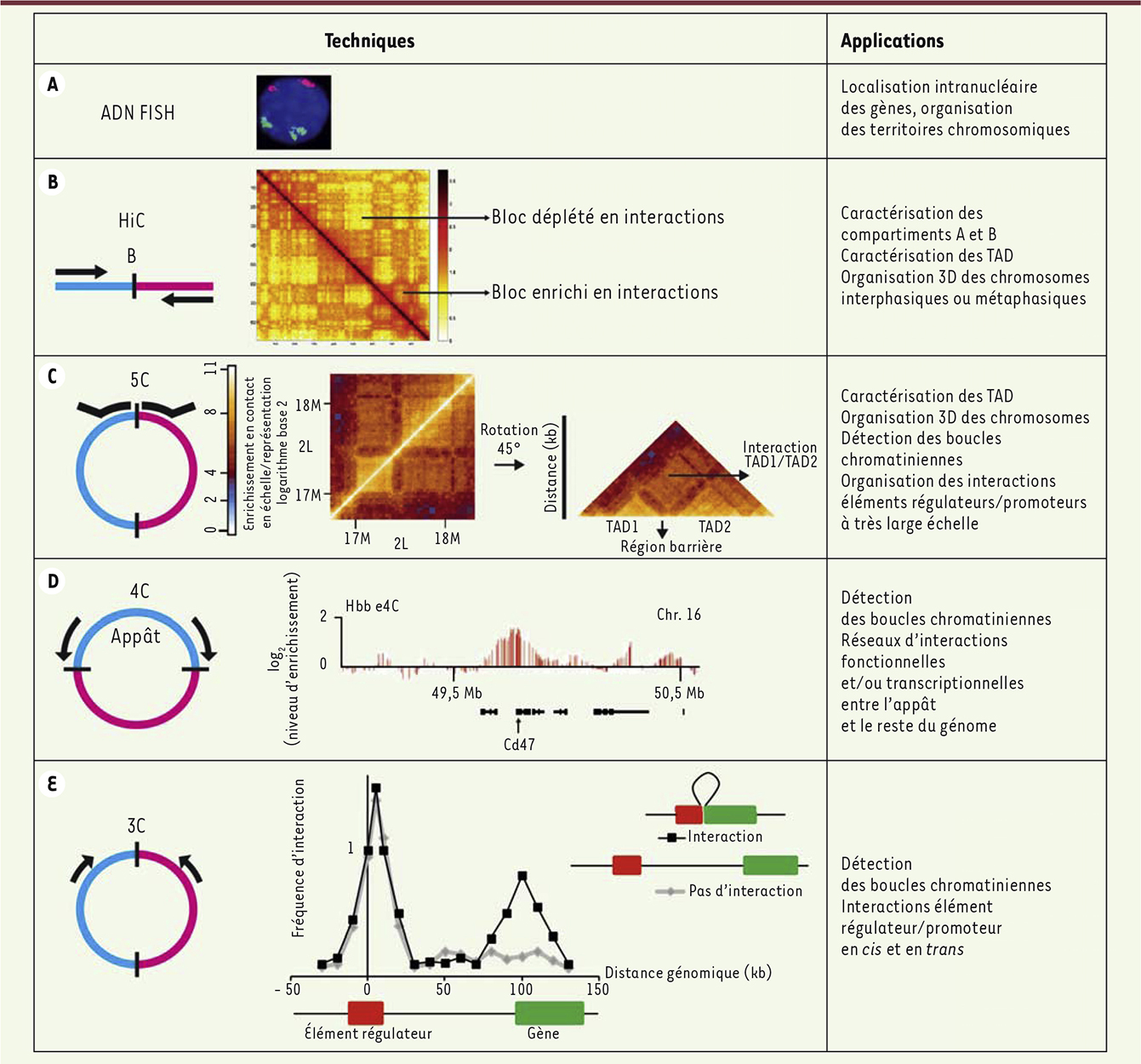
Les niveaux d’organisation du génome révélés par les techniques dérivées de la 3C. A. Les techniques habituelles de microscopie, comme l’ADN FISH, révèlent la position intranucléaire des gènes, de certaines portions de chromosomes ou la distribution des territoires chromosomiques, avec une résolution de quelques centaines de nanomètres. B. L’HiC permet de générer des matrices d’interactions à l’échelle du génome, représentées sous forme de matrices de corrélation associées à un code couleur. Ceci révèle des blocs de chromatine enrichis ou déplétés en interactions. Leur traitement mathématique permet de décomposer les chromosomes en compartiments A et B. Une résolution d’environ 1 Mb est suffisante pour reconstruire un modèle d’organisation 3D des chromosomes [ 31]. Les expériences de HiC les plus récentes permettent d’atteindre une résolution de détection d’interaction de 1 kb [ 32]. C. À une profondeur de séquençage suffisante, la HiC et la 5C permettent de caractériser des domaines s’associant topologiquement, ou TAD ( topologically associating domains). Ici, les interactions détectées sur une portion du bras gauche du chromosome 2 de drosophile dans des cellules embryonnaires sont montrées (avec l’aimable autorisation de Tom Sexton, IGBMC, Strasbourg, France). Pour une représentation simplifiée, la moitié de cette matrice est orientée à 45°. On observe des régions plus denses en contacts (triangles jaunes sur la diagonale) qui correspondent à des TAD, tandis que les zones carrées orangées indiquent des zones de contact entre TAD. La chute brutale des interactions détectée au niveau du TAD1 marque la présence d’une région barrière. D. La technique 4C révèle les interactions fonctionnelles entre la séquence appât et le reste du génome. Une interaction inter-chromosomique entre le gène Hbb ( hemoglobin β) du locus de la β-globine (chromosome 7) et le gène Cd47 (chromosome 16) dans des lignées érythroïdes de souris est montrée (reproduit avec la permission de Macmillan Publishers Ltd [ 6]). E.
La 3C permet la détection des interactions à longue distance (et donc des boucles chromatiniennes) entre deux séquences connues. Dans cet exemple théorique, la fréquence d’interaction d’un élément régulateur et d’un gène en fonction de la distance génomique est montrée. Elle est calculée à partir d’une succession de PCR effectuées en utilisant une amorce constante située dans l’élément régulateur (positionné arbitrairement à la position 0 sur l’axe des abcisses) et diverses amorces réparties le long de la région analysée. La fréquence d’interactions est toujours élevée entre l’amorce constante et les régions proches. Cette fréquence diminue rapidement en fonction de la distance génomique, sauf lorsqu’une interaction est détectée (courbe noire). |
La probabilité de contact entre deux régions, mesurée comme une fonction de la distance génomique les séparant, permet de construire une structure 3D moyenne du génome qui est représentative d’une population de cellules. Pour des distances génomiques situées entre 500 kb et 7 Mb, la loi de puissance qui régit cette probabilité est proche de celle obtenue par simulation pour un polymère replié de type fractal globule [22]. Ce repliement correspond à un état fortement condensé et non emmêlé du polymère. Sa trajectoire lui permet de remplir de façon optimale l’espace 3D sans jamais se croiser ni former de nœuds, ce qui donne à toute région la capacité de se déplier ou se replier avec facilité [23]. En augmentant la profondeur de séquençage, c’est-à-dire le nombre d’interactions détectées, et en utilisant des techniques telles que la 5C ou des variantes de l’HiC, il a été montré que les compartiments A et B sont constitués d’un assemblage de domaines topologiques (TAD, topologically associating domains) [21, 24, 25]. Conservés au cours de l’évolution, ils correspondent à des portions de fibres chromatiniennes à l’intérieur desquelles de très nombreuses interactions locales prennent place (Figure 4C). De façon remarquable, la majorité des TAD sont des structures relativement stables entre les différents types cellulaires [26]. En revanche, en utilisant une fenêtre d’observation de 20kb, on remarque que ce sont les interactions à l’intérieur d’un même TAD qui sont spécifiques du type cellulaire. De même, les données récentes de la littérature suggèrent l’existence de TAD spécifiques du tissu et qui sont dynamiques au cours du développement [26]. À la jonction entre deux TAD consécutifs existent des régions dites barrières, où une chute brutale des interactions est observée. Les régions barrières sont fréquemment liées par la protéine insulatrice CTCF (CCCTC-binding factor) [25]. Chez la drosophile, il a été montré que d’autres protéines insulatrices, comme Beaf-32 (boundary element-associated factor-32), CP190 (centrosome-associated protein 190) et chromator peuvent lier ces régions [24]. Chez l’homme et la souris, les régions barrières sont aussi fréquemment associées à des gènes domestiques, des gènes ARNt et des séquences de type SINE (short interspersed nuclear element). Cela suggère une activité transcriptionnelle élevée au niveau de ces régions, ce qui est corroboré par la présence de modifications post-traductionnelles des histones associées à une chromatine active [25]. On observe aussi une superposition entre les TAD et certaines marques épigénétiques de la chromatine. Cependant, dans des lignées cellulaires invalidées pour des enzymes qui mettent en place certaines de ces modifications, il n’y a pas de perturbations de la structure des TAD, ce qui suggère que l’étendue de ces marques est délimitée par la répartition du génome en TAD, et non l’inverse [21]. De façon surprenante, il a récemment été montré que les chromosomes métaphasiques perdent totalement cette organisation en compartiments et en TAD, ce qui implique des mécanismes distincts dans le repliement des chromosomes au cours de la mitose [27]. Single cell HiC : the sky is the limit Récemment, le groupe de Peter Fraser a réalisé le tour de force d’utiliser la technique HiC pour déterminer les interactions génomiques prenant place dans le noyau d’une seule cellule [ 28]. À la différence de l’approche classique, toutes les étapes sont réalisées dans le noyau des cellules qui sont ensuite placées individuellement dans des tubes. Après lyse cellulaire, les produits de ligation sont purifiés, puis l’ADN séquencé à haut-débit. Les résultats obtenus confirment la validité des observations à l’échelle d’une population de cellules. La loi de puissance qui décrit la probabilité de contact entre deux régions est la même que pour une HiC standard. On note aussi une stabilité et une invariabilité de la localisation des TAD que l’on peut aligner entre les différentes cellules. Ces données suggèrent que les contacts au sein d’un même TAD sont conservés d’une cellule à l’autre. Cependant, les contacts entre les différents TAD d’un même chromosome sont très différents entre cellules, ce qui indique une grande variabilité dans la façon dont un chromosome peut se replier. Les données de la single cell HiC peuvent être exploitées pour modéliser ces différences de repliement (Figure 5). Appliqué au chromosome X, on obtient pour une cellule un grand nombre de conformations possibles, toutes similaires entre elles. Cependant, ces conformations seront très différentes de celles obtenues pour une autre cellule individuelle. Ceci rend compte des différences de contacts entre les TAD. Si on corrèle ensuite ces modèles aux interactions interchromosomiques détectées, on observe que les régions situées à la surface du territoire chromosomique sont celles dans lesquelles on détecte le plus d’interactions interchromosomiques. De façon analogue à ce que montrait la HiC, elles sont enrichies en modifications d’histones associées à une chromatine active [22, 29]. Ces résultats sont en accord avec des données de microscopie qui montrent une localisation préférentielle des régions actives, ou potentiellement actives, à la surface de leurs territoires chromosomiques respectifs [5].
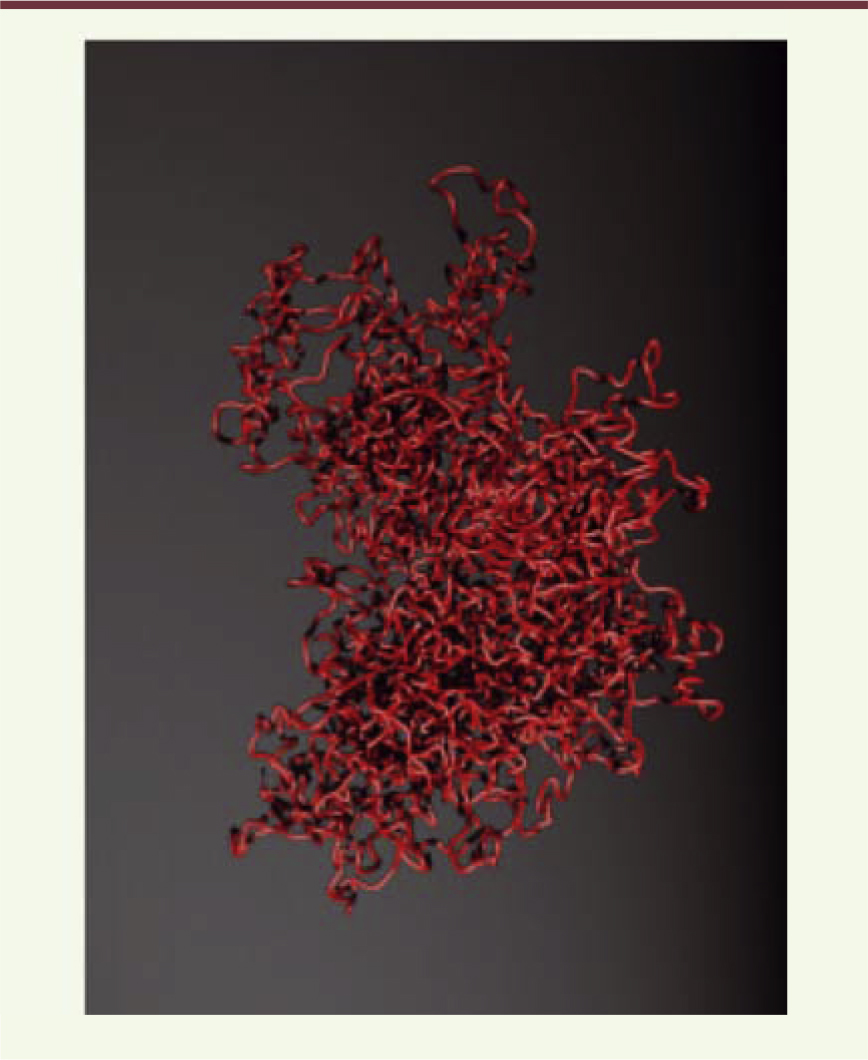
 | Figure 5.
Les données HiC permettent de modéliser le génome. Structure possible d’un chromosome X de souris prédite à partir de données HiC obtenues sur cellule unique (avec l’aimable autorisation de Takashi Nagano, Tim Stevens et Peter Fraser, Royaume-Uni). |
|
Grâce aux techniques de type HiC, la structure des territoires chromosomiques et les interactions qui prennent place à l’intérieur des différents niveaux d’organisation que nous venons de décrire peuvent être caractérisées avec précision. L’organisation du génome semble en majeure partie être inhérente aux propriétés de la fibre chromatinienne elle-même. Un exemple en est la stabilité et l’invariabilité de la majorité des TAD. Dès lors, comment expliquer la spécificité des programmes transcriptionnels mis en place au cours du développement et de la différenciation cellulaire ? Premièrement, l’existence de TAD spécifiques du stade de développement et/ou du type cellulaire a été suggérée [26]. Un autre élément de réponse réside dans la variabilité des interactions qui prennent place au sein d’un même TAD et entre les TAD (illustré par la grande diversité des repliements chromosomiques existant entre cellules). Un des futurs défis sera de déterminer si c’est ce type d’organisation nucléaire qui gouverne l’expression des gènes, ou si elle n’en est simplement qu’une conséquence. De même, l’étude de la dynamique de ces interactions, ainsi que la caractérisation des acteurs impliqués, seront tout aussi importantes. Les données récentes de la littérature ont confirmé l’existence d’un nucléosquelette constitué de protéines de structure et de protéines moteurs jouant un rôle clé dans l’architecture nucléaire et, notamment, dans l’expression des gènes [30]. Il semble qu’un véritable réseau d’interactions protéiques distribué du noyau jusqu’à la membrane plasmique soit capable d’intégrer les signaux reçus par la cellule afin de produire une réponse transcriptionnelle adaptée. Comprendre l’impact de ce nucléosquelette sur la plasticité du génome constitue d’ores et déjà l’ère post-HiC. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Nous tenons tout particulièrement à remercier Kerstin Bystricky, Patrice Vitali et Sylvain Egloff (LBME, Toulouse, France) pour leur lecture attentive du manuscrit, Peter Fraser et Lyubomira Chakalova (Babraham Institute Cambridge, Royaume-Uni) pour les images présentées en Figure 1 ainsi que Thomas Sexton (IGBMC, Strasbourg, France), Takashi Nagano (Babraham Institute Cambridge, Royaume-Uni) et Tim Stevens (department of biochemistry, university of Cambridge, Royaume-Uni) pour les images présentées dans les Figures 4 et 5. David Umlauf est financé par l’université Paul Sabatier de Toulouse (France).
|
1.
Cremer T, Cremer M. Chromosome territories . Cold Spring Harb Perspect Biol. 2010; ; 2 : :a003889.. 2.
Lanctot C, Cheutin T, Cremer M et al. Dynamic genome architecture in the nuclear space: regulation of gene expression in three dimensions . Nat Rev Genet. 2007; ; 8 : :104.–115. 3.
Dundr M. Nuclear bodies: multifunctional companions of the genome . Curr Opin Cell Biol. 2012; ; 24 : :415.–422. 4.
Iborra FJ, Pombo A, Jackson DA, Cook PR. Active RNA polymerases are localized within discrete transcription factories in human nuclei . J Cell Sci. 1996; ; 109 : :1427.–1436. 5.
Osborne CS, Chakalova L, Brown KE et al. Active genes dynamically colocalize to shared sites of ongoing transcription . Nat Genet. 2004; ; 36 : :1065.–1071. 6.
Schoenfelder S, Sexton T, Chakalova L et al. Preferential associations between co-regulated genes reveal a transcriptional interactome in erythroid cells . Nat Genet. 2010; ; 42 : :53.–61. 7.
Darzacq X, Yao J, Larson DR et al. Imaging transcription in living cells . Annu Rev Biophys. 2009; ; 38 : :173.–196. 8.
Furey TS. ChIP-seq and beyond: new and improved methodologies to detect and characterize protein-DNA interactions . Nat Rev Genet. 2012; ; 13 : :840.–852. 9.
Dekker J, Rippe K, Dekker M, Kleckner N. Capturing chromosome conformation . Science. 2002; ; 295 : :1306.–1311. 10.
Hagege H, Klous P, Braem C et al. Quantitative analysis of chromosome conformation capture assays (3C-qPCR) . Nat Protoc. 2007; ; 2 : :1722.–1733. 11.
Dekker J. The three Cs of chromosome conformation capture: controls, controls, controls . Nat Methods. 2006; ; 3 : :17.–21. 12.
Tolhuis B, Palstra RJ, Splinter E et al. Looping and interaction between hypersensitive sites in the active beta-globin locus . Mol Cell. 2002; ; 10 : :1453.–1465. 13.
Spilianakis CG, Lalioti MD, Town T et al. Interchromosomal associations between alternatively expressed loci . Nature. 2005; ; 435 : :637.–645. 14.
Dekker J, Marti-Renom MA, Mirny LA. Exploring the three-dimensional organization of genomes: interpreting chromatin interaction data . Nat Rev Genet. 2013; ; 14 : :390.–403. 15.
Simonis M, Klous P, Splinter E et al. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C) . Nat Genet. 2006; ; 38 : :1348.–1354. 16.
Zhao Z, Tavoosidana G, Sjolinder M et al. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions . Nat Genet. 2006; ; 38 : :1341.–1347. 17.
Sexton T, Kurukuti S, Mitchell JA et al. Sensitive detection of chromatin coassociations using enhanced chromosome conformation capture on chip . Nat Protoc. 2012; ; 7 : :1335.–1350. 18.
Van de Werken HJ, Landan G, Holwerda SJ et al. Robust 4C-seq data analysis to screen for regulatory DNA interactions . Nat Methods. 2012; ; 9 : :969.–972. 19.
Dostie J, Richmond TA, Arnaout RA et al. Chromosome conformation capture carbon copy (5C): a massively parallel solution for mapping interactions between genomic elements . Genome Res. 2006; ; 16 : :1299.–1309. 20.
Sanyal A, Lajoie BR, Jain G, Dekker J. The long-range interaction landscape of gene promoters . Nature. 2012; ; 489 : :109.–113. 21.
Nora EP, Lajoie BR, Schulz EG et al. Spatial partitioning of the regulatory landscape of the X-inactivation centre . Nature. 2012; ; 485 : :381.–385. 22.
Lieberman-Aiden E, van Berkum NL, Williams L et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome . Science. 2009; ; 326 : :289.–293. 23.
Mirny LA. The fractal globule as a model of chromatin architecture in the cell . Chromosome Res. 2011; ; 19 : :37.–51. 24.
Sexton T, Yaffe E, Kenigsberg E et al. Three-dimensional folding and functional organization principles of the Drosophila genome . Cell. 2012; ; 148 : :458.–472. 25.
Dixon JR, Selvaraj S, Yue F et al. Topological domains in mammalian genomes identified by analysis of chromatin interactions . Nature. 2012; ; 485 : :376.–380. 26.
Noordermeer D, Leleu M, Splinter E et al. The dynamic architecture of Hox gene clusters . Science. 2011; ; 334 : :222.–225. 27.
Naumova N, Imakaev M, Fudenberg G et al. Organization of the mitotic chromosome . Science. 2013; ; 342 : :948.–953. 28.
Nagano T, Lubling Y, Stevens TJ et al. Single-cell Hi-C reveals cell-to-cell variability in chromosome structure . Nature. 2013; ; 502 : :59.–64. 29.
Yaffe E, Tanay A. Probabilistic modeling of Hi-C contact maps eliminates systematic biases to characterize global chromosomal architecture . Nat Genet. 2011; ; 43 : :1059.–1065. 30.
Simon DN, Wilson KL. The nucleoskeleton as a genome-associated dynamic network of networks . Nat Rev Mol Cell Biol. 2011; ; 12 : :695.–708. 31.
Marti-Renom MA, Mirny LA. Bridging the resolution gap in structural modeling of 3D genome organization . PloS Comput Biol. 2011; ; 7 : :e1002125.. 32.
Rao SSP, Huntley MH, Durand NC et al. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping . Cell. 2014; ; 159 : :1.–16. |