| |
| Med Sci (Paris). 2014 November; 30(11): 937–940. Published online 2014 November 10. doi: 10.1051/medsci/20143011002.La chromatine naissante révélée Constance Alabert1* 1Biotech Research and Innovation Centre (BRIC), Laboratoire d'Anja Groth, Université de Copenhague, Ole Maaløes Vej 5, 2200Copenhague, Danemark MeSH keywords: Acides aminés, analyse, Biotinylation, Cellules cultivées, Chromatine, composition chimique, isolement et purification, ultrastructure, Protéines chromosomiques nonhistones, physiologie, Chromosomes, Réplication de l'ADN, Nucléotides désoxyuridyliques, Histone, Humains, Marquage isotopique, méthodes, Spectrométrie de masse, Nucléosomes, Phase S |
Duplication des chromosomes et maintien de l’identité cellulaire La séquence d’ADN contient l’ensemble de l’information génétique nécessaire au développement et au maintien des organismes multicellulaires. Si la quasi-totalité des cellules d’un organisme contiennent une copie intégrale de son génome, les gènes sont en revanche différentiellement exprimés en fonction du type cellulaire de manière à définir l’identité et la fonction biologique de chaque cellule. L’état transcriptionnel d’un gène dépend des facteurs de transcription et des machineries transcriptionnelles, mais aussi de multiples constituants de la chromatine, dits épigénétiques, qui participent à la régulation de l’expression génique et à sa transmission au cours des divisions cellulaires. Parmi ces constituants, on compte les nucléosomes, leur densité et leur positionnement, leur composition (par exemple l’incorporation de variants d’histone) ou leurs modifications post-traductionnelles (par exemple les marques d’histone). L’addition de groupes chimiques sur l’ADN, tels que la méthylation, compte aussi parmi les modifications épigénétiques qui modulent l’expression génique. On doit également ajouter à cette liste les protéines et complexes de protéines qui remodèlent les nucléosomes et déposent ou effacent les marques d’histone ou d’ADN, ou sont spécifiquement recrutés par elles. Le maintien de l’identité cellulaire est essentiel au développement et au renouvellement tissulaire normal chez les organismes métazoaires, alors que son altération est déterminante dans le processus de tumorigenèse. En conséquence, chaque cellule doit répliquer avec fidélité non seulement la séquence de son génome, mais aussi la composition et la structure de son épigénome. La molécule d’ADN enroulée autour d’un octamère d’histones forme le nucléosome (Figure 1). À chaque phase S du cycle cellulaire, les nucléosomes sont désassemblés à l’avant de la fourche de réplication et réassemblés rapidement derrière la fourche sur l’ADN nouvellement synthétisé [
1–
3,
16]. La densité des nucléosomes est maintenue par l’addition de nouvelles histones et, en 15 minutes, la résistance aux nucléases de la chromatine nouvellement assemblée est identique à celle du reste de la chromatine, ce qui suggère une restauration de la chromatine rapide et étroitement liée au replisome [
4–
6]. Plusieurs enzymes (dont l’enzyme qui restaure la méthylation de l’ADN) ainsi que des facteurs d’assemblage et de remodelage de la chromatine ont été détectés au niveau des fourches de réplication [
7]. Cependant, l’intégralité de l’information épigénétique ne semble pas être restaurée de cette façon. En effet, d’autres facteurs pourraient être recrutés après le passage de la fourche, sur la chromatine nouvellement répliquée [
8]. De plus, la restauration de certaines marques d’histones peut nécessiter jusqu’à plusieurs heures [
9]. Par ailleurs, il est important de souligner que les mécanismes permettant la restauration de la structure chromatinienne n’ont pu être explorés que pour un nombre limité de facteurs [7]. Pour remédier à cette lacune et dans le but de mieux comprendre comment l’ensemble des composants épigénétiques sont transmis au cours de la réplication, nous avons développé une nouvelle approche qui permet d’explorer la composition de la chromatine nouvellement répliquée.
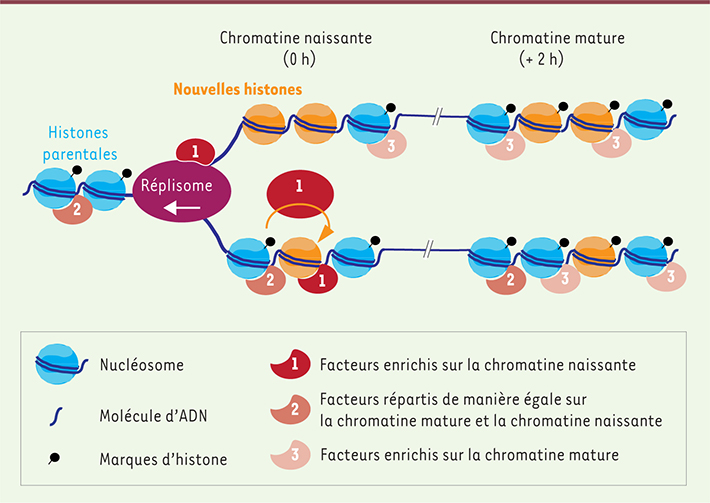
 | Figure 1.
Facteurs impliqués dans la réplication chromatinienne isolés par NCC. Pour ces expériences de NCC, les même régions chromatiniennes sont isolées à deux moments différents : immédiatement après leur réplication (chromatine naissante), ou deux heures plus tard (chromatine mature). Les trois catégories de facteurs isolés sont représentées. |
|
La technique de capture de la chromatine naissante (NCC) : un outil d’exploration moléculaire Dans un effort d’élucidation des mécanismes de réplication de la chromatine, trois groupes de recherche ont récemment développé des méthodes originales visant à isoler biochimiquement les facteurs présents aux fourches de réplication ou sur la chromatine naissante. Toutes ces méthodes se fondent sur le marquage in vivo de l’ADN nouvellement répliqué par un désoxyribonucléotide modifié. Après une fixation au formaldéhyde (permettant la fixation des liaisons protéine-protéine et ADN-protéine), l’ADN nouvellement répliqué et les facteurs chromatiniens qui y sont associés sont isolés via les nucléotides modifiés (voir ci-dessous) et identifiés par spectrométrie de masse. Les protocoles d’iPOND (isolation of proteins on nascent DNA) [
10] et de Dm-ChP (DNA mediated chromatin pull-down) [
11] utilisent un analogue du dTTP, l’EdU. Une réaction chimique sur la chromatine fixée permet d’attacher une étiquette de biotine à l’EdU afin de purifier la chromatine nouvellement répliquée. Dans l’approche NCC [
12], le nucléotide modifié utilisé (dUTP) est déjà fusionné à l’étiquette biotine. Ces méthodes de marquage de l’ADN ont déjà été utilisées pour étudier la réplication [
13]. Un désavantage de l’utilisation de la biotine-dUTP est qu’elle est incorporée dans les cellules par choc osmotique. En revanche, dans le protocole NCC, la fusion de l’étiquette biotine au nucléotide a l’avantage d’éviter le bruit de fond généré par la réaction chimique d’attache de l’étiquette sur l’EdU dans la chromatine fixée, nécessaire dans les deux autres protocoles. Outre le choix du système de marquage, la méthode de NCC a été spécifiquement élaborée et optimisée dans le but d’obtenir une sensibilité maximale afin d’obtenir l’image la plus fidèle possible de la composition de la chromatine naissante. Plusieurs paramètres permetttent d’augmenter la sensibilité de cette approche : (1) les cellules humaines sont cultivées en suspension et sont synchronisées en milieu de phase S afin qu’une majorité d’entre elles incorporent la biotine-dUTP. La synchronisation permet aussi d’homogénéiser le type de régions chromatiniennes marquées ; (2) les conditions de fixation au formaldéhyde sont deux fois plus élevées que dans les deux autres protocoles ; (3) deux séries consécutives de purification de la chromatine sont exécutées ; (4) la composition des régions chromatiniennes naissantes analysées est comparée à celle de ces mêmes régions, deux heures plus tard (chromatine mature) (Figure 1) [12]. Pour cela, la méthode NCC a été combinée à la méthode SILAC (stable isotope labeled aminoacids in cell culture, [
17]), ce qui permet de quantifier de façon relative et précise les protéines contenues dans les deux conditions par spectrométrie de masse. En parallèle, pour améliorer le ratio signal/bruit de fond pour la spectrométrie de masse, les conditions de lavage ont été mises au point afin d’être les plus stringentes possibles. Le protocole de NCC, ainsi que la conception des expériences, sont donc optimisés pour étudier la chromatine naissante et les processus de restauration de la chromatine. |
Près de 4 000 facteurs identifiés Le NCC a permis d’identifier 3 995 facteurs sur la chromatine naissante et mature. Ces facteurs sont répartis en trois catégories (Figure 1) : (1) les facteurs enrichis sur la chromatine naissante, comme les facteurs de réplication de l’ADN. Cette catégorie englobe 32 % des protéines identifiées, contient les facteurs impliqués dans les processus de restauration de la chromatine couplée au replisome (comme les facteurs d’assemblage des nucléosomes et de la méthylation de l’ADN, ou qui ont lieu sur la chromatine naissante ; (2) les facteurs répartis de manière égale sur la chromatine naissante et mature (56 %). Cette catégorie contient les composants de base de la chromatine comme les histones, et les facteurs n’ayant pas de fonction associée à la réplication, ce qui est le cas de la plupart des facteurs de remodelage de la chromatine ; (3) les facteurs présents en plus grand nombre sur la chromatine mature (12 %). Cette dernière catégorie peut inclure des composants de la chromatine qui nécessitent d’autres évènements préliminaires pour être recrutés. Par exemple, l’histone H1, qui requiert la déacétylation des histones nouvellement synthétisées pour être incorporée. Cette analyse fournit donc une ressource unique pour comprendre comment est restaurée l’information épigénétique. Cependant, la raison et les déterminants moléculaires responsables de cette spécificité de recrutement restent à élucider pour la majorité de ces facteurs. |
Filtrage bio-informatique afin d’augmenter la valeur prédictive des résultats générés par NCC Bien que le protocole de NCC ait été optimisé pour limiter au maximum le bruit de fond inhérent aux analyses à grande échelle de spectrométrie de masse [
14], une partie des 3 995 facteurs identifiés sont associés à la chromatine de manière non spécifique. Le bruit de fond représente un réel obstacle pour déterminer si les facteurs dont la fonction est à ce jour inconnue jouent un rôle dans la duplication de la chromatine. Nous avons donc utilisé une méthode de filtrage bio-informatique conçue dans le laboratoire de Juri Rappsilber (Université d’Edimbourg et de Berlin). Pour développer cette méthode, plusieurs dizaines d’extraits de chromatine ont été analysés par spectrométrie de masse en utilisant différents types de cellules, à différentes phases du cycle cellulaire et en utilisant différents protocoles d’extraction de la chromatine. Ces données ont permis de calculer un facteur de probabilité d’association à la chromatine (ICP) pour 7 635 protéines [14]. Les protéines ayant une liaison forte à l’ADN (comme les histones) ont un ICP plus élevé que les protéines dont l’association avec la fibre chromatinienne est plus faible (comme les enzymes de remodelage). En conséquence, cette procédure peut être utilisée pour exclure les protéines qui s’associent à la chromatine de manière artéfactuelle. En assignant un ICP à chaque facteur identifié dans notre analyse de NCC, nous avons pu affiner la liste de nouveaux facteurs putatifs de réplication ou de maturation de la chromatine (Figure 2). La fiabilité de cette méthode de filtration bio-informatique a été testée [14] et validée pour une dizaine de nouveaux facteurs (sélectionnés ou exclus) par microscopie [12]. En outre, la pertinence et l’efficacité de ces méthodes combinées (NCC et ICP) ont pu être démontrées par l’identification d’un nouveau facteur de réplication FAM111A (family with sequence similarity 111, member A). La liste de nouveaux facteurs obtenus par ces méthodes combinées devrait donc constituer une ressource majeure pour éclairer et accélérer la recherche sur la restauration de l’information épigénétique.
 | Figure 2.
Dynamique d’association des protéines identifiées par NCC. Quelques exemples de facteurs connus sont mentionnés à gauche. Le nombre de facteurs dont la fonction est inconnue et qui ont été sélectionnés comme potentiellement intéressants par NCC et ICP est indiqué en blanc. CAF-1 : chromatin assembly factor 1 ; DNMT1/3a : DNA methyltransferase 1/3a ; ESCO2 : acetyltransferase ; PRC1 : Polycomb repressive complex 1 ; H3 : histone H3 ; H2A : histone H2A ; H2A.Z : histone variant H2A.Z (adaptée de [ 12]). |
|
Le NCC, de même que l’iPOND et le Dm-ChP, ne permettent pas de déterminer si les facteurs identifiés sont requis pour dupliquer toutes les régions chromatiniennes ou seulement certaines d’entre elles. En effet, la chromatine isolée provient de différents types de chromatine répliquée. Il faudrait pouvoir réaliser un NCC sur un type unique de chromatine ou une région chromosomique spécifique. Si le développement d’un NCC spécifique d’un locus demande un développement technique certain, réduire les temps de marquage de l’ADN et/ou utiliser une synchronisation plus fine du cycle cellulaire seraient des moyens simples et rapides de pouvoir cibler des grands types de chromatine qui ne sont pas répliqués au même moment au cours du cycle cellulaire. L’utilisation de la technique de NCC s’est limitée jusqu’à maintenant à une partie de la composition chromatinienne. En effet, la méthylation de l’ADN et les ARN non codants sont d’autres constituants de l’information épigénétique qui n’ont pas été encore analysés. Or, le matériel isolé par NCC contient ces informations. Un simple couplage du NCC avec des techniques déjà existantes, comme les techniques d’analyse du transcriptome ou du méthylome, permettrait de compléter notre compréhension de la réplication de la chromatine. |
Ce travail contient la liste la plus exhaustive de protéines impliquées ou affectées par la duplication des chromosomes. Il est aussi important de noter que plusieurs protéines identifiées par NCC sont liées à des maladies comme le cancer. Par exemple, la mutation du gène codant pour la protéine FAM111A a récemment été associée au syndrome de petite taille sévère dit de Kenny Caffey [
15]. Ainsi, outre leur importance fondamentale pour la compréhension de la réplication de la chromatine, la méthode de NCC et ses résultats offrent un outil et une ressource précieuse pour comprendre le rôle de protéines impliquées dans des pathologies. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Je voudrais remercier Itys Comet pour la relecture critique de ce manuscrit. La recherche dans le laboratoire d’Anja Groth est soutenue par l’ERC starting grant (ERC2011StG no. 281,765), la fondation Lundbeck, la sociéte danoise du cancer, la fondation nationale danoise de recherche (DNRF82), le conseil médical de recherche, la fondation Novo Nordisk et le FP7 Marie Curie actions ITN nucleosome4D. La recherche dans le laboratoire de Juri Rappsilber est soutenue par le Wellcome Trust (084229, 077707, 092076, 091020). C.A a bénéficié de bourses post doctorales EMBO, HFSP, du conseil médical de recherche danois et de l’ERC2011StG.
|
1.
McKnight
SL
,
Miller
OL, Jr
. Electron microscopic analysis of chromatin replication in the cellular blastoderm Drosophila melanogaster embryo . Cell.
1977; ; 12 : :795.–804. 2.
Sogo
JM
,
Stahl
H
,
Koller
T
,
Knippers
R
. Structure of replicating simian virus 40 minichromosomes. The replication fork, core histone segregation and terminal structures . J Mol Biol.
1986; ; 189 : :189.–204. 3.
DePamphilis
ML
,
Wassarman
PM
. Replication of eukaryotic chromosomes: a close-up of the replication fork . Annu Rev Biochem.
1980; ; 49 : :627.–666. 4.
Taddei
A
,
Roche
D
,
Sibarita
JB
, et al.
Duplication and maintenance of heterochromatin domains . J Cell Biol.
1999; ; 147 : :1153.–1166. 5.
Sobel
RE
,
Cook
RG
,
Perry
CA
, et al.
Conservation of deposition-related acetylation sites in newly synthesized histones H3 and H4 . Proc Natl Acad Sci USA.
1995; ; 92 : :1237.–1241. 6.
Seale
RL
. Assembly of DNA and protein during replication in HeLa cells . Nature.
1975; ; 255 : :247.–249. 7.
Alabert
C
,
Groth
A
. Chromatin replication and epigenome maintenance . Nat Rev Mol Cell Biol.
2012; ; 13 : :153.–167. 8.
Margueron
R
,
Justin
N
,
Ohno
K
, et al.
Role of the polycomb protein EED in the propagation of repressive histone marks . Nature.
2009; ; 461 : :762.–767. 9.
Scharf
AN
,
Barth
TK
,
Imhof
A
. Establishment of histone modifications after chromatin assembly . Nucleic Acids Res.
2009; ; 37 : :5032.–5040. 10.
Sirbu
BM
,
Couch
FB
,
Feigerle
JT
, et al.
Analysis of protein dynamics at active, stalled, and collapsed replication forks . Genes Dev.
2011; ; 25 : :1320.–1327. 11.
Kliszczak
AE
,
Rainey
MD
,
Harhen
B
,
NA
D
, et al.
mediated chromatin pull-down for the study of chromatin replication . Sci Rep.
2011; ; 1 : :95.. 12.
Alabert
C
,
Bukowski-Wills
JC
,
Lee
SB
, et al.
Nascent chromatin capture proteomics determines chromatin dynamics during DNA replication and identifies unknown fork components . Nat Cell Biol.
2014; ; 16 : :281.–293. 13.
Gilbert
DM
. Evaluating genome-scale approaches to eukaryotic DNA replication . Nat Rev Genet.
2010; ; 11 : :673.–684. 14.
Kustatscher
G
,
Hégarat
N
,
Wills
KL
, et al.
Proteomics of a fuzzy organelle: interphase chromatin . EMBO J.
2014; ; 33 : :648.–664. 15.
Unger, S
,
Górna
MW
,
Le Béchec
A
, et al.
FAM111A mutations result in hypoparathyroidism and impaired skeletal development . Am J Hum Genet.
2013; ; 92 : :990.–995. 16.
Bertin
A
,
Mangenot
S
. Structure et dynamique de la particule cœur de nucléosome . Med Sci (Paris).
2008; ; 24 : :715.–719. 17.
Emadali
A
,
Gallagher-Gambarelli
M
. La protéomique quantitative par la méthode SILAC . Med Sci (Paris).
2009; ; 25 : :835.–842. |