| |
| Med Sci (Paris). 2014 February; 30(2): 186–193. Published online 2014 February 24. doi: 10.1051/medsci/20143002017.L’ingénierie des génomes par les TALEN Barbara Dupret1 and Pierre-Olivier Angrand1,2* 1Inserm U908, université de Lille 1, bâtiment SN3, Cité scientifique, 59655Villeneuve d’Ascq, France 2Institut de recherche interdisciplinaire, CNRS USR 3078, parc scientifique de la Haute Borne, 50, avenue Halley, 59658Villeneuve d’Ascq, France |
Depuis le début de la biologie expérimentale, les animaux sont utilisés afin d’étudier divers phénomènes physiologiques, et il est admis que l’expérimentation animale a considérablement accéléré notre compréhension de la fonction des gènes. Pendant de nombreuses années, l’obtention de mutants se basait sur des méthodes de génétique classique, telles que la mutagenèse chimique ou insertionnelle. Cependant, ces approches ne permettent pas une étude ou une « édition » ciblée du génome. De plus, le séquençage complet ou partiel du génome des organismes modèles a ouvert de nouvelles perspectives pour l’étude fonctionnelle des gènes. Parallèlement, la génération, au cours des dernières décennies, de modèles murins génétiquement modifiés a permis de reproduire et d’étudier un grand nombre de maladies humaines [
1]. Toutefois, l’obtention de ces modèles par l’inactivation classique de gènes nécessite l’utilisation de cellules souches embryonnaires (ES, embryonic stem) et reste un processus onéreux, long et réservé aux modèles murins (souris) et rat. Récemment, l’émergence de nouvelles technologies a révolutionné l’ère de la génétique inverse en permettant l’introduction de modifications génétiques précises, de manière efficace et rapide, chez de nombreux génomes modèles [
51]. Les méthodes qui utilisent les TALEN sont actuellement parmi les plus prometteuses. Les TALEN sont des protéines chimériques, constituées d’un domaine de liaison à l’ADN et d’un domaine endonucléasique, qui vont induire une cassure double-brin d’ADN en un site spécifique du génome. La réparation de la cassure peut alors s’accompagner de mutations. De cette façon, les TALEN peuvent être utilisées pour cibler des mutations au niveau de sites choisis du génome. |
Cassures double-brin d’ADN et modifications génomiques Les méthodes de modification des génomes nécessitent des cassures double-brin d’ADN et dépendent des systèmes cellulaires de réparation de ces cassures [
52]. Chez les eucaryotes, il existe deux mécanismes de réparation des cassures double-brin d’ADN : la réparation par jonction d’extrémités non homologues (NHEJ, non-homologous end joining) et la réparation par recombinaison homologue (HDR, homology directed repair) [
2,
3]. La jonction d’extrémités non homologues lie les extrémités cassées et peut s’accompagner de la perte ou du gain de quelques nucléotides, résultant d’évènements de mutagenèse différents au niveau du site de clivage de l’ADN [
4]. En revanche, la réparation par recombinaison homologue utilise une séquence d’ADN homologue comme modèle pour la restauration de l’intégrité de l’ADN génomique. Aussi, la réparation peut être précise et contrôlée, identique à la séquence de l’ADN qui sert de source pour la recombinaison. Toutefois, la réparation des cassures double-brin d’ADN par recombinaison homologue reste, en général, beaucoup moins efficace que la réparation par jonction d’extrémités non homologues. |
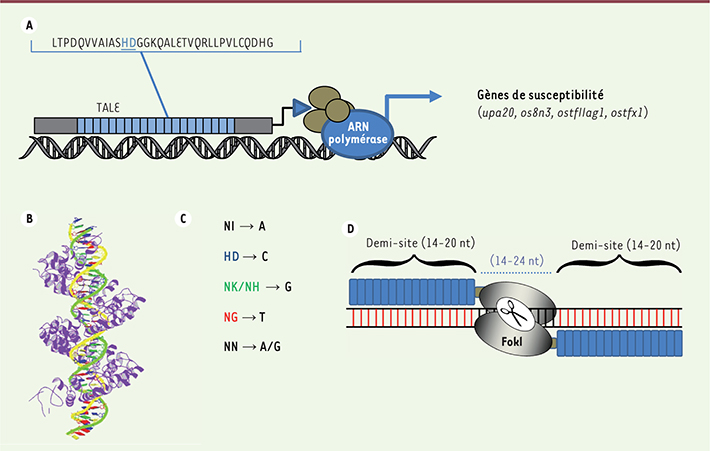
TALEN et ingénierie génomique Les TALEN combinent les propriétés de liaison à l’ADN des TALE (transcription activator-like effector) et de clivage de l’ADN par l’endonucléase FokI (Flavobacterium okeanokoites endonuclease I). Les TALE sont des facteurs de transcription identifiés chez certains phytopathogènes du genre Xanthomonas, qui entrent dans le noyau et activent la transcription de gènes cibles chez l’hôte (Figure 1A) [
5,
6]. La région centrale des TALE est composée d’un nombre variable de répétitions (7 à 30). Chaque répétition est constituée de 34 acides aminés et se lie à un nucléotide cible unique. Les TALE s’associent alors à l’ADN en formant une hélice qui s’enroule autour de l’ADN où chaque répétition reconnaît un nucléotide de la séquence cible (Figure 1B) [
7,
8]. Au sein des répétitions, ce sont les acides aminés 12 et 13 qui déterminent la spécificité de liaison aux nucléotides de chacun de ces domaines répétés [
9–
11] (Figure 1C). Ainsi, parmi les di-résidus variables 12 et 13 les plus utilisés, NI (Asn-Ile), HD (His-Asp), NK (Asn-Lys)/NH (Asn-His) et NG (Asn-Gly) reconnaissent respectivement les nucléotides A, C, G et T. Les di-résidus NN (Asn-Asn), quant à eux, reconnaissent préférentiellement les nucléotides G et, dans une moindre mesure, les nucléotides A. En dépit d’une plus faible spécificité des di-résidus NN pour les nucléotides G, ces derniers sont souvent préférés aux di-résidus NK et NH en raison d’une plus forte affinité pour leur cible [
12,
13]. La région de liaison à l’ADN des TALE se termine par la dernière répétition (LR, last repeat) qui n’est composée que de 20 résidus, mais possède toutefois les di-résidus variables qui assurent la spécificité de liaison à un nucléotide particulier. La région amino-terminale adjacente aux répétitions se lie au nucléotide T et est également importante pour la reconnaissance de l’ADN. La séquence cible des TALE doit donc posséder un T en 5’.
 | Figure 1.
Organisation des TALE et TALEN. A. Les TALE ( transcription activator-like effector) sont des facteurs de transcription produits par les bactéries pathogènes du genre Xanthomonas. Injectés dans les plantes par le système de sécrétion de type III, ils sont importés dans le noyau, se lient aux boîtes UTP et activent les gènes de susceptibilité, tels upa20, os8n3, ostfIIag1 ou ostfx1 de la plante hôte. Les TALE sont constitués de répétition d’un motif de 34 acides aminés. Au sein de ces répétitions, les acides aminés 12 et 13 (en bleu) reconnaissent un nucléotide spécifique. B. Les TALE se lient à leurs cibles en formant une hélice qui s’enroule autour de l’ADN. Chaque répétition du TALE reconnaît un nucléotide adjacent (d’après [ 7]). Source : protein data bank (PDB ID 3UGM) http://www.rcsb.org/pdb/home/home.do. C. Il existe un code de reconnaissance précis entre les acides aminés 12-13 et les nucléotides cibles : N, Asp ; I, Ile ; H, His ; D, Asp ; K, Lys. D. Association d’une paire de TALEN et structure de son site d’ADN cible. Nt : nucléotide. |
L’assemblage modulaire de répétitions choisies permet donc de développer des TALE artificiels capables de reconnaître théoriquement n’importe quelle séquence d’ADN génomique. En outre, la présence éventuelle de séquences génomiques hors cibles (off-targets) pour ces TALE artificiels est facilement prédictible [
14]. Cependant, malgré l’existence d’algorithmes de recherche de ces séquences hors cibles et de la haute spécificité de liaison des TALE à leur séquence, la recherche expérimentale d’éventuels sites hors cible, dont le ciblage peut conduire à des effets non désirés, reste nécessaire [
15–
17]. En fusionnant le domaine de clivage de la nucléase FokI à un TALE artificiel, il devient possible d’induire une cassure de l’ADN en un site spécifique et, en conséquence, d’éditer le génome par réparation de la cassure double-brin [13,
18]. La dimérisation du domaine nucléasique de FokI est nécessaire au clivage de l’ADN. Aussi, il faut une paire de TALEN pour induire une cassure double-brin d’ADN en un point donné du génome (Figure 1D). Le site de reconnaissance par les TALEN consiste en deux séquences de 14-20 paires de bases (pb) reconnues par chaque monomère de la paire de TALEN, séparées par une séquence de 14-24 pb qui facilite la dimérisation de FokI (Figure 1D). La taille de la séquence d’espacement entre les sites de liaison de la paire de TALEN dépend, notamment, de la nature de la région carboxy-terminale adjacente aux répétitions [
16, 18]. C’est au sein de cette séquence d’espacement que la cassure double-brin d’ADN se produit. |
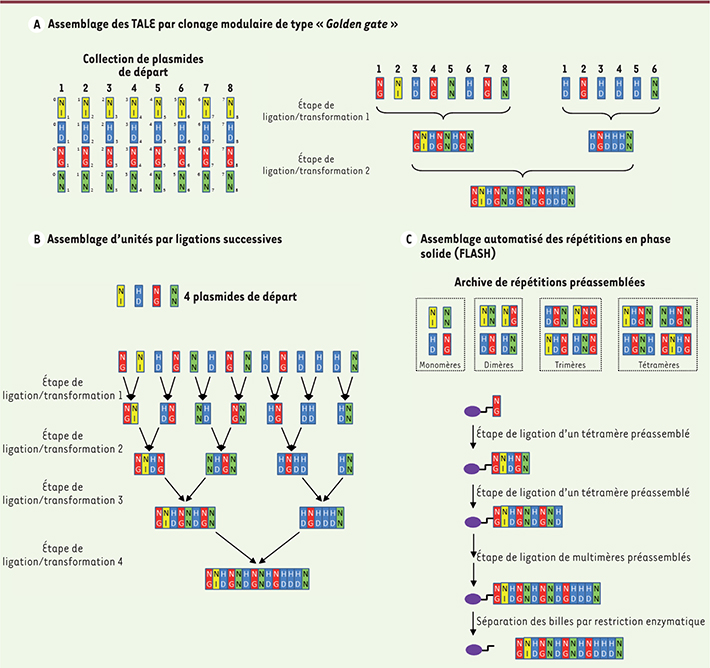
Afin d’atteindre une bonne spécificité dans la reconnaissance de l’ADN cible, les TALEN doivent se lier à des séquences de 14 à 20 pb. L’utilisation des TALEN repose donc sur un assemblage rapide et efficace des répétitions du TALE artificiel. Cependant, la construction des TALEN peut s’avérer difficile en raison du grand nombre de modules répétitifs qui constituent les TALE. Aussi, plusieurs méthodes d’ingénierie ont été mises au point afin d’assembler efficacement les TALEN (Figure 2). Certaines méthodes sont basées sur le clonage modulaire Golden gate qui utilise des enzymes de restriction de type IIS. Cette stratégie permet d’assembler jusqu’à 10 répétitions en une seule réaction de ligation, et la réalisation de TALEN en cinq jours [
19–
22]. Une deuxième stratégie, l’assemblage d’unités par ligations successives (UA, unit assembly ; REAL, restriction enzyme and ligation), utilise des enzymes de restriction compatibles qui produisent des extrémités cohésives complémentaires (par exemple les isocaudamères NheI et SpeI). À partir de quatre plasmides qui contiennent les répétitions se liant spécifiquement aux nucléotides A, C, G et T, une infinité de TALE artificiels peuvent être facilement générés par les techniques de restriction/ligation conventionnelles [
23–
25]. Le clonage sans ligation (LIC, ligation-independent cloning), qui utilise de longues extrémités cohésives hybridées in vitro, puis liées par les enzymes bactériennes après transformation, est également une méthode simple et rapide qui permet l’assemblage des TALEN à grande échelle [
26]. D’autres approches, qui n’utilisent pas les méthodes de clonage traditionnelles, ni la sélection et l’amplification des bactéries après les étapes de ligation, mais qui sont basées sur l’assemblage automatisé des répétitions en phase solide (FLASH, fast ligation-based automatable solid-phase high-throughput ; ICA, iteractive capped assembly), sont également développées. Ces stratégies permettent l’ingénierie des TALEN en quelques heures et peuvent être utilisées à plus grande échelle [
27,
28]. Ces différentes approches rendent la génération des TALEN accessible à des coûts relativement faibles à tous les laboratoires, même les plus petits.
 | Figure 2.
Stratégies d’assemblage des TALEN. A. Les méthodes d’assemblages par clonage modulaire de type « Golden gate » utilisent les enzymes de restriction de type IIS et permettent l’assemblage d’au plus dix répétitions orientées en une seule étape de ligation, grâce à l’utilisation d’extrémités cohésives différentes [ 19]. B. L’assemblage d’unités par ligations successives permet l’ingénierie de TALEN contenant un nombre illimité de répétitions à partir de quatre plasmides de départ par des séries de ligations successives [ 23]. C. Les méthodes de ligation en phase solide permettent l’assemblage des TALEN sur un support sans passer par les étapes de transformation et de sélection des bactéries durant les étapes intermédiaires [ 27]. |
|
Champs d’application des TALEN Depuis leur première utilisation rapportée en 2010 [13], les TALEN ont été largement utilisés aussi bien in vitro dans des cellules en culture, qu’in vivo dans un grand nombre d’organismes modèles, tels que la levure [13,
20], les plantes [19, 22,
29], le xénope [
30,
31], les poissons [19, 23,
32–
35], la drosophile [25], Caenorhabditis elegans [
36], le rat [17], la souris [
37], le lapin [
38], la vache ou le cochon [
39] (Tableau I). Ainsi, les TALEN peuvent donc être employés pour manipuler beaucoup, voir tous les génomes.
Tableau I.
| Organismes |
Description |
Références |
| Levure (Saccharomyces cerevisiae) |
Insertion de mutations de type indel dans les gènes ura3, lys2, ade2 et remplacement du gène ura3 par recombinaison homologue induite par les TALEN |
[20] |
| Poisson zèbre (Danio rerio) |
Première utilisation des TALEN chez un vertébré modèle : insertion de mutations de type indel dans les gènes tnikb, hey2 et gria3a chez le poisson zèbre |
[23,
24] |
| Drosophile (Drosophila melanogaster) |
Première utilisation des TALEN chez la drosophile : insertion de mutations de type indel dans le gène yellow
|
[25] |
| Nématode (Caenorhabditis elegans) |
Première utilisation des TALEN chez C. elegans : insertion de mutations de type indel dans le gène ben-1
|
[36] |
| Souris |
Première utilisation des TALEN chez la souris : génération de souris porteuses de mutations de type indel dans les gènes Pibf1 et Sepw1
|
[37] |
| Rat |
Première utilisation des TALEN chez le rat : génération de rats porteurs de mutations de type indel dans le gène Igm
|
[17] |
| Lapin |
Première utilisation des TALEN chez le lapin : génération de lapins porteurs de mutations de type indel dans les gènes Rag1 et Rag2
|
[38] |
| Xénope (Xenopus tropicalis) |
Génération de xénopes porteurs de mutations de type indel dans le gène tyr
|
[30] |
| Poisson médaka (Oryzias latipes) |
Génération de mutations de type indel dans le gène Dj-1 chez le médaka |
[35] |
| Vache |
Génération de mutations de type indel dans les gènes Acan12 et GDF83.1
|
[39] |
| Cochon |
Génération de mutations de type indel dans les gènes Rela et Ldlr
|
[39] |
| Riz (Oryza sativa) |
Génération de mutations de type indel dans le promoteur du gène Os11n3 responsable de la résistance des plantes aux souches pathogènes de Xanthomonas (X. orysae) |
[29] |
| Poisson zèbre (Danio rerio) |
Insertion d’un site EcoRV dans le gène ponzr1 et d’un site loxP dans le gène crhr2 par réparation des cassures double-brin d’ADN induites par les TALEN, en utilisant une matrice d’ADN simple-brin |
[
33] |
| Poisson zèbre (Danio rerio) |
Introduction de la GFP dans le gène th par réparation par recombinaison homologue des cassures double-brin d’ADN induites par les TALEN |
[
34] |
| Cochon |
Génération de larges délétions et inversions (environ 7-kpb) dans le gène Dmd à l’aide de deux paires de TALEN dans des fibroblastes de cochon |
[39] |
| Ver à soie (Bombyx mori) |
Génération de larges délétions (environ 800-pb) dans le gène Bmblos2 à l’aide de deux paires de TALEN |
[
42] |
| Cellules humaines (Jurkat et RPE-1) |
Génération d’une translocation entre les gènes NPM1 (nucleolar phosphoprotein B23, numatrin) et ALK (anaplastic lymphoma kinase), respectivement situés sur les chromosomes 5 et 2, à l’aide de deux paires de TALEN afin d’induire la translocation t(2;5)(p23;q35) trouvée chez les patients atteints de lymphomes de type ALCL (anaplasique à grandes cellules) |
[
43] |
| Cellules humaines (iPS) |
Restauration du gène α1-antitrypsine fonctionnel par réparation par recombinaison homologue des cassures double-brin d’ADN induites par les TALEN dans des cellules iPS (induced pluripotent stem cells) provenant de patients |
[
44] |
Exemples d’utilisation des TALEN.
|
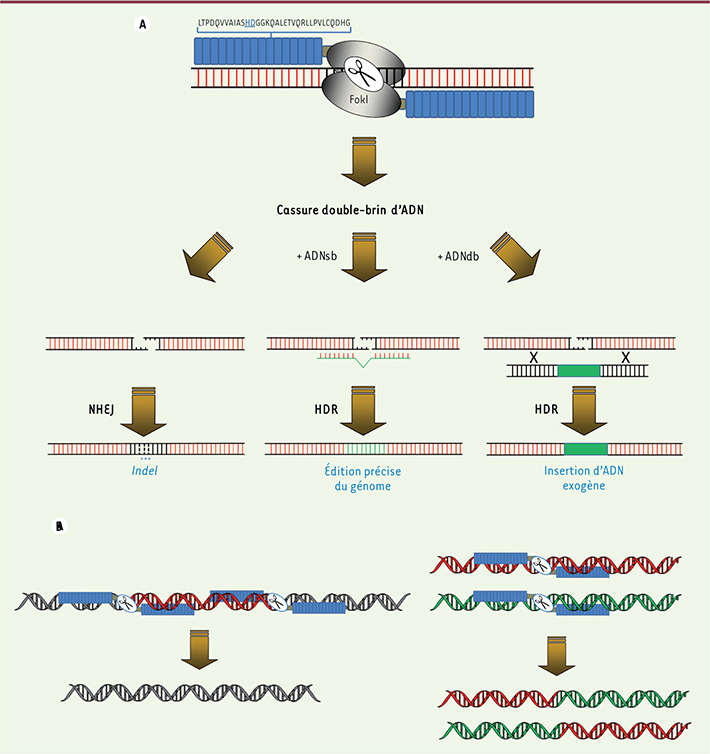
Une paire de TALEN permet de cibler une cassure double-brin d’ADN en un site choisi du génome (Figure 3A). Cette cassure de l’ADN est généralement réparée par jonction d’extrémités non homologues et peut s’accompagner de petites insertions et/ou délétions de paires de bases (mutations de type indel) qui conduisent à des modifications du génome. Les mutations de type indel induites par l’action des TALEN sont de taille variable et conduisent souvent à des décalages de la phase de lecture quand elles sont situées dans les régions codantes du génome. Selon la paire de TALEN considérée, la fréquence de mutation est variable et peut se situer entre 0 % à plus de 70 % [
40]. L’utilisation d’un ADN matrice avec les TALEN permet de modifier le génome de manière particulièrement précise. En présence d’un ADN homologue donneur simple-brin ou double-brin, la cassure double-brin d’ADN peut stimuler la voie de réparation par recombinaison homologue, permettant l’insertion d’une séquence d’ADN exogène [18, 33, 34] ou le remplacement précis d’un nucléotide particulier [
41]. La réparation des cassures double-brin d’ADN par jonction d’extrémités non homologues est beaucoup plus efficace que la réparation par recombinaison homologue. Aussi, l’inactivation de gènes par les TALEN est plus aisée que le remplacement de gènes ou l’insertion d’ADN exogènes. Une paire de TALEN permet donc la génération de mutations de type indel, l’insertion de mutations ponctuelles ou l’insertion des séquences exogènes. Il est également possible d’utiliser deux paires de TALEN qui ciblent le même chromosome afin d’induire des délétions et/ou des inversions de larges segments chromosomiques (plusieurs centaines de kpb) [27, 42]. La délétion de larges régions génomiques par les TALEN est particulièrement intéressante pour l’étude de la fonction des longs ARN non codants. En effet, les mutations ponctuelles ou de type indel ne sont certainement pas suffisantes pour abroger la fonction de ces ARN qui ne possèdent pas de phase de lecture ouverte. Enfin, l’utilisation de deux paires de TALEN qui ciblent des chromosomes différents permet d’induire une translocation réciproque [43] (Figure 3B).
 | Figure 3.
Ingénierie des génomes par les TALEN. A. Les TALEN induisent une cassure double-brin (db) d’ADN en un site spécifique du génome. La réparation de cette cassure par jonction d’extrémités non homologues (NHEJ) conduit à des insertions ou délétions d’un nombre variable de nucléotides (mutations indel). En présence d’ADN matriciel simple-brin (sb) ou double-brin (db), la réparation des cassures par recombinaison homologue (HDR) permet l’introduction de substitutions nucléotidiques précises ou l’insertion de séquences d’ADN exogènes. B. L’utilisation de deux paires de TALEN en cis sur le même chromosome conduit à la délétion des séquences (en rouge) localisées entre les deux cassures (à gauche), alors que l’introduction de cassures double-brin d’ADN sur deux chromosomes permet de générer des translocations réciproques (à droite). |
Ainsi, la technologie d’édition des génomes à l’aide des TALEN, en induisant un grand nombre de modifications génomiques différentes, ouvre de larges champs d’application et offre de nombreuses perspectives thérapeutiques. Les TALEN sont largement employés pour induire l’inactivation de gènes [19, 20, 22–25, 29–31, 34–39], mais aussi pour restaurer la fonction de gènes mutants [41], introduire des marqueurs de l’expression génique, tels que la GFP (green fluorescent protein) [34], ou générer des translocations chromosomiques [43]. La possibilité d’introduire des sites LoxP et/ou FRT (Flp recombinase target) par recombinaison homologue impliquant les TALEN [33] permet également d’augmenter le répertoire des manipulations génomiques possibles en ouvrant la voie aux stratégies d’inactivation de gènes conditionnelles ou spécifiques de tissus. Le fait que les TALEN puissent être utilisés chez de nombreux organismes afin de générer des modifications génomiques va inévitablement conduire à l’expansion considérable du nombre d’animaux modèles pour l’étude des maladies humaines et pour la recherche médicale. Il est également probable que l’utilisation des TALEN remplace progressivement les méthodes classiques de génétique inverse chez la souris. En effet, l’emploi des TALEN permet l’inactivation de gènes, la modification de gènes ou l’introduction de séquences exogènes dans le génome murin sans passer par les étapes du ciblage de gènes dans les cellules ES et de génération des souris chimères avant transmission germinale. De la même manière, l’utilisation des TALEN, en permettant l’insertion de séquences exogènes en des sites particuliers et contrôlés du génome, peut servir d’alternative à la transgenèse classique chez la souris ou chez d’autres organismes modèles tels que le poisson zèbre ou la drosophile. En combinaison avec l’utilisation de technologies cellulaires basées sur les cellules souches pluripotentes induites (iPS), les TALEN se révèlent être des outils prometteurs en médecine régénératrice [15, 44,
45]. Choi et al. [44] ont, par exemple, utilisé la stratégie de réparation par recombinaison homologue des cassures double-brin d’ADN induite par les TALEN pour restaurer un gène α1-antitrypsine fonctionnel dans des cellules iPS dérivées de patients porteurs d’une déficience dans le gène codant pour l’α1-antitrypsine, avec une efficacité de ciblage des deux allèles de 25 % à 33 %. Cette étude montre la faisabilité des stratégies de transplantations autologues dans lesquelles les cellules dérivées de patients pourraient être corrigées ex vivo avant leur réimplantation ; ces approches sont associées, en principe, à une diminution des risques de complications immunitaires. |
Conclusion et perspectives Les TALEN sont des ciseaux moléculaires capables d’induire des cassures double-brin d’ADN en un site choisi, au sein de n’importe quel génome. Cette spécificité est conférée par l’existence d’un code de reconnaissance précis des nucléotides par les répétitions des TALE. La réparation des cassures double-brin d’ADN, par jonction d’extrémités non homologues ou par recombinaison homologue, permet la modification précise des génomes. Grâce au développement de plusieurs stratégies, l’assemblage des TALEN artificiels devient un exercice aisé et rapide. Aussi, au cours des trois dernières années, l’utilisation des TALEN a pris une ampleur considérable. L’application des TALEN pour induire toutes sortes de modifications génétiques dans de nombreux organismes modèles démontre la robustesse et la simplicité de cette approche, et de nombreuses preuves de concept appliquées à l’ingénierie des génomes ont été décrites (Tableau I). Les TALEN sont au cœur d’une véritable révolution dans le domaine de la génétique inverse, et il est probable qu’ils deviennent, au cours des prochaines années, un des outils les plus efficaces et les plus utilisés en recherche fondamentale, tout comme en recherche médicale. Les TALEN s’ajoutent à d’autres outils qui permettent d’induire des cassures double-brin d’ADN ciblées afin de modifier les génomes, tels que les méganucléases [
46], les nucléases à doigts de zinc (ZFN, zinc-finger nuclease) [
47] ou le système CRISPR (clustered regularly interspaced short palindromic repeat)/Cas9 nouvellement développé [
48]. Les méganucléases sont des endonucléases qui se caractérisent par un site de reconnaissance de l’ADN de grande taille (12 à 40 bp), généralement absent ou présent en peu d’exemplaires dans un génome donné. Cependant, la réalisation de recombinaisons génétiques induites par des méganucléases est limitée par le répertoire d’enzymes disponibles, car la probabilité de trouver une méganucléase susceptible de couper un gène donné en un site spécifique reste faible, et ce malgré les efforts d’ingénierie de nouvelles méganucléases pourvues de nouveaux sites de reconnaissance. Les ZFN sont des protéines chimériques, entre un domaine de liaison à l’ADN, constitué de répétitions de doigts de zinc de type C2H2 et le domaine endonucléasique de FokI. Chaque doigt de zinc reconnaît une série de trois nucléotides. Cependant, l’ingénierie des génomes par les ZFN est difficilement applicable aux régions dépourvues ou pauvres en trinucléotides GNN (où N représente n’importe quel nucléotide), car ces séquences sont préférentiellement ciblées par les doigts de zinc de type C2H2. Cette contrainte limite l’utilisation large des ZFN afin de modifier des génomes. À l’inverse du code de reconnaissance de l’ADN par les TALE ou les doigts de zinc de type C2H2, le système CRISPR/Cas9 reconnaît une séquence d’ADN cible grâce à un ARN guide, tandis que la protéine bactérienne Cas9 associée clive l’ADN sur les deux brins. Le clivage par le système CRISPR/Cas9 nécessite la présence d’une séquence nucléotidique particulière appelée PAM (protospacer adjacent motif) adjacente au site de reconnaissance de l’ARN guide, sur le brin complémentaire. Pour le système CRISPR/Cas9 dérivé de Streptococcus pyogenes, la séquence PAM est NGG. La nécessité de sa présence fait qu’environ 40 % du génome peut être la cible du clivage par le complexe ribonucléoprotéique CRISPR/Cas9. Actuellement, encore relativement peu d’applications du système CRISPR/Cas9 sont décrites, mais il semble que cette approche de modification des génomes soit aussi efficace que celle basée sur les TALEN. Plus récemment, le code de reconnaissance des séquences d’ADN par les TALE a été utilisé pour cibler des protéines ou activités enzymatiques autres que nucléasiques, au niveau de régions génomiques particulières. En particulier, Miyanari et al. [
49] ont utilisé les TALE pour cibler la GFP au niveau de séquences génomiques spécifiques afin d’étudier la dynamique des chromosomes. De manière similaire, Maeder et al. ont décrit la déméthylation d’îlots CpG spécifiques grâce à l’utilisation de TALE artificiels fusionnés au domaine catalytique hydroxylase de la protéine TET1 [
50]. Il est fort probable que d’autres applications qui utilisent les TALE artificiels soient rapidement développées. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
L’activité du laboratoire est soutenue par l’Inserm, le CNRS, l’université de Lille 1, la région Nord-Pas de Calais et le Fond européen de développement régional (FEDER) au travers du contrat de projets État-région (CPER) 2007-2013.
|
Footnotes |
1.
International mouse knockout consortium. ,
Collins
FS
,
Rossant
J
,
Wurst
W
. A mouse for all reasons . Cell.
2007; ; 128 : :9.–13. 2.
Lieber
MR
. The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway . Annu Rev Biochem.
2010; ; 79 : :181.–211. 3.
Van den Bosch
M
,
Lohman
PH
,
Pastink
A
. DNA double-strand break repair by homologous recombination . Biol Chem.
2002; ; 383 : :873.–892. 4.
Grabarz
A
,
Barascu
A
,
Guirouilh-Barbat
J
,
Lopez
BS
. Initiation of DNA double strand break repair: signaling and single-stranded resection dictate the choice between homologous recombination, non-homologous end-joining and alternative end-joining . Am J Cancer Res.
2012; ; 2 : :249.–268. 5.
Kay
S
,
Hahn
S
,
Marois
E
, et al.
A bacterial effector acts as a plant transcription factor and induces a cell size regulator . Science.
2007; ; 318 : :648.–651. 6.
Bogdanove
AJ
,
Schornack
S
,
Lahaye
T
. TAL effectors: finding plant genes for disease and defense . Curr Opin Plant Biol.
2010; ; 13 : :394.–401. 7.
Mak
AN
,
Bradley
P
,
Cernadas
RA
, et al.
The crystal structure of TAL effector PthXo1 bound to its DNA target . Science.
2012; ; 335 : :716.–719. 8.
Deng
D
,
Yan
C
,
Pan
X
, et al.
Structural basis for sequence-specific recognition of DNA by TAL effectors . Science.
2012; ; 335 : :720.–723. 9.
Boch
J
,
Scholze
H
,
Schornack
S
, et al.
Breaking the code of DNA binding specificity of TAL-type III effectors . Science.
2009; ; 326 : :1509.–1512. 10.
Moscou
MJ
,
Bogdanove
AJ
, A simple cipher governs DNA recognition by TAL effectors . Science.
2009; ; 326 : :1501.. 11.
Cong
L
,
Zhou
R
,
Kuo
YC
, et al.
Comprehensive interrogation of natural TALE DNA-binding modules, transcriptional repressor domains . Nat Commun.
2012; ; 3 : :968.. 12.
Streubel
J
,
Blücher
C
,
Landgraf
A
,
Boch
J
. TAL effector RVD specificities and efficiencies . Nat Biotechnol.
2012; ; 30 : :593.–595. 13.
Christian
M
,
Cermak
T
,
Doyle
EL
, et al.
Targeting DNA double-strand breaks with TAL effector nucleases . Genetics.
2010; ; 186 : :757.–761. 14.
Grau
J
,
Boch
J
,
Posch
S
. TALEN offer: genome-wide TALEN off-target prediction . Bioinformatics.
2013; ; 29 : :2931.–2932. 15.
Hockemeyer
D
,
Wang
H
,
Kiani
S
, et al.
Genetic engineering of human pluripotent cells using TALE nucleases . Nat Biotechnol.
2011; ; 29 : :731.–734. 16.
Mussolino
C
,
Morbitzer
R
,
Lütge
F
, et al.
A novel TALE nuclease scaffold enables high genome editing activity in combination with low toxicity . Nucleic Acids Res.
2011; ; 39 : :9283.–9293. 17.
Tesson
L
,
Usal
C
,
Ménoret
S
, et al.
Knockout rats generated by embryo microinjection of TALENs . Nat Biotechnol.
2011; ; 29 : :695.–696. 18.
Miller
JC
,
Tan
S
,
Qiao
G
, et al.
A TALE nuclease architecture for efficient genome editing . Nat Biotechnol.
2011; ; 29 : :143.–148. 19.
Cermak
T
,
Doyle
EL
,
Christian
M
, et al.
Efficient design, assembly of custom TALEN, other TAL effector-based constructs for DNA targeting . Nucleic Acids Res.
2011; ; 39 : :e82.. 20.
Li
T
,
Huang
S
,
Zhao
X
, et al.
Modularly assembled designer TAL effector nucleases for targeted gene knockout and gene replacement in eukaryotes . Nucleic Acids Res.
2011; ; 39 : :6315.–6325. 21.
Li
L
,
Piatek
MJ
,
Atef
A
, et al.
Rapid and highly efficient construction of TALE-based transcriptional regulators and nucleases for genome modification . Plant Mol Biol.
2012; ; 78 : :407.–416. 22.
Morbitzer
R
,
Elsaesser
J
,
Hausner
J
,
Lahaye
T
. Assembly of custom TALE-type DNA binding domains by modular cloning . Nucleic Acids Res.
2011; ; 39 : :5790.–5799. 23.
Huang
P
,
Xiao
A
,
Zhou
M
, et al.
Heritable gene targeting in zebrafish using customized TALENs . Nat Biotechnol.
2011; ; 29 : :699.–700. 24.
Sander
JD
,
Cade
L
,
Khayter
C
, et al.
Targeted gene disruption in somatic zebrafish cells using engineered TALENs . Nat Biotechnol.
2011; ; 29 : :697.–698. 25.
Liu
J
,
Li
C
,
Yu
Z
, et al.
Efficient and specific modifications of the Drosophila genome by means of an easy TALEN strategy . J Genet Genomics.
2012; ; 39 : :209.–215. 26.
Schmid-Burgk
JL
,
Schmidt
T
,
Kaiser
V
, et al.
A ligation-independent cloning technique for high-throughput assembly of transcription activator-like effector genes . Nat Biotechnol.
2013; ; 31 : :76.–81. 27.
Reyon
D
,
Tsai
SQ
,
Khayter
C
, et al.
FLASH assembly of TALENs for high-throughput genome editing . Nat Biotechnol.
2012; ; 30 : :460.–465. 28.
Briggs
AW
,
Rios
X
,
Chari
R
, et al.
Iterative capped assembly: rapid, scalable synthesis of repeat-module DNA such as TAL effectors from individual monomers . Nucleic Acids Res.
2012; ; 40 : :e117.. 29.
Li
T
,
Liu
B
,
Spalding
MH
, et al.
High-efficiency TALEN-based gene editing produces disease-resistant rice . Nat Biotechnol.
2012; ; 30 : :390.–392. 30.
Ishibashi
S
,
Cliffe
R
,
Amaya
E
. Highly efficient bi-allelic mutation rates using TALENs in Xenopus tropicalis
. Biol Open.
2012; ; 1 : :1273.–1276. 31.
Lei
Y
,
Guo
X
,
Liu
Y
, et al.
Efficient targeted gene disruption in Xenopus embryos using engineered transcription activator-like effector nucleases (TALENs) . Proc Natl Acad Sci USA.
2012; ; 109 : :17484.–17489. 32.
Huang
P
,
Zhu
Z
,
Lin
S
,
Zhang
B
. Reverse genetic approaches in zebrafish . J Genet Genomics.
2012; ; 39 : :421.–433. 33.
Bedell
VM
,
Wang
Y
,
Campbell
JM
, et al.
In vivo genome editing using a high-efficiency TALEN system . Nature.
2012; ; 491 : :114.–118. 34.
Zu
Y
,
Tong
X
,
Wang
Z
, et al.
TALEN-mediated precise genome modification by homologous recombination in zebrafish . Nat Methods.
2013; ; 10 : :329.–331. 35.
Ansai
S
,
Sakuma
T
,
Yamamoto
T
, et al.
Efficient targeted mutagenesis in medaka using custom-designed transcription activator-like effector nucleases . Genetics.
2013; ; 193 : :739.–749. 36.
Wood
AJ
,
Lo
TW
,
Zeitler
B
, et al.
Targeted genome editing across species using ZFNs, TALENs . Science.
2011; ; 333 : :307.. 37.
Sung
YH
,
Baek
IJ
,
Kim
DH
, et al.
Knockout mice created by TALEN-mediated gene targeting . Nat Biotechnol.
2013; ; 31 : :23.–24. 38.
Song
J
,
Zhong
J
,
Guo
X
, et al.
Generation of RAG 1- and 2-deficient rabbits by embryo microinjection of TALENs . Cell Res.
2013; ; 23 : :1059.–1062. 39.
Carlson
DF
,
Tan
W
,
Lillico
SG
, et al.
Efficient TALEN-mediated gene knockout in livestock . Proc Natl Acad Sci USA.
2012; ; 109 : :17382.–17387. 40.
Cade
L
,
Reyon
D
,
Hwang
WY
, et al.
Highly efficient generation of heritable zebrafish gene mutations using homo- and heterodimeric TALENs . Nucleic Acids Res.
2012; ; 40 : :8001.–8010. 41.
Sun
N
,
Liang
J
,
Abil
Z
,
Zhao
H
. Optimized TAL effector nucleases (TALENs) for use in treatment of sickle cell disease . Mol Biosyst.
2012; ; 8 : :1255.–1263. 42.
Ma
S
,
Zhang
S
,
Wang
F
, et al.
Highly efficient, specific genome editing in silkworm using custom TALENs . PLoS One.
2012; ; 7 : :e45035.. 43.
Piganeau
M
,
Ghezraoui
H
,
De Cian
A
, et al.
Cancer translocations in human cells induced by zinc finger and TALE nucleases . Genome Res.
2013; ; 23 : :1182.–1193. 44.
Choi
SM
,
Kim
Y
,
Shim
JS
, et al.
Efficient drug screening and gene correction for treating liver disease using patient-specific stem cells . Hepatology.
2013; ; 57 : :2458.–2468. 45.
Ding
Q
,
Lee
YK
,
Schaefer
EA
, et al.
A TALEN genome-editing system for generating human stem cell-based disease models . Cell Stem Cell.
2013; ; 12 : :238.–251. 46.
Arnould
S
,
Delenda
C
,
Grizot
S
, et al.
The I-CreI meganuclease and its engineered derivatives: applications from cell modification to gene therapy . Protein Eng Des Sel.
2011; ; 24 : :27.–31. 47.
Urnov
FD
,
Rebar
EJ
,
Holmes
MC
, et al.
Genome editing with engineered zinc finger nucleases . Nat Rev Genet.
2010; ; 11 : :636.–646. 48.
Hwang
WY
,
Fu
Y
,
Reyon
D
, et al.
Efficient genome editing in zebrafish using a CRISPR-Cas system . Nat Biotechnol.
2013; ; 31 : :227.–229. 49.
Miyanari
Y
,
Ziegler-Birling
C
,
Torres-Padilla
ME
. Live visualization of chromatin dynamics with fluorescent TALEs . Nat Struct Mol Biol.
2013; ; 20 : :1321.–1324. 50.
Maeder
ML
,
Angstman
JF
,
Richardson
ME
, et al.
Targeted DNA demethylation and activation of endogenous genes using programmable TALE-TET1 fusion proteins . Nat Biotechnol.
2013; ; 31 : :1137.–1142. 51.
Cohen-Tannoudji
M
,
Guénet
JL
. Une nouvelle ère pour la génétique du rat . Med Sci (Paris).
2011; ; 27 : :387.–390. 52.
Gatinois
V
,
Puechberty
J
,
Lefort
G
, et al.
Les remaniements chromosomiques complexes : un paradigme pour l’étude de l’instabilité chromosomique . Med Sci (Paris).
2014; ; 30 : :55.–63. |