| |
| Med Sci (Paris). 2013 April; 29(4): 358–361. Published online 2013 April 26. doi: 10.1051/medsci/2013294008.Évolution et cancer Expansion des familles de gènes dangereux par duplication du génome Séverine Affeldt,1** Param Priya Singh,1** Ilaria Cascone,2 Rasim Selimoglu,2 Jacques Camonis,2 and Hervé Isambert1* 1CNRS-UPMC UMR168, France.
2Inserm U830, Institut Curie, Centre de recherche, 26, rue d’Ulm, 75248Paris, France. MeSH keywords: Animaux, Évolution biologique, ADN tumoral, Évolution moléculaire, Humains, Mutation, Tumeurs, génétique, Sélection génétique |
Si la conservation des gènes essentiels à la vie des organismes se conçoit intuitivement bien, à l’inverse, l’étonnante expansion des familles de gènes à l’origine des cancers ou d’autres maladies génétiques chez les vertébrés pose question. Alors qu’on pourrait supposer que la multiplication de ces gènes « dangereux » confère malgré tout un avantage sélectif, nos travaux récents [
1] suggèrent en fait que ces gènes ont été multipliés et conservés en raison de leur dangerosité à la suite de deux accidents génétiques majeurs correspondant à des duplications globales de génome. |
De l’expansion des familles de gènes dangereux chez les vertébrés Pour comprendre l’origine de l’expansion des familles de gènes dangereux chez les vertébrés, il faut remonter à l’ancêtre commun de tous les vertébrés, il y a quelque 500 millions d’années. Par un mécanisme presque toujours létal, mais qui a joué un rôle essentiel au cours de l’évolution, notre lignée encore invertébrée a entièrement dupliqué son génome deux fois de suite et a survécu à ces deux accidents génétiques majeurs. Ces deux duplications globales du génome ont conduit à l’émergence et à la complexification des vertébrés dont certains gènes ont conservés jusqu’à quatre copies. Au total, un quart à un tiers de nos gènes serait directement issu de ces deux duplications de génome à l’origine des vertébrés [
2]. Ces gènes sont appelés gènes ohnologues en l’honneur du généticien Susumu Ohno qui fut le premier à avancer l’hypothèse de ces duplications globales du génome chez les vertébrés [
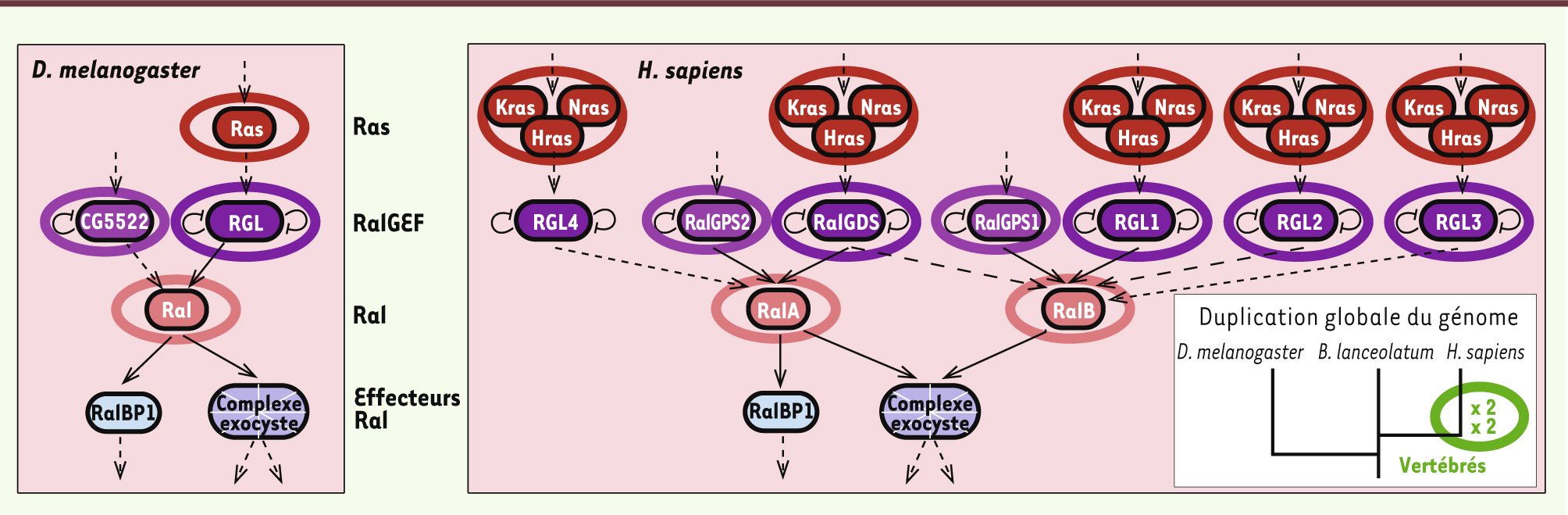
3]. Après une duplication globale du génome, les organismes perdent généralement près de 80 à 90 % des gènes dupliqués, ce qui entraîne une expansion hétérogène de leurs voies de signalisation (Figure 1) et de leurs réseaux de gènes [
4–
6]. Cependant, de façon surprenante, on constate que les copies ohnologues retenues dans le génome humain comportent un nombre élevé de gènes dangereux, c’est-à-dire présentant une forte susceptibilité aux mutations délétères dominantes comme les oncogènes notamment. Certains de ces gènes dangereux ont même gardé leurs quatre copies depuis l’origine des vertébrés ! C’est le cas par exemple des gènes RalGEF (Ral guanine-nucleotide exchange factor) (Figure 1) qui activent les voies Ras-Ral impliquées dans la migration et la prolifération cellulaires dans des tumeurs [
7]. De même, le gène Ras, qu’on retrouve en un seul exemplaire chez les invertébrés comme la drosophile (Figure 1), a conservé chez la plupart des vertébrés trois ohnologues proto-oncogéniques (KRas, HRas et NRas) (
Figure 1
) qui présentent des mutations constitutivement actives dans plus de 25 % des cas de cancer chez l’homme.
 | Figure 1Expansion des voies de signalisation Ras-Ral. Voies de signalisation Ras-Ral chez la drosophile ( A) et chez l’homme ( B). Après les deux duplications globales de génomes chez l’ancêtre des vertébrés, chez l’homme, le gène RGL a conservé ses quatre copies ohnologues ( RalGDS, RGL1, RGL2, RGL3), le gène Ras a conservé trois copies ohnologues ( Kras, Hras, Nras) et le gène Ral a conservé deux copies ohnologues ( RalA, RalB) [ 7]. Cet exemple illustre la nécessaire réorganisation des voies de signalisation et des réseaux d’interactions protéine-protéine après un évènement de duplication globale du génome, et l’élimination par divergence d’une partie des gènes dupliqués [ 4– 6]. |
Au delà de ces exemples, nous avons effectué une étude d’exploration de données à grande échelle sur l’ensemble des gènes humains, à partir des bases de données accessibles en ligne (comme COSMIC [
8], CancerGenes [
9]) et de publications. Ceci nous a permis de mettre en évidence une forte association entre la dangerosité des ohnologues et leur rétention dans le génome humain. Dans ces analyses, nous avons considéré différentes classes de gènes susceptibles d’être affectés par des mutations délétères. Ces classes comprennent notamment des gènes dont l’implication dans les cancers est connue et des gènes dont les mutations induisent typiquement des phénotypes délétères dominants, par exemple via une perte d’interaction d’auto-inhibition entraînant un gain de fonction permanent pour le gène mutant. Les résultats obtenus [1] indiquent que les 8 095 gènes impliqués dans des cancers ou dans des maladies génétiques comportent significativement plus d’ohnologues que l’ensemble des 20 506 gènes codant pour les protéines humaines, soit 48 % contre 35 % (48 % ; 3 844/8 095 ; p = 1,3 x 10−129, test χ2). En outre, ce biais de rétention augmente fortement dans le cas des oncogènes (61 %; 493/813; p = 1,4 x 10−54, test χ2) ou des gènes dont la protéine est auto-inhibée (76 %; 350/461 ; p = 2,7 x 10−77, test χ2). De même, lorsque l’on considère des classes de gènes combinant plusieurs facteurs de dangerosité, telles que la classe des oncogènes dont la protéine est auto-inhibée, ces biais de rétention dépassent les 90 % (91 %; 104/114; p = 6,9 x 10−37, test χ2). À l’inverse, nous avons montré que les gènes associés à des mutations délétères récessives chez l’homme, ainsi que la plupart des gènes essentiels connus chez la souris, n’ont pas conservé un excès d’ohnologues. Il apparaît donc que la rétention des gènes ohnologues serait directement liée à leur susceptibilité aux mutations délétères dominantes et non pas à leur nature fondamentale pour l’organisme. Nous avons en fait démontré [1], au moyen d’analyses statistiques d’inférences bayésiennes, telles que l’analyse de Médiation [
10], que la dangerosité est bien la cause principale de la rétention des gènes ohnologues chez l’homme. En particulier, les équilibres entre les niveaux d’expression génétique, fréquemment proposés comme la principale cause des biais de rétention des ohnologues, semblent en fait résulter indirectement des effets des mutations délétères dominantes [1]. |
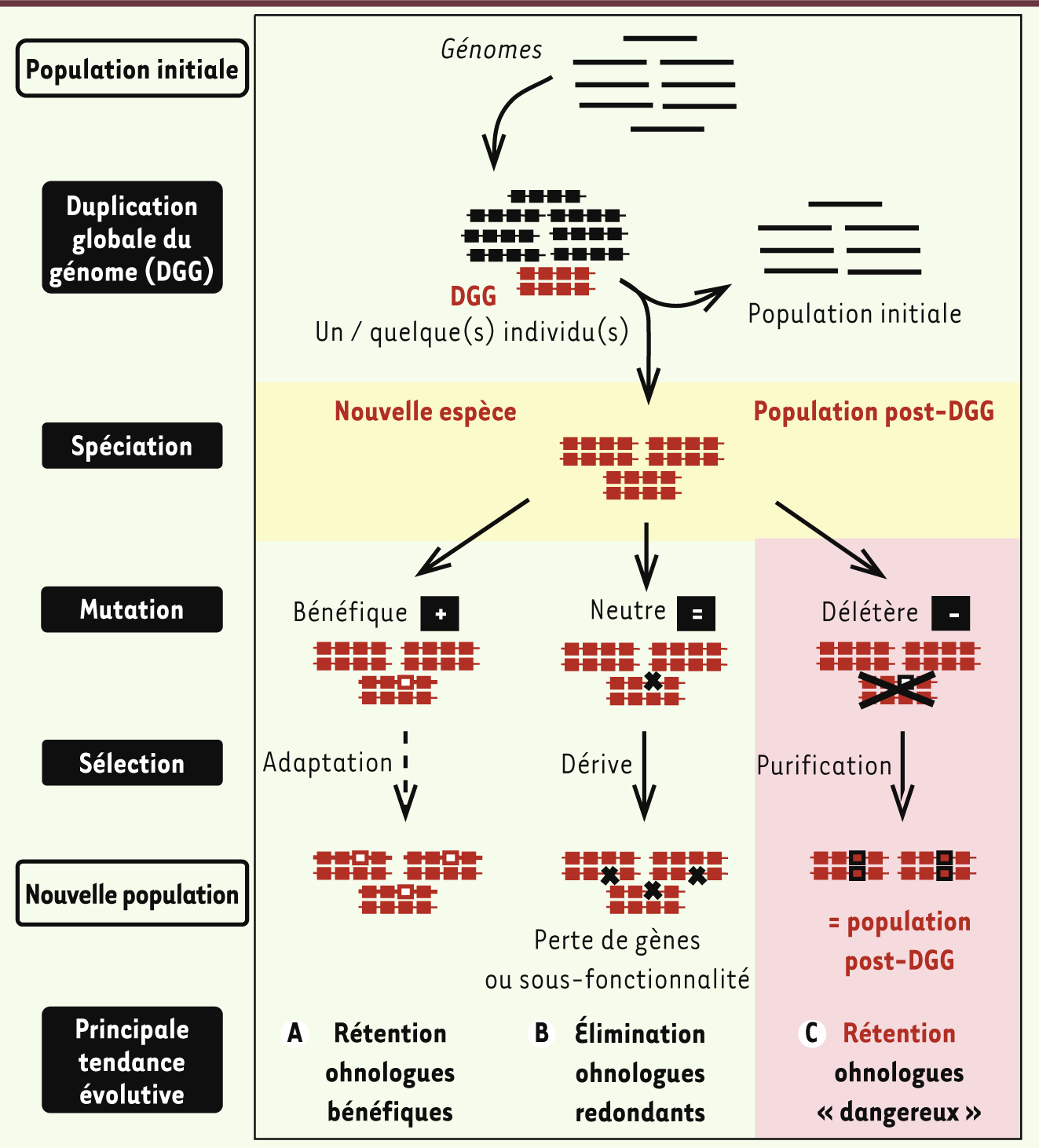
Le mécanisme de conservation des ohnologues Pourquoi les multiples copies ohnologues de ces gènes dangereux, à l’origine de nombreux cancers et maladies génétiques sévères, n’ont-elles pas été éliminées chez les vertébrés ? Pour le comprendre, il faut avoir en tête deux points essentiels (Figure 2) : (1) la duplication globale du génome, lorsqu’elle n’est pas létale, implique nécessairement l’apparition d’une nouvelle espèce composée d’individus possèdant tous initialement leurs gènes en double ; et (2) l’inégalité des gènes face aux mutations. Alors que la plupart des gènes tendent à perdre leur fonction par mutation, les gènes dangereux se caractérisent par le fait que leurs mutations entraînent fréquemment une suractivation, c’est-à-dire un gain de fonction, plutôt qu’une perte de fonction. En général, la perte de fonction d’un ohnologue ne pose pas de problème tant qu’il reste une copie fonctionnelle de ce gène, ce qui conduit à l’élimination progressive d’une des copies de la plupart des ohnologues non dangereux. En revanche, la survenue de mutations conduisant à des gains de fonction ou, plus généralement, à des phénotypes dominants délétères qui caractérisent les ohnologues dangereux, va entraîner des pathologies du développement ou des tumeurs. Celles-ci pénalisent les organismes atteints et, plus ou moins directement, leur descendance qui finira par s’interrompre. Pour autant, les gènes dangereux impliqués ne sont pas éliminés mais au contraire conservés dans la population, puisqu’ils sont encore présents sous une forme non délétère dans le reste de la population issue de la duplication du génome (Figure 2). Ce processus évolutif par élimination de mutants (sélection de purification) se distingue du concept d’avantage sélectif généralement associé à l’évolution (sélection naturelle ou adaptative).
 | Figure 2Évolution des gènes dupliqués après duplicaton globale du génome. A-C. Les lignes horizontales représentent le génome de différents individus. Les carrés symbolisent les gènes dupliqués (rouges) ou non dupliqués (noirs). Les croix noires représentent la perte d’un seul gène (petites croix) ou l’élimination d’un individu (grande croix), tandis que les carrés avec bordures indiquent les copies mutées. Les scénarios d’évolution sont décrits comme il se doit au niveau d’une population d’individus. Lorsqu’elle n’est pas létale, une duplication globale de génome chez un ou plusieurs individus de la population initiale (deux lignes horizontales rouges pour un individu avec duplication complète) implique nécessairement l’émergence d’une nouvelle espèce dont tous les gènes ont été dupliqués. La sélection de purification favorise alors indirectement la conservation des ohnologues dangereux non mutés dans le génome des individus qui survivent. Adapté de [ 1]. |
La multiplication des gènes dangereux chez les vertébrés est donc liée à ce phénomène spécifique et très rare de duplication globale du génome qui existe en fait dans la plupart des branches eucaryotes. Sa compréhension doit se faire au niveau de la génétique des populations au sein des nouvelles espèces issues d’une duplication de génome. Tout se passe comme si les individus de ces populations n’avaient pas pu se débarrasser de nombreux gènes dangereux redondants, directement fixés par spéciation dans leur génome dupliqué et non pas fixés progressivement grâce à un avantage sélectif comme pour la plupart des gènes dupliqués individuellement. Ensuite, les gènes ohnologues conservés se différencient et deviennent souvent des acteurs majeurs du développement, de la signalisation et de la régulation cellulaires. Par exemple, les cadhérines, sorte de colle qui lient les cellules entre elles, ont conservé de multiples ohnologues exprimés dans différents tissus, comme la E-cadhérine qui lie les cellules épithéliales entre elles, ou la N-cadhérine exprimée dans le tube neural et les neurones. Mais les mutations des cadhérines qui entraînent la décohésion des cellules entre elles sont aussi impliquées dans la migration des cellules tumorales et leur dissémination vers d’autres organes. |
Ces accidents génétiques majeurs de doublement du génome survenus il y a 500 millions d’années dans l’évolution des vertébrés ont donc permis l’émergence d’organismes plus complexes, mais aussi la multiplication des gènes dangereux chez les vertébrés. Ces résultats éclairent d’un point de vue évolutif nouveau l’expansion des familles de gènes fréquemment impliquées dans des maladies génétiques et de nombreux cancers. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Singh
PP
,
Affeldt
S
,
Cascone
I
, etal.
On the expansion of dangerous gene repertoires by whole-genome duplications in early vertebrates . Cell Rep.
2012; ; 2 : :1387.–1398. 2.
Makino
T
,
McLysaght
A
. Ohnologs in the human genome are dosage balanced and frequently associated with disease . Proc Natl Acad Sci USA.
2010; ; 107 : :9270.–9274. 3.
Ohno
S
. Evolution by gene duplication . New York: : Springer-Verlag;
1970. 4.
Evlampiev
K
,
Isambert
H
. Modeling protein network evolution under genome duplication and domain shuffling . BMC Syst Biol.
2007; ; 1 : :49.. 5.
Evlampiev
K
,
Isambert
H
. Conservation and topology of protein interaction networks under duplication-divergence evolution . Proc Natl Acad Sci USA.
2008; ; 105 : :9863.–9868. 6.
Stein
RR
,
Isambert
H
. Logistic map analysis of biomolecular network evolution . Phys Rev E.
2011; ; 84 : :051904.. 7.
Cascone
I
,
Selimoglu
R
,
Ozdemir
C
, etal.
Distinct roles of RalA and RalB in the progression of cytokinesis are supported by distinct RalGEFs . EMBO J.
2008; ; 27 : :2375.–2387. 8.
Forbes
SA
,
Bindal
N
,
Bamford
S
, etal.
COSMIC: mining complete cancer genomes in the catalogue of somatic mutations in cancer . Nucleic Acids Res.
2011; ; 39 : :D945.–D950. 9.
Higgins
ME
,
Claremont
M
,
Major
JE
, etal.
CancerGenes: a gene selection resource for cancer genome projects . Nucleic Acids Res.
2007; ; 35 : :D721.–D726. 10.
Pearl
J
. Causality: models, reasoning and inference. , New York: : Cambridge University Press; , 2009. |