| |
| Med Sci (Paris). 2013 February; 29(2): 133–135. Published online 2013 February 28. doi: 10.1051/medsci/2013292006.Un trio de choc pour pallier la chimiorésistance des cancers : c-MYC, PARP1, BIN1 Slovénie Pyndiah1,2* and Daitoku Sakamuro1,3 1Molecular signaling program, Stanley S. Scott cancer center, Louisiana state university, Health sciences center, New Orleans, Louisiana70112, États-Unis 2Institut de génétique moléculaire, cibles thérapeutiques, Inserm U940, 27, rue Juliette Dodu, 75010Paris, France 3Department of biochemistry and molecular biology, GHSU cancer center, Georgia health sciences university, Augusta, Georgia30912, États-Unis MeSH keywords: Protéines adaptatrices de la transduction du signal, génétique, physiologie, Altération de l'ADN, Résistance aux médicaments antinéoplasiques, Régulation de l'expression des gènes tumoraux, Gènes suppresseurs de tumeur, Humains, Modèles biologiques, Thérapie moléculaire ciblée, méthodes, Tumeurs, thérapie, Protéines nucléaires, Poly (ADP-Ribose) polymerase-1, Poly(ADP-ribose) polymerases, Protéines proto-oncogènes c-myc, Protéines suppresseurs de tumeurs |
Vaincre la résistance des cellules cancéreuses à la chimiothérapie est devenu un vrai défi en oncologie clinique. Cette résistance peut survenir dès les premières cures de chimiothérapie, sans phase de sensibilité initiale, témoignant d’une résistance intrinsèque des cellules. En revanche, la résistance acquise se traduit par une progression tumorale survenant secondairement, après une phase de grande chimiosensibilité. Les mécanismes moléculaires impliqués dans ces phénomènes de résistance ont été très étudiés au cours de ces dernières décennies. La dérégulation de gènes codant pour des protéines membranaires, comme le gène multi drug resistance (MDR) qui code pour des pompes d’efflux rejetant le médicament à l’extérieur de la cellule, réduit l’efficacité thérapeutique des molécules anticancéreuses [
1]. Au niveau du foie, le MDR joue un rôle physiologique dans la détoxication cellulaire. Dans les cellules cancéreuses ayant acquis une résistance, le MDR est alors activé. Cependant, d’autres mécanismes de résistance existent probablement. Ainsi, certaines tumeurs possèdent des altérations spécifiques du système de réparation des dommages à l’ADN les rendant vulnérables ou résistantes aux médicaments qui ont pour cible l’ADN. Par exemple, le système de réparation de l’ADN des cellules cancéreuses qui sont porteuses d’une mutation des gènes breast cancer 1 ou 2 (BRCA1 ou 2) est défectueux, ce qui rend ces cellules extrêmement sensibles aux agents intercalants de l’ADN et aux inhibiteurs de la poly-ADP-ribose polymérase (PARP) [
2,
3]. |
BIN1, un suppresseur de tumeur Protéine participant à de multiples fonctions cellulaires, BIN1 (bridging integrator 1) intervient, entre autres, dans la réorganisation du cytosquelette, la polarité, le trafic cellulaire et dans la régulation de la transcription. Au moins dix isoformes ont été décrites, qui diffèrent par leur répartition tissulaire et leur localisation subcellulaire qui sont spécifiques. L’isoforme 9 de BIN1 (qui contient l’exon 13), majoritairement nucléaire, est présente dans pratiquement tous les types cellulaires [
4]. Cette isoforme interagit avec c-MYC et peut inhiber le pouvoir tumorigène des cellules malignes surexprimant c-MYC [
5]. Le taux de BIN1 (toutes isofomes confondues) est diminué dans la majorité des tumeurs et des lignées cancéreuses humaines, et la réexpression de BIN1 (isoforme 9) dans ces cellules entraîne alors un déclin de la croissance cellulaire, voire la mort cellulaire programmée [5,
6]. Ces observations suggèrent que BIN1 pourrait jouer le rôle d’un suppresseur de tumeur qui serait dérégulé dans divers cancers chez l’homme (Figure 1).
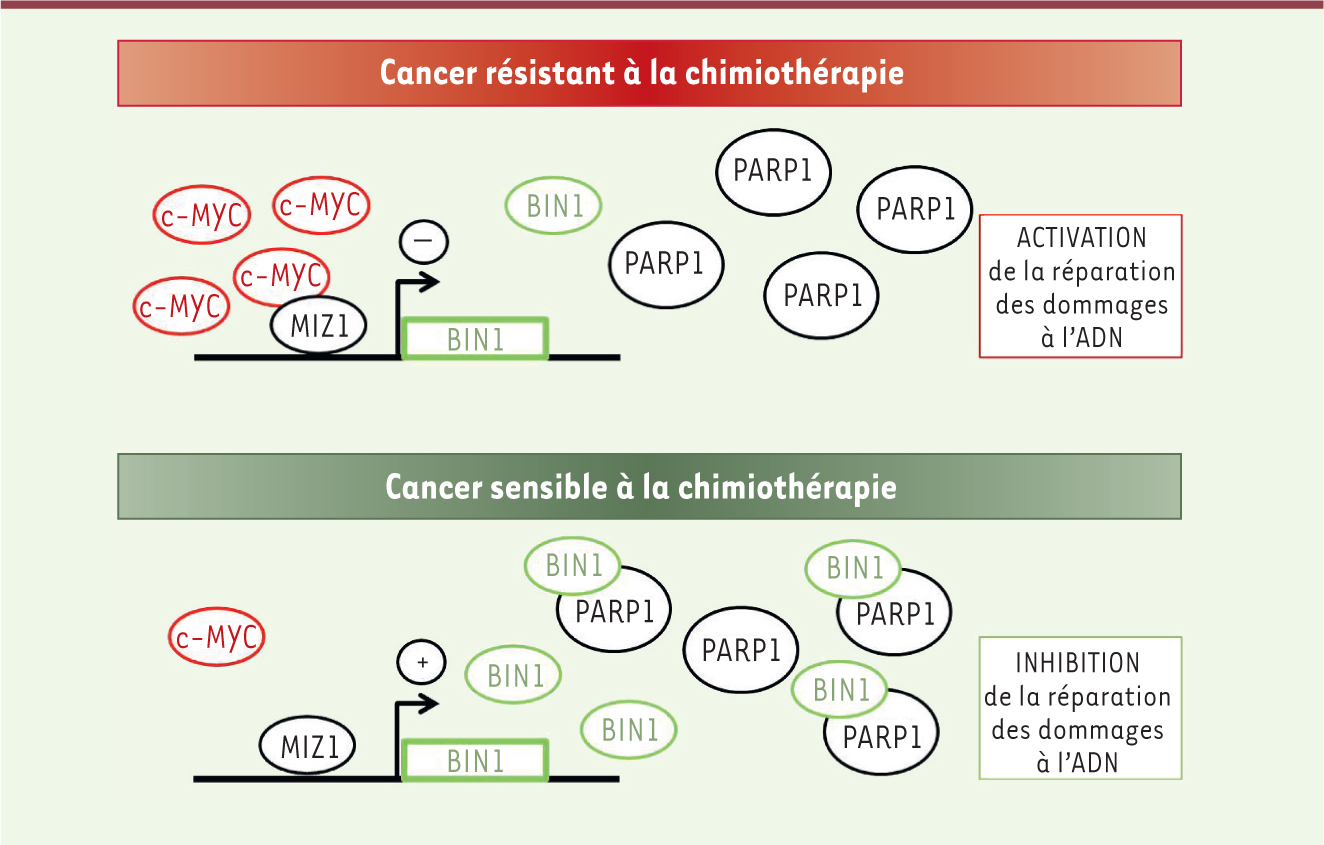
 | Figure 1.
Modèle de régulation de l’expression de BIN1 et de l’activité de PARP1 sous la dépendance de c-MYC. Dans les cancers où c-MYC est abondant, l’interaction de c-MYC avec MIZ1 bloque l’activation transcriptionnelle de BIN1 par le biais de MIZ1. La diminution conséquente de BIN1 libère l’activité de PARP1 déclenchant alors la résistance des cellules cancéreuses à la chimiothérapie. Dans le cas des cancers sensibles à la chimiothérapie, la faible abondance de c-MYC libère l’activité transcriptionnelle de MIZ1 causant une augmentation de BIN1. L’interaction de BIN1 et PARP1 inhibe la capacité de PARP1 à réparer les lésions de l’ADN. |
Dans un travail récent, nous montrons que BIN1 peut aussi réguler la machinerie de réparation de l’ADN en inhibant PARP1 (Poly [ADP-ribose] polymerase 1), une enzyme impliquée dans la réparation de l’ADN [
7]. L’abondance cellulaire de BIN1 est corrélée positivement à la sensibilité aux composés chimiques à base de platine, comme le cisplatine, anticancéreux bien connus provoquant des dommages à l’ADN. En revanche, l’absence de BIN1 est associée à une importante résistance aux traitements à base de cisplatine. L’apport exogène de BIN1 dans des lignées cancéreuses promeut la sensibilité au traitement, indépendamment du statut de p53 [7]. Par ailleurs, dans des essais in vitro, les cellules développant spontanément des résistances au cisplatine ont un taux faible de BIN1, et un apport exogène de cette molécule restaure une sensibilité au traitement [7]. Par conséquent, une déficience de BIN1 semble être associée à une progression de la résistance aux composés à base de platine. |
BIN1 inhibe PARP1, une enzyme clé dans la réparation des dommages à l’ADN Une analyse protéomique a permis d’identifier une interaction directe entre BIN1 et PARP1. PARP1 est abondamment associée à la chromatine, ce qui lui permet de jouer son rôle de « gendarme » et de garantir la stabilité du génome. Lorsque PARP1 détecte des lésions de l’ADN, elle s’active et catalyse la synthèse de polymères d’ADP-ribose (PAR) à partir de la nicotinamide adénine dinucléase (NAD+), utilisée comme précurseur. Les unités de PAR sont transférées à différentes protéines nucléaires cibles et PARP1 constitue l’une des principales cibles. Cette production de PAR modifie localement la structure de la chromatine pour faciliter le recrutement des protéines impliquées dans la réparation de l’ADN cellulaire et fixer les dommages à l’ADN [
8]. Des inhibiteurs chimiques de PARP augmentent l’efficacité des thérapies utilisant des molécules créant des dommages à l’ADN. Ce type de traitement est radical dans le cas où BRCA1 ou BRCA2 est muté1, [3]. Nous avons démontré que le domaine BAR (bin amphiphysine rvs) de BIN1 interagit et inhibe le domaine central de PARP1, supprimant ainsi l’activité catalytique de l’enzyme [7] (Figure 2).
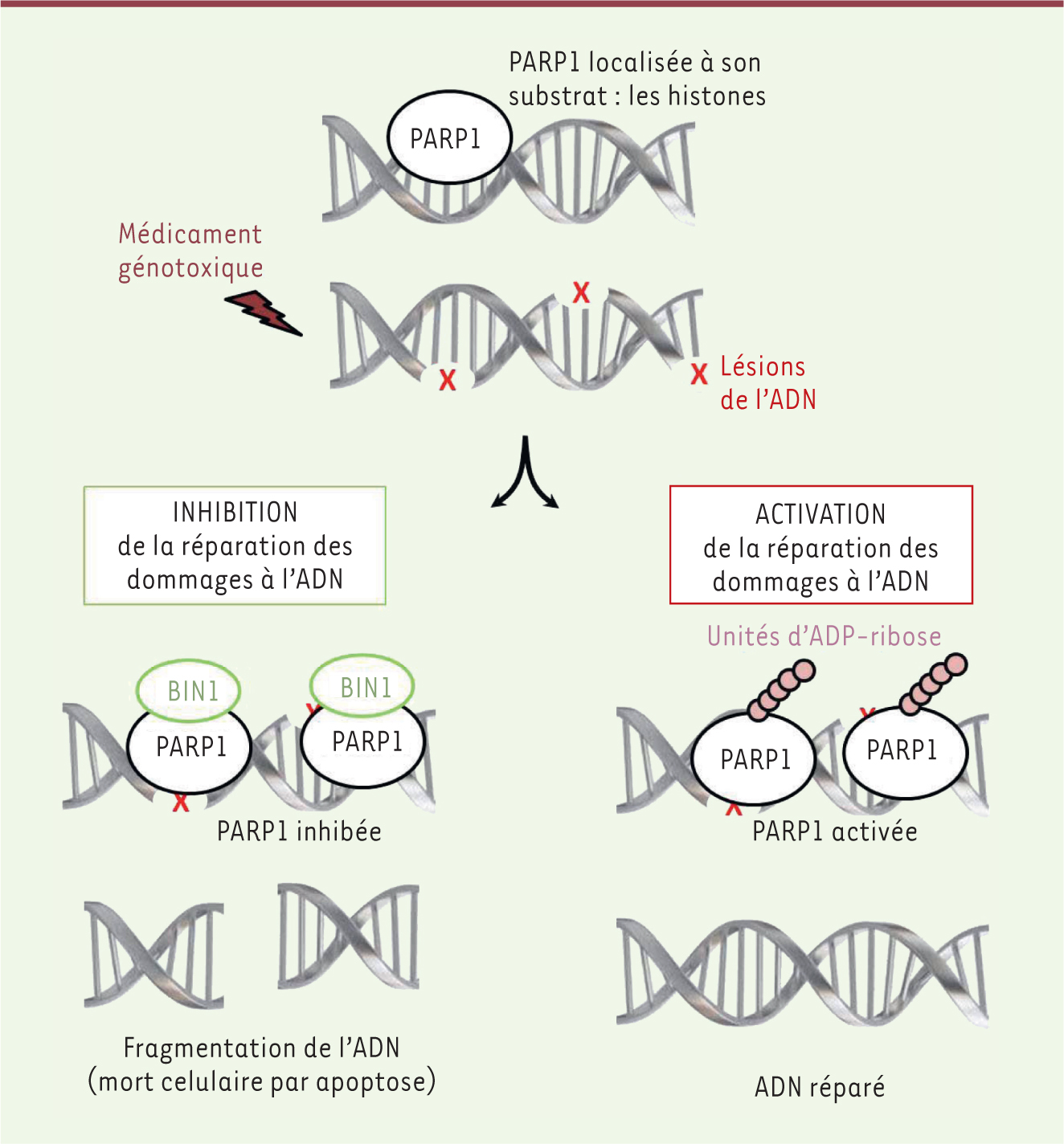
 | Figure 2.
Représentation schématique de la fonction de PARP1 après une cure par un médicament génotoxique. Suite à des cassures de l’ADN, PARP1 est activée et catalyse la synthèse d’unités d’ADP-ribose au niveau de son domaine d’automodification. Cette modification contribue à la réparation de l’ADN endommagé. L’interaction du domaine BAR de BIN1 avec le domaine d’automodification de PARP1 inhibe l’activation de PARP1. En raison de cette inactivation de PARP1, les dommages à l’ADN persistent, provoquant la mort cellulaire par apoptose. |
Outre son rôle dans la réparation de l’ADN, PARP1 est aussi impliquée dans la régulation transcriptionnelle en servant de cofacteur à de multiples facteurs de transcription [
9]. Ainsi, l’inhibition de l’activité de PARP1 par une interaction directe avec BIN1 pourrait non seulement réprimer le système de réparation de l’ADN, mais également être impliquée dans l’inhibition de la transactivation des gènes cibles de c-MYC. |
c-MYC, MIZ1, BIN1 et PARP1 : une boucle de régulation Une augmentation du taux de c-MYC peut inhiber l’expression de BIN1. Cette réaction antagoniste entre c-MYC et BIN1 implique indirectement le facteur de transcription MIZ1 (MYC-interacting zinc finger 1). MIZ1 se fixe au promoteur de BIN1 et active sa transcription. Mais un taux élevé de c-MYC réprime l’effet activateur de MIZ1 sur l’expression de BIN1. Dans ce cas, c-MYC interagit directement avec MIZ1, bloquant alors l’activation de la transcription de BIN1, et induisant une diminution de la concentration de la protéine. Cette diminution de BIN1 libère l’activité de PARP1 facilitant alors la transactivation des gènes cibles de c-MYC [7]. Cette boucle de régulation met l’accent sur la corrélation entre une forte abondance de c-MYC et le développement d’une chimiorésistance aux traitements qui agissent via la création de dommages à l’ADN [
10]. Inversement, une forte quantité de BIN1 inhibe l’activité de PARP1, causant alors une augmentation de la sensibilité au traitement et une inhibition de l’activité de c-MYC. Les conséquences des interactions de BIN1 avec ses partenaires cellulaires dans la transformation cellulaire, les voies de signalisation de réparation de l’ADN, le métabolisme et l’apoptose sont encore mystérieuses. Par exemple, BIN1 interagit avec Ku70 et Ku80, des protéines qui se fixent aux extrémités libres de l’ADN, pour promouvoir la réparation de l’ADN et la stabilité des télomères [
11]. Par ailleurs, en réponse à des dommages à l’ADN, la transcription de BIN1 est activée par le facteur de transcription E2F1, et ce mécanisme joue un rôle dans le déclenchement de l’apoptose dépendant d’E2F1 [
12]. |
Le modèle de régulation que nous avons proposé pourrait avoir des implications cliniques. La régulation de PARP1 par BIN1 implique que dans les cancers où prévaut une dérégulation de c-MYC (citons l’exemple du cancer du sein sans altération de BRCA1 ou BRCA2), BIN1 sera présente en faible abondance et PARP1 hyperactive. L’interaction entre ces trois acteurs déclenche le mécanisme de résistance des cancers aux drogues qui provoquent des lésions de l’ADN. Cela suggère que l’inhibition de PARP pourrait être un moyen de sensibiliser les cancers présentant une dérégulation de c-MYC. En effet, un essai clinique a dévoilé que l’effet combiné de l’inhibition de PARP à la chimiothérapie classique apporte un bénéfice considérable au traitement des cancers du sein « triple négatifs » (absence des récepteurs des oestrogènes [ER], des progestérones [PR] et de l’epidermal growth factor EGF [HER2]) où c-MYC est systématiquement dérégulé [
13]. L’évaluation quantitative ou qualitative de BIN1 pourrait être utilisée comme marqueur biologique prédictif identifiant les cancers qui devraient réellement bénéficier de l’inhibition thérapeutique de PARP1. Il serait fort intéressant de déterminer qui, de l’activation de c-MYC ou de la perte de BIN1, serait le meilleur marqueur pour identifier les cancers qui devraient bénéficier de l’ajout de l’inhibiteur chimique de PARP à la chimiothérapie classique. Si le modèle que nous proposons est validé sur des tumeurs primaires humaines, de nouvelles approches thérapeutiques visant la chimiorésistance des cancers contrôlés par c-MYC pourraient voir le jour. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Goda
K
,
Bacsó
Z
,
Szabó
Z.
Multidrug resistance through the spectacle of P-glycoprotein . Curr Cancer Drug Targets.
2009; ; 9 : :281.–297. 2.
Bryant
HE
,
Schultz
N
,
Thomas
HD
, et al.
Specific killing of BRCA2-deficient tumours with inhibitors of poly(ADP-ribose) polymerase . Nature.
2005; ; 434 : :913.–917. 3.
Helleday
T
,
Bryant
HE
,
Schultz
N.
Poly(ADP-ribose) polymerase (PARP-1) in homologous recombination and as a target for cancer therapy . Cell Cycle.
2005; ; 4 : :1176.–1178. 4.
Wechsler-Reya
R
,
Sakamuro
D
,
Zhang
J
, et al.
Structural analysis of the human BIN1 gene. Evidence for tissue-specific transcriptional regulation and alternate RNA splicing . J Biol Chem.
1997; ; 272 : :31453.–31458. 5.
Sakamuro
D
,
Elliott
KJ
,
Wechsler-Reya
R
,
Prendergast
GC.
BIN1 is a novel MYC-interacting protein with features of a tumour suppressor . Nat Genet.
1996; ; 14 : :69.–77. 6.
Ghaneie
A
,
Zemba-Palko
V
,
Itoh
H
, et al.
Prendergast, Bin1 attenuation in breast cancer is correlated to nodal metastasis and reduced survival . Cancer Biol Ther.
2007; ; 6 : :192.–194. 7.
Pyndiah
S
,
Tanida
S
,
Ahmed
KM
, et al.
C-MYC supresses BIN1 to release poly(ADP-ribose) polymerase 1: a mechanism by which cancer cells acquire cisplatin resistance . Sci Signal.
2011; ; 4 : :ra19.. 8.
Bryant
HE
,
Schultz
N
,
Thomas
HD
, et al.
Specific killing of BRCA2-deficient tumours with inhibitors of poly(ADP-ribose) polymerase . Nature.
2005; ; 434 : :913.–917. 9.
Ji
Y
,
Tulin
AV.
The roles of PARP1 in gene control and cell differentiation . Curr Opin Genet Dev.
2010; ; 20 : :512.–518. 10.
Sklar
MD
,
Prochownik
EV.
Modulation of cis-platinum resistance in Friend erythroleukemia cells by c-myc . Cancer Res.
1991; ; 51 : :2118.–2123. 11.
Ramalingam
A
,
Farmer
GE
,
Stamato
TD
,
Prendergast
GC.
Bin1 interacts with and restrains the DNA end-binding protein complex Ku . Cell Cycle.
2007; ; 6 : :1914.–1918. 12.
Cassimere
EK
,
Pyndiah
S
,
Sakamuro
D.
The c-MYC-interacting proapoptotic tumor suppressor BIN1 is a transcriptional target for E2F1 in response to DNA damage . Cell Death Differ.
2009; ; 16 : :1641.–1653. 13.
O’Shaughnessy
J
,
Osborne
C
,
Pippen
JE
, et al.
Iniparib plus chemotherapy in metastatic triple-negative breast cancer . N Engl J Med.
2011; ; 364 : :205.–214. |