| |
| Med Sci (Paris). 2012 December; 28(12): 1121–1127. Published online 2012 December 21. doi: 10.1051/medsci/20122812024.Penser la biologie dans un cadre phylogénétique
L’exemple de l’évolution des vertébrés Didier Casane1,2 and Patrick Laurenti1,2* 1Laboratoire évolution, génomes et spéciation, UPR 9034 CNRS, avenue de la Terrasse, Bâtiment 13, 91190Gif-sur-Yvette, France 2Université Paris Diderot, Sorbonne Paris Cité, France |
Les arbres phylogénétiques permettent de décrire de façon très synthétique et facile à lire les deux principaux processus de l’évolution : (1) l’anagenèse ou évolution dans une lignée qui est représentée par une branche, et (2) la cladogenèse ou spéciation, qui est représentée par un nœud d’où sont issues deux espèces à partir d’un ancêtre commun. Un arbre phylogénétique qui présente uniquement des dichotomies successives représente très bien les cas de spéciation par division d’une espèce ancestrale en unités indépendantes. Comme tout modèle, c’est une représentation très simplifiée de l’évolution qui ne permet pas de représenter toute la complexité des généalogies des individus et des gènes qu’ils transmettent au cours des générations [
1]. Un arbre phylogénétique ne peut représenter ni les cas de formation d’espèces par hybridation entre espèces, ni les cas de réhomogénéisation des génomes à la suite de transferts de gènes entre espèces proches, ni ceux de transfert de gènes entre espèces éloignées. Il est cependant frappant de constater que c’est en construisant des arbres à partir de différents échantillons de gènes et d’espèces, et en analysant l’incongruence des résultats obtenus que l’on a pu identifier tous ces processus qui s’écartent du modèle de départ [
2]. Les outils de reconstruction des phylogénies moléculaires sont aujourd’hui très sophistiqués et donnent des résultats de plus en plus fiables, principalement grâce à l’application d’approches probabilistes, au développement de modèles plus réalistes pour décrire l’évolution des séquences et à l’utilisation de grandes quantités de séquences [
3]. Nous ne souhaitons pas développer ici une discussion sur l’intérêt et les limites de la construction d’arbres phylogénétiques. Nous concentrerons cette revue sur les problèmes rencontrés à la lecture des arbres par la majorité des lecteurs non spécialistes. En effet, la reconstruction des relations de parenté est devenue depuis quelques dizaines d’années un puissant outil d’analyse en biologie qui est utilisé bien au-delà de son domaine initial, la systématique évolutive [
4,
5]. Désormais, l’utilisation de phylogénies permet, non seulement de mieux comprendre l’évolution des gènes [4,
6], mais aussi de mettre en évidence des transferts horizontaux de gènes entre espèces [
7], d’identifier des événements de recombinaison entre virus [
8], de retracer l’histoire d’une épidémie [
9], de dater des évènements de spéciation [
10], d’étudier la structuration géographique de la diversité génétique des populations [2,
11], de reconstruire l’histoire démographique des populations [
12], de mettre en évidence des évènements d’hybridation entre espèces [
13]. Notre expérience d’enseignant-chercheur nous a appris qu’au-delà des difficultés réelles qui existent pour bien comprendre les méthodes employées, peu familières à la plupart des biologistes, le problème récurrent le plus grave est la mauvaise compréhension de la méthode de lecture des arbres. Dans les copies de nos étudiants, nous retrouvons régulièrement des erreurs bien identifiées [
14–
16] qui sont dues à la persistance du concept obsolète d’échelle des êtres (scala naturae) [
17]. Malheureusement, ce biais d’interprétation des arbres phylogénétiques est également présent dans de nombreuses publications scientifiques récentes. Aussi, nous souhaitons ici reformuler des concepts de base et expliquer leurs représentations afin d’éviter, en particulier, le biais induit par une lecture linéaire des arbres phylogénétiques, et illustrer chez les vertébrés comment ce biais entraîne la persistance de scénarios évolutifs erronés. |
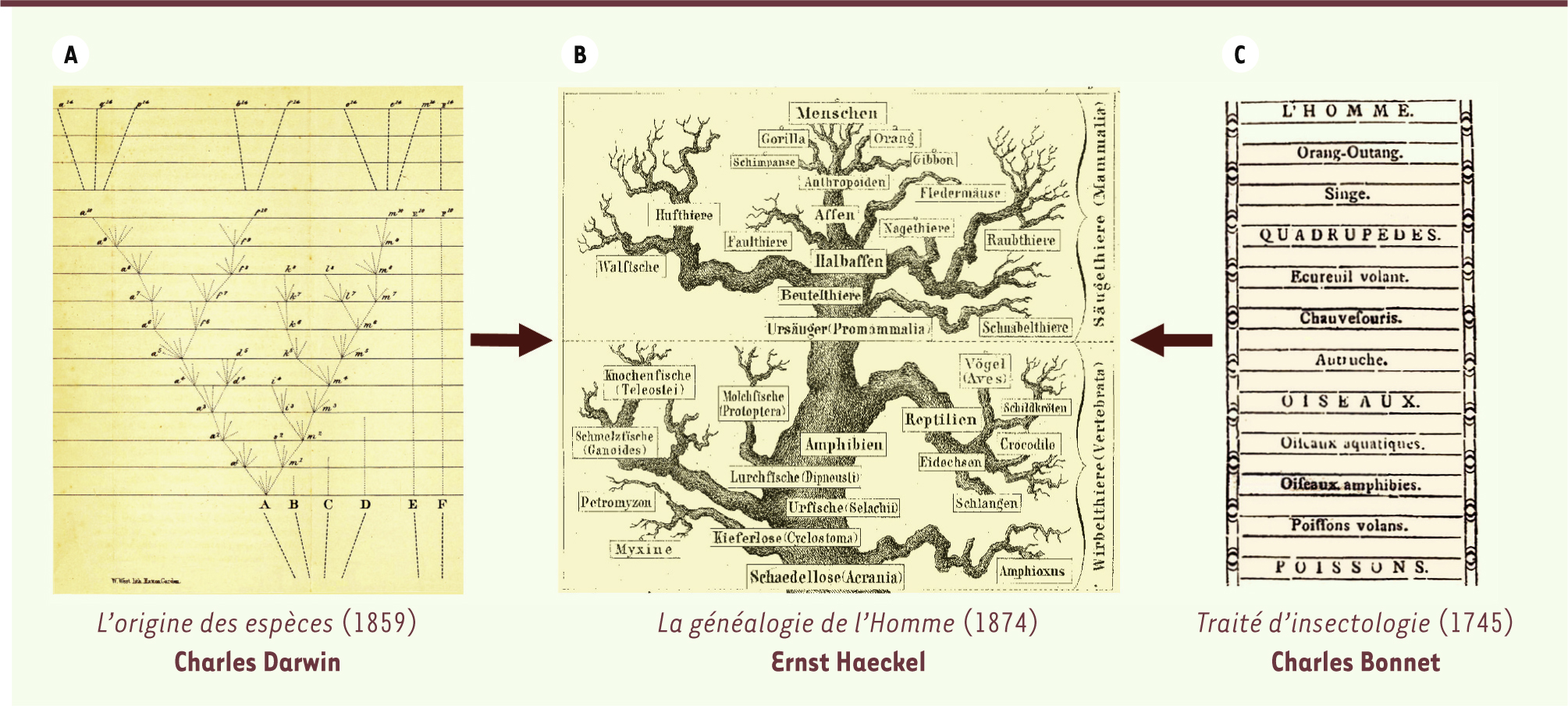
Une histoire qui a très bien commencé, mais qui a rapidement mal tourné La phylogénie et sa représentation la plus courante, l’arbre phylogénétique, apparaissent pour la première fois en 1859 dans « L’origine des espèces » de Charles Darwin [
18]. C’est la seule illustration (partiellement reproduite
Figure 1A
) de cet ouvrage fondateur. Dans cette phylogénie théorique, le temps est symbolisé par les lignes horizontales numérotées de 0 à 14, de la plus ancienne à la plus récente. Les relations phylogénétiques sont matérialisées par des lignes en pointillés qui lient les espèces actuelles (sur la ligne 14) à des ancêtres communs qui sont eux-mêmes liés à des ancêtres communs, et ainsi de suite jusqu’à un ancêtre commun unique. À la suite de Darwin, Ernst Haeckel a reconstruit des phylogénies du vivant, mais sa conception de l’évolution était très différente de celle de Darwin. Par exemple, en 1874, il publie une généalogie de l’homme (dont la partie haute est reproduite sur la
Figure 1B
) qui illustre bien cette conception de l’évolution. Cet arbre représente deux processus qu’il veut illustrer : d’une part, le processus de diversification par spéciation à l’origine des différentes relations de parenté entre taxons1 représentées par les différentes branches de l’arbre, qui est une question secondaire pour Haeckel ; d’autre part, les étapes de l’évolution de l’homme qui constitue, pour lui, le processus principal à illustrer et qui est représenté par le tronc de l’arbre. Du fait de ce choix, quelques noms de taxons sont pris dans le tronc de l’arbre, alors que d’autres sont aux bouts des branches de cet arbre. La dimension temporelle de l’arbre phylogénétique de Darwin est perdue pour mieux illustrer le processus de complexification croissante au cours de l’évolution qui aurait mené à l’homme, sommet de l’évolution (
Figure 1B
). Cette conception de l’évolution rappelle des idées pré-évolutionnistes sur l’organisation de la nature : une classification linéaire des objets de la nature en fonction de leur supposée perfection croissante. Cette conception, qualifiée de « scaliste », car héritée du concept d’échelle des êtres, place invariablement l’homme au sommet en tant que chef d’œuvre de la création (Figure 1C). L’arbre généalogique de Haeckel est une représentation d’idées a priori sur l’évolution et n’est pas le résultat d’une analyse des données. Cette conception n’était pas celle de Darwin qui écrit clairement qu’il ne voit aucune direction privilégiée dans l’évolution. Pour lui, comme pour la quasi-totalité des évolutionnistes actuels, les différentes lignées évolutives ont un statut équivalent. L’homme représente l’état actuel de l’évolution dans une lignée qui n’a pas de position particulière dans l’arbre de la vie. Penser l’évolution dans un cadre phylogénétique permet de reconstruire l’évolution des différentes espèces, sans introduire d’idées préconçues sur une ou des directions privilégiées de l’évolution.
 | Figure 1.
De l’évolution buissonnante darwinienne à l’évolution dirigée vers l’homme proposée par Haeckel.
|
|
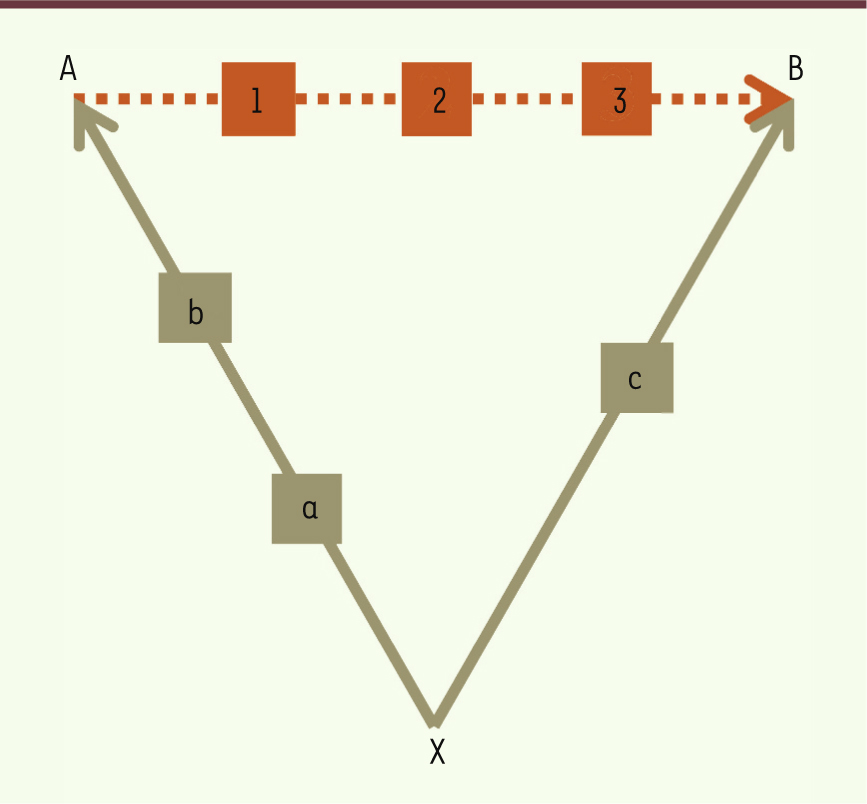
Du bon usage de l’approche comparative La mise en garde de Darwin La persistance d’une lecture linéaire des arbres est d’autant plus étonnante que ce biais fut identifié par Darwin lui-même dès la première édition de L’Origine des espèces (chapitre IX ou X suivant l’édition ; des identificateurs entre parenthèses ont été ajoutés pour faire correspondre cette citation à l’illustration présentée dans la
Figure 2
) : «Il faut d’abord se faire une idée exacte de la nature des formes intermédiaires qui, d’après ma théorie, doivent avoir existé antérieurement. Lorsqu’on examine deux espèces quelconques (A, B) , il est difficile de ne pas se laisser entraîner à se figurer des formes exactement intermédiaires entre elles (1, 2, 3) . C’est là une supposition erronée ; il nous faut toujours chercher des formes intermédiaires (a, b, c) entre chaque espèce et un ancêtre (X) commun, mais inconnu, qui aura généralement différé sous quelques rapports de ses descendants modifiés. »
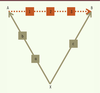 | Figure 2.
Un ancêtre n’est jamais directement observable.
|
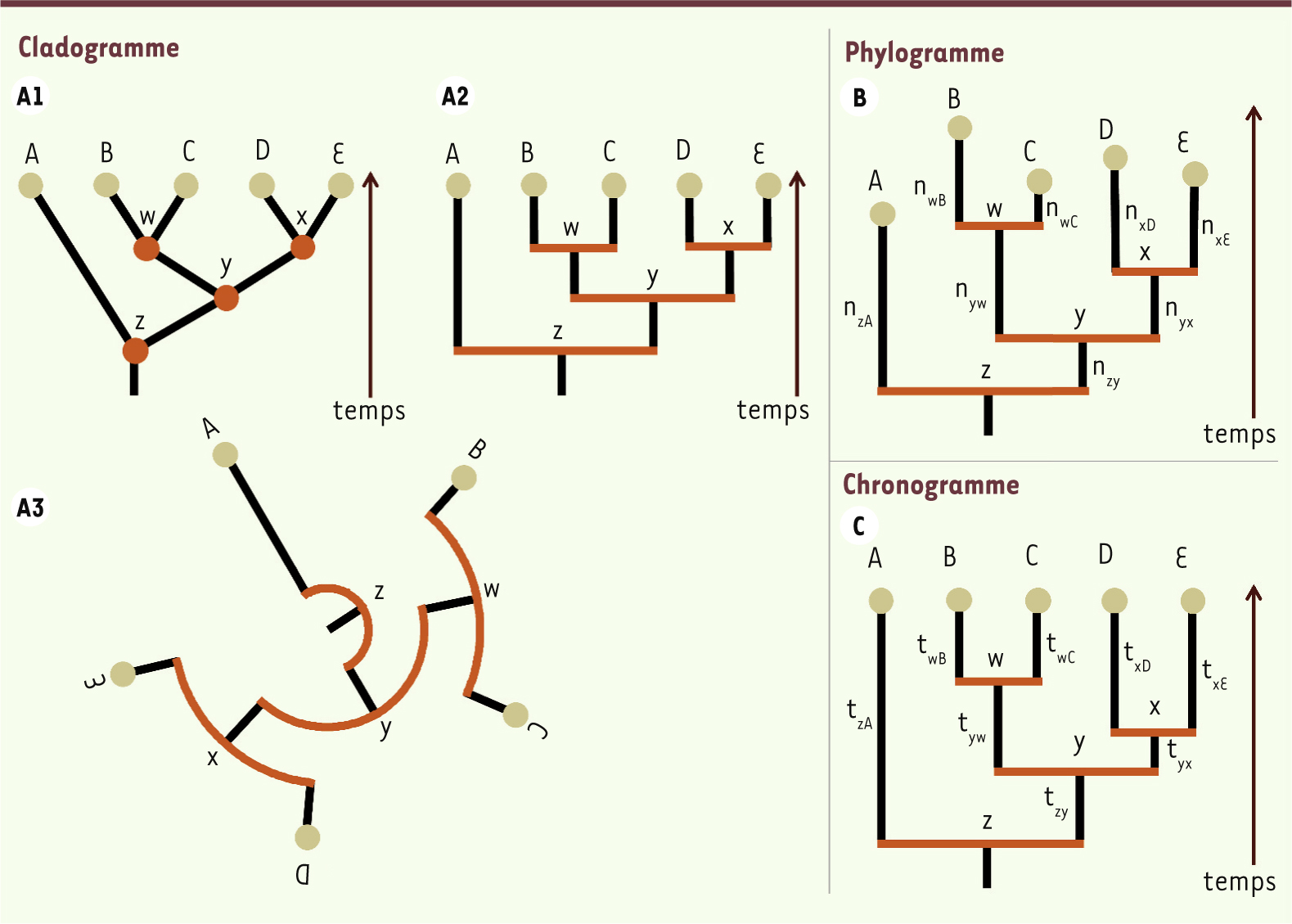
Ce texte attire notre attention sur deux points essentiels : (1) aucune espèce actuelle n’est l’ancêtre d’une autre espèce ; et (2) l’ancêtre commun n’est pas identique à l’une des deux espèces comparées. Ainsi, par exemple, l’homme ne descend pas du chimpanzé ; et l’ancêtre de l’homme et du chimpanzé n’est pas un chimpanzé. L’ancêtre est inconnu, on ne peut en faire qu’une reconstruction plus ou moins précise sur la base de la comparaison des espèces actuelles et fossiles : tout ce qui est commun à l’homme et au chimpanzé était probablement déjà présent chez leur ancêtre. La comparaison de plus de deux espèces implique de tenir compte de toutes les relations de parenté représentées par un arbre phylogénétique. Nous verrons avec l’exemple des vertébrés que si l’on néglige cette recommandation, on peut obtenir une histoire évolutive erronée. Les principales façons de représenter des phylogénies Un arbre phylogénétique est un graphe composé de branches reliant des nœuds par un chemin unique. À l’extrémité des branches terminales, les nœuds externes (ou feuilles) figurent les taxons étudiés (points beiges dans la
Figure 3
). Les nœuds internes correspondent à des ancêtres communs inférés (points ou traits de couleur orange dans la
Figure 3
), à partir desquels divergent des lignées différentes. La longueur des branches peut n’avoir aucune signification (l’arbre est un cladogramme,
Figure 3A.1, 3A.2, 3A.3
) ou contenir une information comme le nombre de changements entre deux nœuds (l’arbre est un phylogramme,
Figure 3B
) ou le temps écoulé entre deux nœuds (l’arbre est un chronogramme,
Figure 3C
). Dans une phylogénie, les nœuds internes sont parfois représentés par des traits horizontaux à la place de points, mais il est important de comprendre que dans tous les cas, la longueur de ces traits n’a aucune signification (voir traits oranges,
Figure 3
). Les phylogénies sont enracinées (un nœud représente l’ancêtre commun de tous les taxons étudiés) et si la racine est en bas, le temps figuré s’écoule du bas vers le haut (voir les flèches du temps,
Figure 3A.1, A.2, 3B, 3C
). Une rotation d’un quart de tour vers la droite, produit un arbre où le temps s’écoule de la gauche vers la droite (Figure 4). Il existe une autre représentation des phylogénies qui est circulaire, le temps s’écoule alors du centre vers l’extérieur (Figure 3A.3). Cette représentation permet de mettre un nombre plus important de taxons dans une phylogénie, mais elle est très difficile à lire quand il y a un grand nombre de taxons. Toutes les phylogénies de la
Figure 3
présentent une même topologie, c’est-à-dire qu’elles indiquent les mêmes relations de parenté entre les taxons. Par exemple, E est plus proche parent de D (ancêtre commun x) que de B (ancêtre commun y). Le taxon le moins apparenté à E est A (ancêtre commun z). Dans un phylogramme, les deux branches qui se rejoignent à un nœud n’ont pas la même longueur si les changements s’accumulent à une plus grande vitesse dans une lignée par rapport à l’autre (par exemple n wB > n wC dans le phylogramme de la
Figure 3B
). Dans un chronogramme, si tous les taxons étudiés sont actuels, ils devront être placés sur une même ligne horizontale qui représente le temps présent (il existe donc une contrainte sur la longueur des branches). En effet, les sommes des longueurs des branches qui relient différents taxons actuels à leur ancêtre commun doivent être égales. Par exemple, pour B et C, t wB et t wC sont égaux. Pour B et E, t wB + t yW et égal à t xE + t yX
(Figure 3C). Ceci est tout simplement dû au fait que le temps s’écoule de la même façon pour toutes les lignées. Ceci peut paraître très trivial, mais nous verrons que beaucoup d’erreurs naissent de l’oubli de cette évidence.
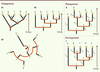 | Figure 3.
Les différentes représentations d’un arbre phylogénétique.
|
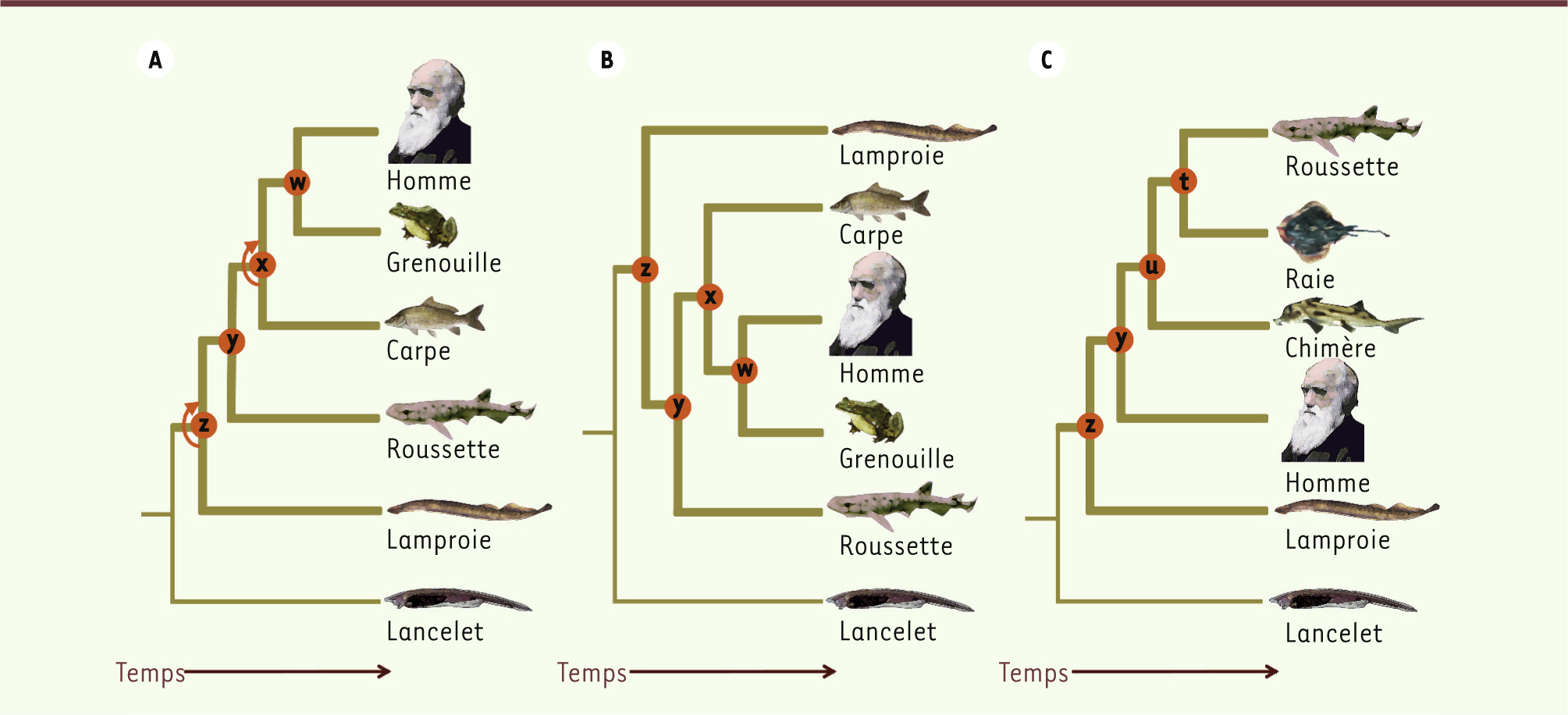
 | Figure 4.
Trois phylogénies des vertébrés. A. B. Deux représentations équivalentes du même arbre phylogénétique. C. Un autre échantillonnage des espèces donne un arbre phylogénétique compatible mais différent. |
|
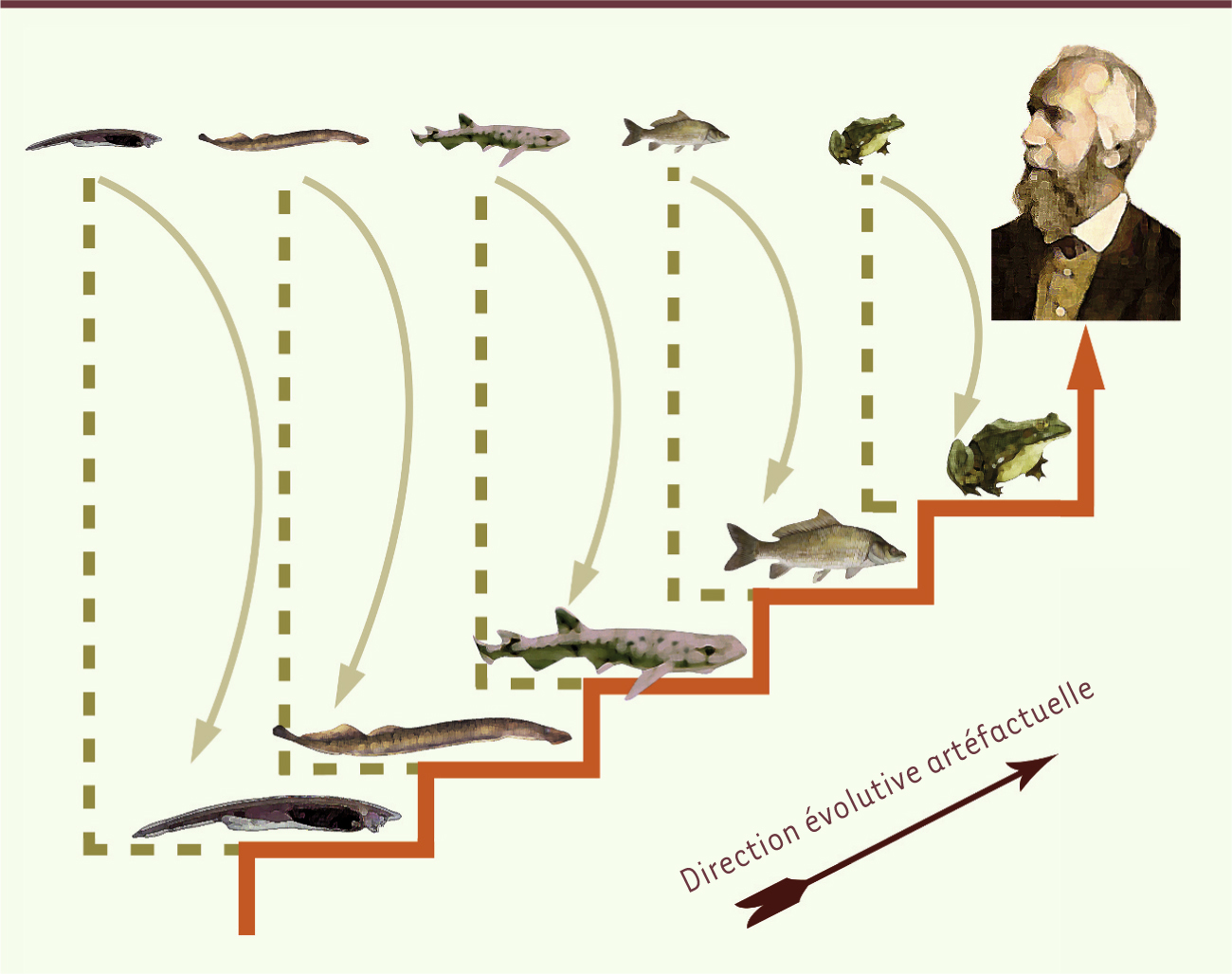
Bien lire un arbre phylogénétique La rotation des branches à un nœud ne change pas la topologie de l’arbre Considérons un arbre phylogénétique de vertébrés (Figure 4A). Il indique que nous sommes plus proches parents d’une grenouille (ancêtre commun w) que d’une carpe (ancêtre commun x), et que nous sommes encore plus éloignés des requins (ancêtre commun y). Le vertébré le plus éloigné de l’homme est la lamproie (ancêtre commun z). Si nous lisons l’arbre de feuille en feuille, nous recréons de façon artéfactuelle une échelle des êtres : la lamproie est à la base, le requin au-dessus, puis viennent le poisson, la grenouille et enfin l’homme. Cette lecture linéaire conduit à une interprétation de l’évolution par paliers, du plus primitif au plus évolué : un vertébré sans mâchoires (la lamproie) semble avoir donné naissance à un vertébré à mâchoires au squelette cartilagineux (le requin), dont serait issu un poisson osseux (la carpe), qui se serait transformé en tétrapode (la grenouille) et qui aurait finalement évolué en homme. Non seulement cette vision est fausse, mais de plus, elle n’est en rien inscrite dans cette phylogénie. Pour le montrer, effectuons des rotations aux nœuds z et x, ce qui ne change en rien la topologie de l’arbre. La nouvelle représentation obtenue présente exactement les mêmes relations de parenté, mais ne permet plus d’organiser les noms d’espèces dans une reconstruction a priori des étapes de l’évolution (Figure 4B). La persistance du concept d’échelle des êtres conduit inconsciemment le lecteur à émonder l’arbre et à rabattre les feuilles au niveau des nœuds internes (Figure 5). Cette erreur inconsciente reconstitue une échelle qui indique une direction évolutive artéfactuelle. C’est pourquoi il est recommandé de ne pas placer l’espèce humaine à une extrémité d’un arbre phylogénétique comme dans la représentation la plus fréquemment choisie (Figure 4A), et de préférer des représentations comme celle de la
Figure 4B
qui évitent de conforter une vision de l’évolution obsolète, mais encore très répandue, héritée de Haeckel.
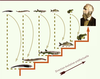 | Figure 5.
La rémanence de l’échelle des êtres conduit à la lecture erronée des arbres phylogénétiques.
|
Chassez le primitif, il revient à la base Plus ou moins conscients de ce biais méthodologique, beaucoup d’auteurs évitent - à raison - le terme primitif, mais incluent dans leurs études comparatives des espèces qu’ils qualifient de « lignée basale » : lamproies pour les vertébrés ou requins pour les gnathostomes (vertébrés à mâchoires). Une lignée basale est définie comme une lignée qui s’isole particulièrement tôt dans l’arbre phylogénétique (Figure 4A
et
4B) et qui serait donc plus ancienne et moins évoluée. Est-ce vrai ? Et bien non, car si deux nœuds peuvent être plus ou moins basaux l’un par rapport à l’autre, l’identification d’une lignée basale n’a pas de sens, car elle dépend de l’échantillonnage des taxons. Encore une fois, l’erreur consiste à reprendre le concept d’échelle des êtres dans un cadre phylogénétique. Prenons l’exemple d’une phylogénie des vertébrés comprenant trois espèces de chondrichthyens (poissons cartilagineux), un homme et une lamproie (Figure 4C). Dans cette phylogénie, l’homme apparaît comme la lignée basale des gnathostomes sans que pour cela on imagine qu’il représente un organisme ancien, primitif qui a évolué en requin. La lignée basale n’a pas de définition absolue, car elle est dépendante de l’échantillonnage des espèces. Les lignées évolutives n’ont pas de début : tout organisme a des parents dont il descend. En revanche, elles peuvent avoir une fin : la plupart des espèces se sont éteintes au cours de l’évolution. Seuls les groupes monophylétiques ont un âge - représenté par celui de leur ancêtre commun le plus récent -, ils peuvent donc être plus ou moins anciens. Si une roussette n’est pas plus ancienne ou récente qu’un homme, il est vrai que la classe des chondrichthyens à laquelle elle appartient est bien plus ancienne que le genre Homo auquel appartient l’homme. Mais attention, la classe des sarcoptérygiens à laquelle appartient l’homme est bien plus ancienne que le genre Scyliorhinus auquel appartient la roussette. |
Penser l’évolution dans un cadre phylogénétique permet de mettre en évidence des scénarios évolutifs contre intuitifs Explicitons quelques exemples concernant l’évolution des vertébrés. De trop nombreux biologistes considèrent l’histoire évolutive des vertébrés d’une façon intuitive et entachée par la rémanence de concepts scalistes. Si les erreurs les plus courantes concernent des caractères morphologiques comme la mâchoire des gnathosthomes, le squelette des ostéichthyens (poissons osseux) ou le poumon des tétrapodes, nous verrons que certains caractères moléculaires ne sont pas à l’abri d’une interprétation biaisée de leur histoire évolutive. Les agnathes actuels (vertébrés sans mâchoires), groupe constitué par les lamproies et myxines, ne présentent pas l’état ancestral qui précède l’apparition des mâchoires, mais un appareil buccal complexe, partageant une même origine embryologique avec celui des gnathostomes, et qui leur permet de se fixer plus ou moins longtemps sur des animaux afin d’arracher des morceaux de chair ou d’aspirer leur sang. L’état ancestral est difficile à reconstituer car l’organisation de l’appareil buccal des agnathes et celle des gnathostomes sont extrêmement différentes, et les fossiles ne permettent pas de se faire une image claire des états intermédiaires, depuis un ancêtre commun, dans deux directions évolutives radicalement différentes. Les chondrichthyens (requins, raies et chimères) sont souvent présentés comme un état intermédiaire dans l’évolution des gnathostomes, état qui précède l’apparition du squelette minéralisé. Il est toutefois bien connu que le squelette des chondrichthyens peut être minéralisé, mais de façon différente de celui des ostéichthyens. Par ailleurs, le squelette des chondrichthyens n’est en rien inférieur à celui des ostéichthyens, qu’il s’agisse de ses propriétés mécaniques ou de l’adaptation de l’animal à son milieu [
19]. Il suffit d’avoir vu un grand requin blanc en action pour s’en convaincre. La vessie natatoire des actinoptérygiens (poissons à nageoires rayonnées) a une fonction essentiellement hydrostatique, mais peut aussi parfois être impliquée dans la respiration et l’audition. Elle fut longtemps considérée comme l’état ancestral dont dérivent les poumons des sarcoptérygiens (dipneustes, cœlacanthes et tétrapodes). L’existence chez un actinoptérygien, le polyptère, de poumons qui ressemblent par bien des aspects à ceux des sarcoptérygiens, suggère qu’en fait l’ancêtre commun des actinoptérygiens avait des poumons qui auraient évolué en vessie natatoire [
20,
21]. Les erreurs induites par une lecture linéaire « vers l’homme » des taxons terminaux des arbres ne se limitent pas aux caractères morphologiques. Ainsi par exemple, les gènes Hox, impliqués dans la régionalisation des embryons, sont organisés en un ou plusieurs complexes multigéniques. L’identification de quatre complexes Hox chez les amphibiens, les oiseaux et les mammifères [
22], et d’un seul complexe Hox chez l’amphioxus [
23] laissa tout d’abord penser que l’organisation en quatre complexes représentait une solution optimisée partagée par l’ensemble des vertébrés. Cependant, il apparut bientôt que les poissons téléostéens, qui ont connu au moins une duplication supplémentaire de leur génome présentent (aux pertes secondaires près) au moins deux fois plus de complexes Hox que les tétrapodes [
24]. Plus récemment, quatre complexes furent identifiés chez les chimères [
25], mais trois seulement chez les requins et les raies [
26,
27]. L’analyse phylogénétique de ces résultats montre que l’ancêtre de tous les gnathostomes (y sur la
Figure 4
) possédait quatre complexes Hox. Dès lors, l’organisation en quatre complexes chez l’homme (et chez tous les tétrapodes) apparaît comme le maintien d’un état ancestral. À l’inverse, la multiplication des complexes chez les téléostéens, ainsi que la perte d’un complexe chez les requins et les raies apparaissent comme deux états dérivés spécifiques de ces groupes [
28]. En conclusion, seule la comparaison des espèces dans un cadre phylogénétique permet de reconstruire leur histoire évolutive, car toutes les espèces présentent une mosaïque de caractères qui ont plus ou moins évolué au cours du temps. Il n’existe donc pas d’espèces actuelles basales ou ancestrales, ni de « fossile vivant ». Enfin, seule l’étude d’un large échantillon d’espèces, au-delà des quelques espèces dites modèles, permet de reconstruire avec précision l’histoire de la vie sur terre. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Nous voudrions remercier Jacques Van Helden qui a attiré notre attention sur le passage de « L’origine des espèces » cité dans notre manuscrit. Nous remerçions également Véronique Borday-Birraux, Mélanie Debiais-Thibaud, Silvan Oulion, Romain Nattier et nos collègues du service d’enseignement de biologie animale de l’université Paris-Diderot pour nos nombreuses discussions passionnées consacrées à l’enseignement de l’évolution.
|
Footnotes |
1.
Avise
JC
,
Wollenberg
K.
Phylogenetics and the origin of species . Proc Natl Acad Sci USA.
1997; ; 94 : :7748.–7755. 2.
Avise
JC.
Phylogeography: the history and formation of species . Harvard: : Harvard University Press; , 2000. 3.
Delsuc
F
,
Brinkmann
H
,
Philippe
H.
Phylogenomics and the reconstruction of the tree of life . Nat Rev Genet.
2005; ; 6 : :361.–375. 4.
Lopez
P
,
Casane
D
,
Philippe
H.
Phylogénie et évolution moléculaires . Med Sci (Paris).
2002; ; 18 : :1146.–1154. 5.
Forterre
P
,
Gribaldo
S
,
Brochier
C.
Luca. À la recherche du plus proche ancêtre commun universel . Med Sci (Paris).
2005; ; 21 ; :860.–865. 6.
Guichard
C
,
Pedruzzi
E
,
Fay
M
, et al.
Les Nox/Duox : une nouvelle famille de NADPH oxydases . Med Sci (Paris).
2006; ; 22 : :953.–959. 7.
Da Lage
JL
,
Danchin
EG
,
Casane
D.
Where do animal alpha-amylases come from? An interkingdom trip . FEBS Lett.
2007; ; 581 : :3927.–3935. 8.
Colina
R
,
Casane
D
,
Vasquez
S
, et al.
Evidence of intratypic recombination in natural populations of hepatitis C virus . J Gen Virol.
2004; ; 85 : :31.–37. 9.
Peeters
M
,
Chaix
ML
,
Delaporte
E.
Phylogenie des SIV et des VIH : mieux comprendre l’origine des VIH . Med Sci (Paris).
2008; ; 24 : :621.–628. 10.
Douzery
EJ
,
Delsuc
F
,
Philippe
H.
Les datations moléculaires à l’heure de la génomique . Med Sci (Paris).
2006; ; 22 : :374.–380. 11.
Chardin
P.
Nous sommes tous de « race » africaine !
Med Sci (Paris).
2008; ; 24 : :205.–207. 12.
Nee
S
,
Holmes
EC
,
Rambaut
A
,
Harvey
PH.
Inferring population history from molecular phylogenies . Philos Trans R Soc Lond B Biol Sci.
1995; ; 349 : :25.–31. 13.
Arnold
ML.
Natural hybridization and evolution . New York: : Oxford University Press; , 1997. 14.
Halverson
KL.
Improving tree-thinking one learnable skill at a time . Evolution.
2011; ; 4 : :95.–106. 15.
Meisel
RP.
Teaching tree-thinking to undergraduate biology students . Evolution.
2010; ; 3 : :621.–628. 16.
Baum
DA
,
Smith
SD
,
Donovan
SS.
Evolution. The tree-thinking challenge . Science.
2005; ; 310 : :979.–980. 17.
Omland
KE
,
Cook
LG
,
Crisp
MD.
Tree thinking for all biology: the problem with reading phylogenies as ladders of progress . Bioessays.
2008; ; 30 : :854.–867. 18.
Darwin
CR.
On the origin of species by means of natural selection, or the preservation of favoured races in the struggle for life. (1st ed) , 1st ed . London: : John Murray; , 1859. 19.
Cuny
G.
Les requins sont-ils des fossiles vivants ?
Les Ulis : EDP Sciences. , 2002. 20.
Lechleuthner
A
,
Schumacher
U
,
Negele
RD
,
Welsch
U.
Lungs of Polypterus and Erpetoichthys . J Morphol.
1989; ; 201 : :161.–178. 21.
Liem
KF.
Form and function of lungs: the evolution of air breathing mechanisms . Am Zool.
1988; ; 28 : :739.–759. 22.
Scott
MP.
Vertebrate homeobox gene nomenclature . Cell.
1992; ; 71 : :551.–553. 23.
Garcia-Fernandez
J
,
Holland
PW.
Archetypal organization of the amphioxus Hox gene cluster . Nature.
1994; ; 370 : :563.–566. 24.
Amores
A
,
Force
A
,
Yan
YL
, et al.
Zebrafish hox clusters and vertebrate genome evolution . Science.
1998; ; 282 : :1711.–1714. 25.
Ravi
V
,
Lam
K
,
Tay
BH
, et al.
Elephant shark (Callorhinchus milii) provides insights into the evolution of Hox gene clusters in gnathostomes . Proc Natl Acad Sci USA.
2009; ; 106 : :16327.–16332. 26.
Oulion
S
,
Debiais-Thibaud
M
,
d’Aubenton-Carafa
Y
, et al.
Evolution of Hox gene clusters in gnathostomes: insights from a survey of a shark (Scyliorhinus canicula) transcriptome . Mol Biol Evol.
2010; ; 27 : :2829.–2838. 27.
Oulion
S
,
Borday-Birraux
V
,
Debiais-Thibaud
M
, et al.
Evolution of repeated structures along the body axis of jawed vertebrates, insights from the Scyliorhinus canicula Hox code . Evol Dev.
2011; ; 13 : :247.–259. 28.
Oulion
S
,
Laurenti
P
,
Casane
D.
Organisation des gènes Hox : l’étude de vertébrés non-modèles mène à un nouveau paradigme . Med Sci (Paris).
2012; ; 28 : :350.–353. |