| |
| Med Sci (Paris). 2012 December; 28(12): 1067–1069. Published online 2012 December 21. doi: 10.1051/medsci/20122812016.Observer la cellule et ses structures membranaires à l’échelle du nanomètre Amélie Leforestier,1* Nicolas Lemercier,1 and Françoise Livolant1 1CNRS UMR 8502, laboratoire de physique des solides, bâtiment 510, Université Paris-Sud, 91405Orsay Cedex, France MeSH keywords: Animaux, Membrane cellulaire, ultrastructure, Cellules, cytologie, Humains, Protéines membranaires, analyse, composition chimique, Microscopie à force atomique, méthodes, Nanotechnologie, Phospholipides |
L’imagerie cellulaire et tissulaire connaît depuis une dizaine d’années des progrès spectaculaires grâce à l’exploitation des propriétés de la fluorescence. Elle permet une imagerie en trois dimensions (3D), voire 4D, de la cellule vivante à des résolutions toujours améliorées [
1–
3], et permet même la détection et le suivi de molécules uniques. Aussi riches soient-elles, ces approches ne disent rien sur la conformation des macromolécules, la nature de leurs interactions, ou leur organisation supramoléculaire ; d’où le besoin d’informations à plus haute résolution. La microscopie électronique à transmission (MET) est l’outil idéal d’investigation, même si le vide poussé dans lequel se propagent les électrons ne permet pas encore d’explorer la structure de la cellule vivante. Depuis trente ans, la cryo-MET s’est imposée comme un outil majeur en biologie structurale. Associée à des analyses d’images sophistiquées, elle permet d’obtenir des résolutions quasi atomiques de gros complexes macromoléculaires et de particules virales, isolés en solution [
4]. Mais elle reste limitée à des objets de petite dimension (idéalement 50 à 100 nm, au plus 500 à 700 nm). Bien qu’apparue à la même époque, la cryo-MET de sections vitreuses (CEMOVIS pour cryo-electron microscopy of vitreous sections) est une technique encore émergente que seuls quelques laboratoires dans le monde développent à l’heure actuelle. Elle consiste à geler les cellules à très basse température (≤ − 196 °C), suffisamment rapidement pour éviter la cristallisation de l’eau (vitrification), puis à réaliser à une température inférieure à la température de dévitrification de l’eau (− 135 °C), des cryosections ultraminces (25 à 50 nm) qui sont observées à − 180 °C sous de faibles doses d’électrons. On obtient ainsi des instantanés de la cellule dans son état natif, sans coloration ni marquage [
6]. Nous allons montrer ici ce que peut apporter cette méthode à l’étude des systèmes membranaires à l’aide de quelques exemples puisés dans différents types cellulaires [
7]. |
Visualiser les bicouches de phospholipides Les membranes qui délimitent cellules et compartiments cellulaires sont facilement identifiables sur les images de CEMOVIS lorsqu’elles sont observées en section parfaitement transversales. En optimisant les conditions d’imagerie, les deux feuillets de phospholipides peuvent alors être mis en évidence : les membranes se présentent comme deux lignes parallèles, denses aux électrons, correspondant aux têtes polaires des phospholipides, et séparées par une ligne claire correspondant à leurs chaînes aliphatiques (Figure 1). Au-delà de cette structure en bicouche déjà bien connue, on peut ainsi analyser les dissymétries d’épaisseur et de composition des deux feuillets, et découvrir la présence de membranes finement accolées au sein de plusieurs types cellulaires (au niveau des réservoirs alvéolaires calciques de la paramécie ou des corps multilamellaires de lignées cellulaires humaines par exemple [7]). De tels contacts étroits n’avaient jamais été détectés par manque de résolution spatiale. Cependant, ces contacts pourraient être fréquents au sein des cellules eucaryotes, ce qui soulève des interrogations sur leurs implications biologiques et, plus particulièrement, sur les mécanismes de fusion mis en jeu au sein de la cellule.
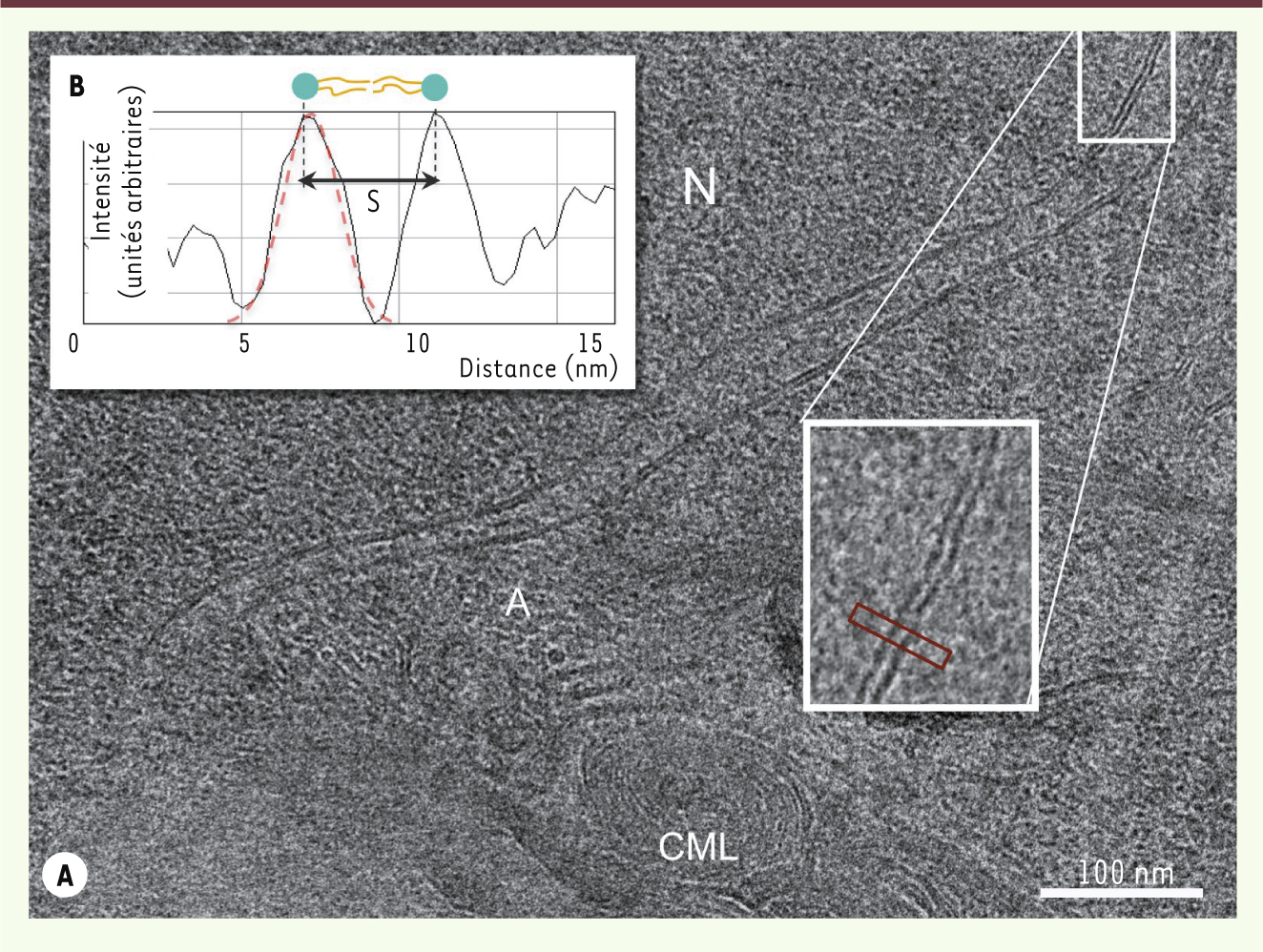
 | Figure 1.
Visualisation des structures membranaires. A. Vue d’une partie d’une cellule de la lignée colique humaine HT29. Le noyau (N) est limité par l’enveloppe nucléaire. Des filaments d’actine (A) et des corps multilamellaires (CML) sont visibles dans le cytoplasme. La structure de la membrane en double feuillet est bien visible. B. Le profil d’intensité, mesuré perpendiculairement à la membrane le long du rectangle rouge, permet de mesurer la distance (S) (ici 3,5 nm) séparant les deux rangées de têtes polaires des phospholipides. |
|
Analyser les réseaux de protéines transmembranaires Les membranes thylakoïdes1 photosynthétiques des chloroplastes fournissent un très bel exemple de réseau protéique fonctionnel ordonné à trois dimensions (Figure 2). La présence des protéines est détectée par une augmentation locale de l’intensité du signal dans la région des chaînes aliphatiques des phospholipides. La résolution atteinte permet d’analyser l’ordre entre protéines dans le plan des membranes aussi bien que dans le plan perpendiculaire aux empilements fonctionnels. La
Figure 2C
montre un exemple des détails accessibles par imagerie CEMOVIS. Les séries de photosystèmes I et II (flèches vertes) sont visibles au sein des bicouches alors que la lumière du thylakoïde présente des densités correspondant au complexe protéique OEC (oxygen evolving complex, flèches mauves), deux composants de la chaîne respiratoire. Alors qu’on connaît aujourd’hui à l’échelle atomique les structures de chacun des principaux complexes protéiques impliqués dans les réactions photosynthétiques, ce type d’approche permet d’appréhender leur organisation spatiale et leurs interactions in situ.
 | Figure 2.
Réseaux de protéines transmembranaires. A. Vue générale d’une algue unicellulaire ( Euglena gracilis). On reconnaît le noyau (N) avec ses chromosomes (Chr), une mitochondrie (M), un granule de stockage ou paramylon (P) et le chloroplaste (Chl). B. Détail d’un chloroplaste montrant l’empilement de thylakoïdes en vue transversale ; les régions luminales des thylakoïdes (l) alternent avec le stroma (S). C. Détail d’un empilement de thylakoïdes. Au sein des bicouches sont visibles des séries de protéines transmembranaires (photosystèmes I et II, flèches vertes). La lumière du thylakoïde (voir l sur panneau B) présente une densité correspondant au complexe protéique OEC ( oxygen evolving complex, flèches bleues). D. Par comparaison, un empilement de thylakoïdes d’épinard observé sur un cryotomogramme est présenté ici à la même échelle et permet d’obtenir directement des données 3D, mais avec une moindre résolution (reproduit de [
13]). |
L’imagerie cellulaire par CEMOVIS ouvre ainsi la voie à un niveau de résolution encore inexploré in situ, celle du nanomètre. Outre l’analyse des systèmes membranaires, cette méthode s’est déjà avérée particulièrement fructueuse pour l’étude de complexes macromoléculaires de grande taille (par exemple microtubules ou desmosomes [
8]) mais aussi de systèmes fibrillaires, tels que l’ADN dans le noyau des spermatozoïdes [
9], ou le nucléoïde bactérien. En couplant l’analyse d’images de cryosections (pour lesquelles les conditions d’obtention des images ont été optimisées et qui procurent ainsi des données haute résolution mais à 2D) et l’acquisition de données par cryo-tomographie [
10,
11] (qui permet de s’abstraire des problèmes de superposition, mais au prix d’une limitation de la résolution à quelques 4-7 nm [Figure 2D]), on peut espérer accéder prochainement à l’imagerie de la cellule entière à une résolution nanométrique, voire subnanométrique. L’une comme l’autre de ces méthodes ont l’avantage de ne requérir la présence d’aucun contrastant ce qui constitue un atout incontestable, puisque c’est la structure native de l’ensemble des molécules qui est observée. Néanmoins, elles sont toutes deux confrontées aux difficultés d’identification des composants de la cellule, pourtant indispensable pour dépasser la simple description structurale. Différentes voies de marquages possibles commencent à être envisagées (marqueurs clonables tels que la métallothionéine, pouvant lier des éléments lourds denses aux électrons [
12]) qui s’avèreront essentielles dans les années qui viennent pour que la méthode s’impose comme majeure dans le paysage de la biologie intégrative. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Trugnan
G
,
Fontanges
P
,
Delautier
D
,
Ait-Slimane
T.
FRAP, FLIP, FRET, BRET, FLIM, PRIM… De nouvelles techniques pour voir la vie en couleur !
Med Sci (Paris).
2004; ; 20 : :1027.–1034. 2.
Débarre
D
,
Pena
AM
,
Supatto
W
, et al.
Microscopies multi-harmoniques pour l’imagerie structurale de tissus intacts . Med Sci (Paris).
2006; ; 22 : :845.–850. 3.
Girard
PP
,
Forget
BC.
Microscopie de fluorescence à feuille de lumière . Med Sci (Paris).
2011; ; 27 : :753.–762. 4.
Chiu
W
,
Baker
ML
,
Almo
SC.
Structural biology of cellular machines . Trends Cell Biol.
2006; ; 16 : :144.–150. 5.
Zhou
ZH.
Atomic reolution cryo electron microscopy of macromolecular complexes . Adv Protein Chem Struct Biol.
2011; ; 82 : :1.–35. 6.
Al-Amoudi
A
,
Chang
JJ
,
Leforestier
A
, et al.
Cryo-electron microscopy of vitreous sections . EMBO J.
2004; ; 23 : :3583.–3588. 7.
Leforestier
A
,
Lemercier
N
,
Livolant
F.
Contribution of cryoelectron microscopy of vitreous sections to the understanding of biological membrane structure . Proc Natl Acad Sci USA.
2012; ; 109 : :8959.–8964. 8.
Al-Amoudi
A
,
Díez
DC
,
Betts
MJ
,
Frangakis
AS.
The molecular architecture of cadherins in native epidermal desmosomes . Nature.
2007; ; 450 : :832.–837. 9.
Sartori Blanc
N
,
Senn
A
,
Leforestier
A
, et al.
DNA in human and stallion spermatozoa forms local hexagonal packing with twist and many defects . J Struct Biol.
2001; ; 134 : :76.–81. 10.
Yahav
T
,
Maimon
T
,
Grosmann
E
, et al.
Cryo-electron tomography: gaining insight into cellular processes by structural approaches . Curr Opin Struct Biol.
2011; ; 21 : :670.–677. 11.
Hoenger
A
,
Bouchet-Marquis
C.
Cellular tomography . Adv Protein Chem Struct Biol.
2011; ; 62 : :2393.–2402. 12.
Mercogliano
CP
,
DeRosier
DJ.
Gold nanocluster formation using metallothionein: mass spectrometry and electron microscopy . J Mol Biol.
2006; ; 355 : :211.–223. 13.
Daum
B
,
Nicastro
D
,
Austin
J
, et al.
Arrangement of photosystem II and ATP synthase in chloroplast membranes of spinach and pea . Plant Cell.
2010; ; 22 : :1299.–1312. |