| |
| Med Sci (Paris). 2012 August; 28(8-9): 710–713. Published online 2012 August 22. doi: 10.1051/medsci/2012288013.L’évolution darwinienne d’un système génétique non conventionnel met en évidence un ancêtre potentiel de l’ARN Annabelle Gillig1* 1Biodesign Institute, Arizona State University, 1001 S. McAllister Ave., Tempe, AZ 85287, États-Unis MeSH keywords: Évolution biologique, Biopolymères, DNA-directed DNA polymerase, métabolisme, Évolution moléculaire, Banque de gènes, Modèles génétiques, Origine de la vie, Liaison aux protéines, ARN, composition chimique, génétique, Délétion de séquence, Relation structure-activité, Spécificité du substrat, Oses phosphates, Thrombine, Transcription génétique, effets des médicaments et des substances chimiques |
La question de l’apparition de la vie sur Terre est l’une des plus fascinantes dans le domaine scientifique. L’hypothèse d’un monde d’acides ribonucléiques (ARN) est fondée sur le principe que la vie sur Terre est apparue au cours des temps géologiques au travers de l’ARN, capable de catalyser des réactions chimiques et d’être le support d’une information génétique [
1]. Il n’est cependant pas évident que l’ARN fût le matériel génétique initial. En effet, les difficultés liées à la synthèse prébiotique du ribose, composant de l’ARN, et à la réplication non enzymatique de ce dernier, ont suscité un vif intérêt pour la théorie d’un monde pré-ARN. Cette théorie considère que l’ARN a été précédé dans l’évolution de la vie par un matériel génétique plus simple et plus accessible dans les temps primitifs [
2,
3]. Afin de comprendre l’origine et l’évolution de la vie, il est fondamental de déterminer les conditions nécessaires à l’apparition d’un monde d’ARN. Dans ce but, une étude systématique a été réalisée sur des systèmes d’acides nucléiques potentiellement naturels afin de déterminer leur capacité à former des appariements de type Watson-Crick et donc à transmettre l’information génétique [
4]. Un métabolisme primitif nécessite également que ces polymères soient capables de se replier dans des structures tertiaires stables ayant des fonctionnalités élaborées telles que la reconnaissance d’un ligand et la catalyse [
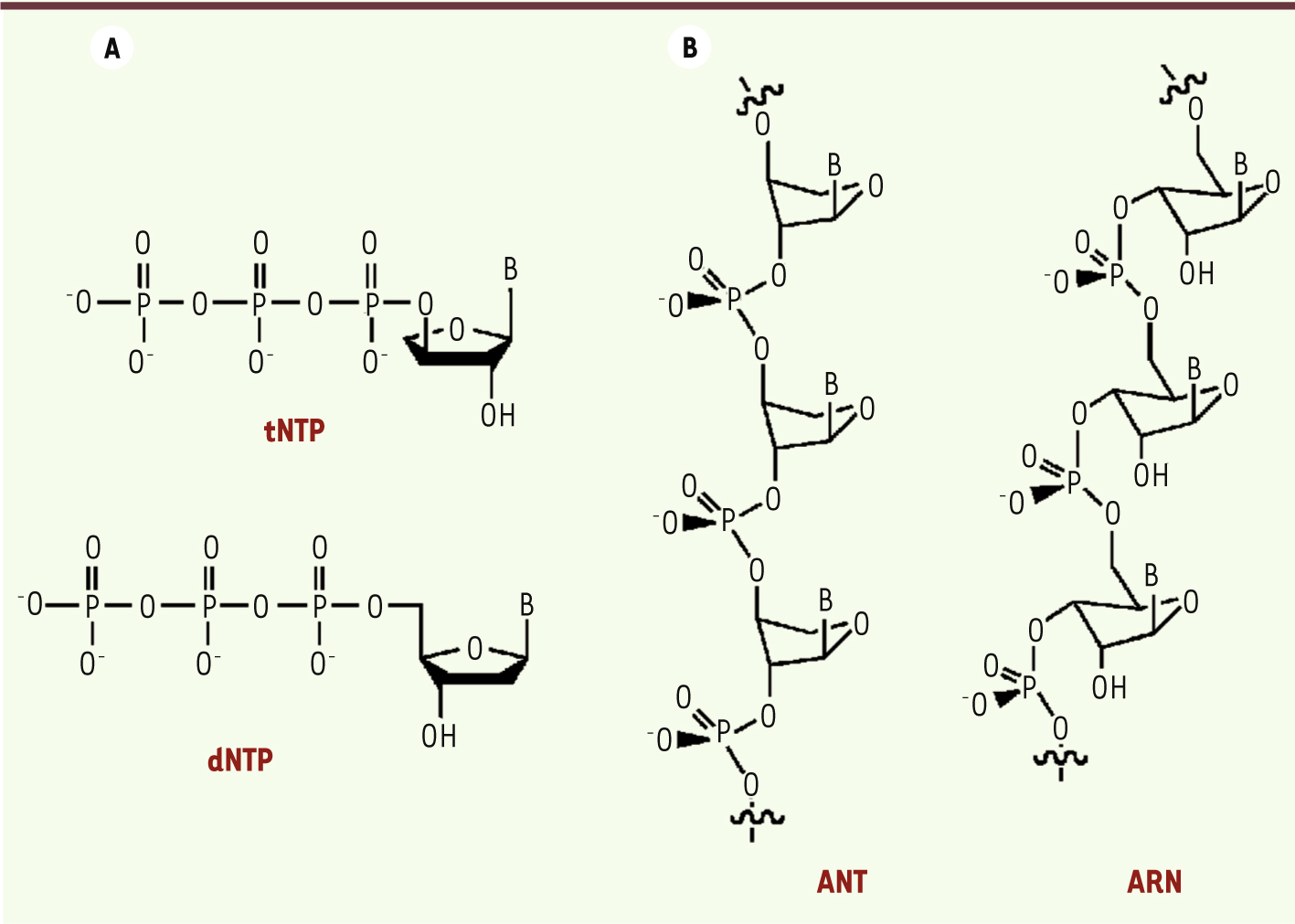
5]. Parmi les systèmes étudiés, l’acide nucléique (3’, 2’)-α-L-thréose (ANT,
Figure 1
) pourrait jouer le rôle de précurseur de l’ARN en raison de sa simplicité structurale - son squelette est constitué de thréoses qui ont un carbone de moins que les riboses de l’ARN - et de sa capacité à former des structures hélicoïdales stables avec des brins complémentaires d’ANT ou d’ARN [
6–
8].
 | Figure 1.
A. Structures des triphosphates d’acide nucléique (3’, 2’)-α-L-thréose (tNTP), et d’ADN (dNTP). B. Structures de nucléotides d’α-L-thréofuranosyl 3’→ 2’ liés (ANT), et de nucléotides de β-D-ribofuranosyl 5’→ 3’ liés (ARN) [
10]. |
La sélection in vitro, ou évolution moléculaire, est une méthode de choix dans l’étude des propriétés fonctionnelles des acides nucléiques. Cette approche a cependant toujours été limitée à l’ADN, à l’ARN ou à d’autres analogues structurels puisque ces derniers sont les seuls qui disposent d’enzymes connues capables de réaliser la transcription, la transcription inverse et l’amplification de l’information génétique. Appliquer cette méthode à un système génétique artificiel tel que l’ANT nécessite :
-
des polymérases capables de transcrire une librairie de séquences aléatoires d’ADN en ANT ;
-
une méthode de sélection qui permet d’isoler les individus présentant une fonction d’intérêt des autres membres de la librairie ;
-
une stratégie d’amplification des molécules fonctionnelles qui maintienne l’intégrité des séquences sélectionnées.
Dans une publication récente de Nature Chemistry [
9], l’équipe de John Chaput (Biodesign Institute, Tempe, Arizona) propose d’étudier cette problématique en utilisant la méthode de l’évolution darwinienne pour identifier un récepteur constitué d’ANT capable de lier un ligand. Ceci suppose que ce récepteur serait capable, tout comme l’ADN et l’ARN, de se replier en structures ayant des fonctions élaborées, condition nécessaire pour tout ancêtre potentiel de l’ARN dans un hypothétique monde pré-ARN. Pour ce faire, une librairie de polymères à base d’ANT a été préparée et soumise à une sélection darwinienne. |
Mise au point de la transcription de l’ADN en ANT Ishida et al. ont montré que l’ADN polymérase therminator est capable de transcrire efficacement un brin d’ADN de 80 nucléotides en ANT avec une haute fidélité [10,
11]. Cette enzyme n’est cependant pas capable de transcrire une librairie de séquences aléatoires d’ADN en ANT [
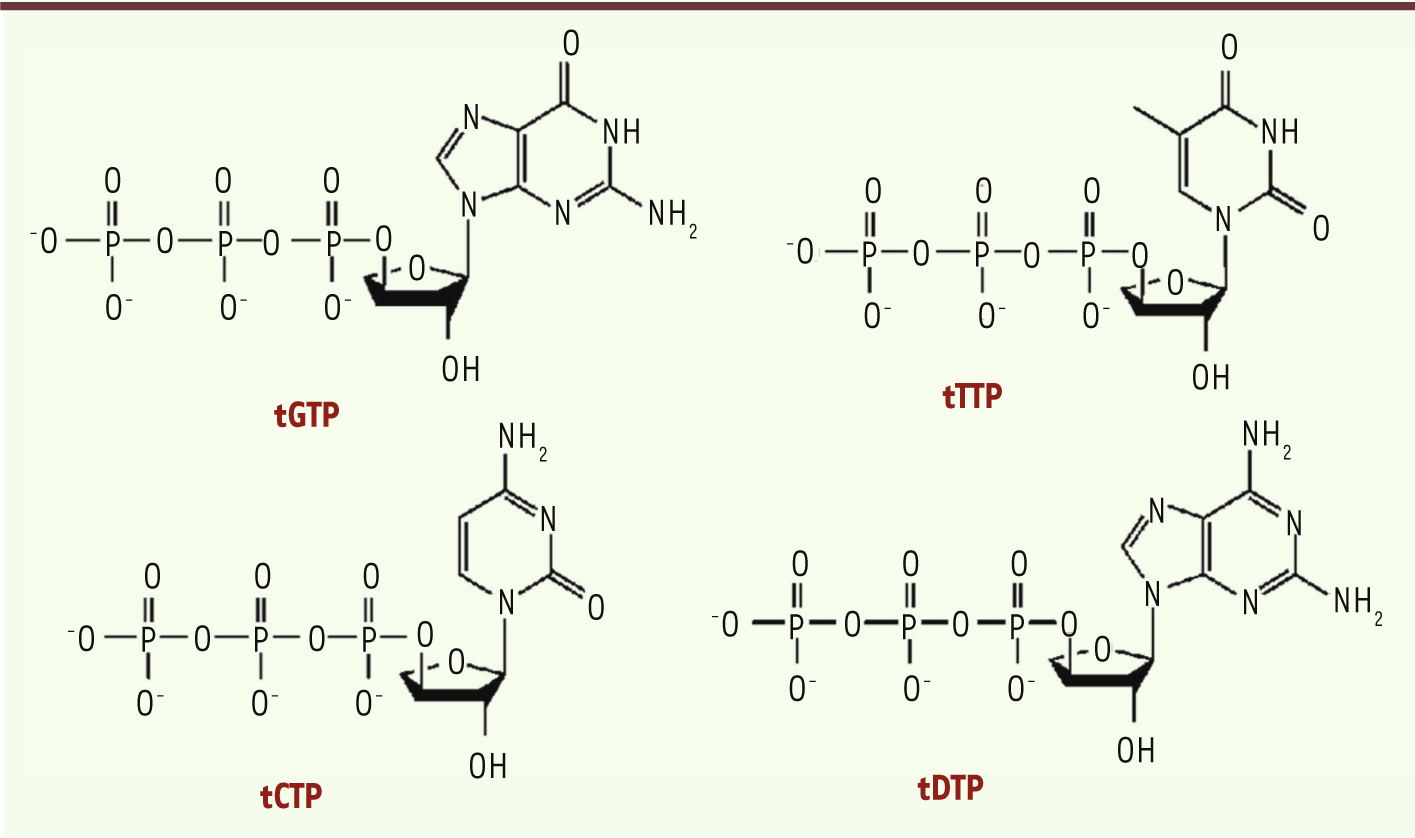
12], ce qui amène à penser que les brins d’ADN individuels utilisés dans l’étude de la polymérase therminator avaient peut-être tous une certaine particularité favorisant la synthèse de l’ANT. Afin de répondre à cette question, une série d’expériences visant à étudier la synthèse de l’ANT a été réalisée. Tout d’abord, les quatre triphosphates d’ANT (tNTP), éléments de base de la synthèse de brins d’ANT, ont été synthétisés (Figure 2). Dans cette étude, l’adénine du 3’-triphosphate d’adénosine a été remplacée par la diaminopurine qui apporte une stabilité thermodynamique à l’hétéroduplex ANT-ADN. L’élongation à partir d’une amorce d’ADN appariée à une librairie d’ADN (L1) contenant une région centrale de 50 nucléotides aléatoires, suivie d’une seconde amorce, a été réalisée avec 70 résidus d’ANT en utilisant la polymérase therminator. Cette première expérience a échoué, confirmant ainsi les résultats précédemment obtenus [12].
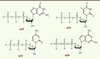 | Figure 2.
Structure des quatre résidus tNTP utilisés dans l’étude de la transcription de l’ADN en ANT [ 10]. |
Afin de déterminer quels nucléotides inhibent cette transcription, quatre nouvelles librairies d’ADN ont été synthétisées, et dans chacune d’entre elles, l’un des quatre tNTP a été remplacé par le triphosphate d’ADN (dNTP) correspondant. Cette expérience a démontré que la librairie d’ADN peut être transcrite efficacement lorsque tCTP est remplacé par dCTP. De plus, la comparaison de séquences représentatives de la librairie L1 avec les brins d’ADN individuels utilisés lors des études préliminaires sur l’enzyme therminator polymérase démontre une récurrence importante de motifs contenant une répétition du nucléotide G dans la librairie L1, alors que de tels motifs étaient inexistants dans les brins d’ADN individuels. Ceci suggère que des motifs comportant une répétition du nucléotide G dans la librairie d’ADN bloquent sa transcription en ANT. Deux nouvelles librairies d’ADN, L2 et L3, ont alors été synthétisées. L2 contient un nombre équivalent de nucléotides A, T et C dans la région de 50 nucléotides aléatoires. L3 contient 50 % de moins de nucléotide G comparé au nombre de nucléotides A, T et C dans cette même région. La diminution du nombre de résidus G a effectivement eu un impact considérable sur la transcription de l’ANT puisque dans chacun des cas, l’amorce d’ADN a été élonguée avec les 70 résidus d’ANT, atteignant un rendement de 60 % avec la librairie L2 qui ne contient pas de nucléotides G, et un rendement de 30 % avec la librairie L3 qui en contient deux fois moins que les trois autres nucléotides. Ce résultat est fondamental car, pour la première fois, il a été démontré que des librairies d’ANT, qui représentent des systèmes d’acides nucléiques ayant une structure différente de celle de l’ADN et de l’ARN, peuvent être générées tout comme l’ADN et l’ARN, par l’élongation d’une amorce d’ADN appariée à une librairie de séquences aléatoires d’ADN avec l’assistance d’une enzyme. |
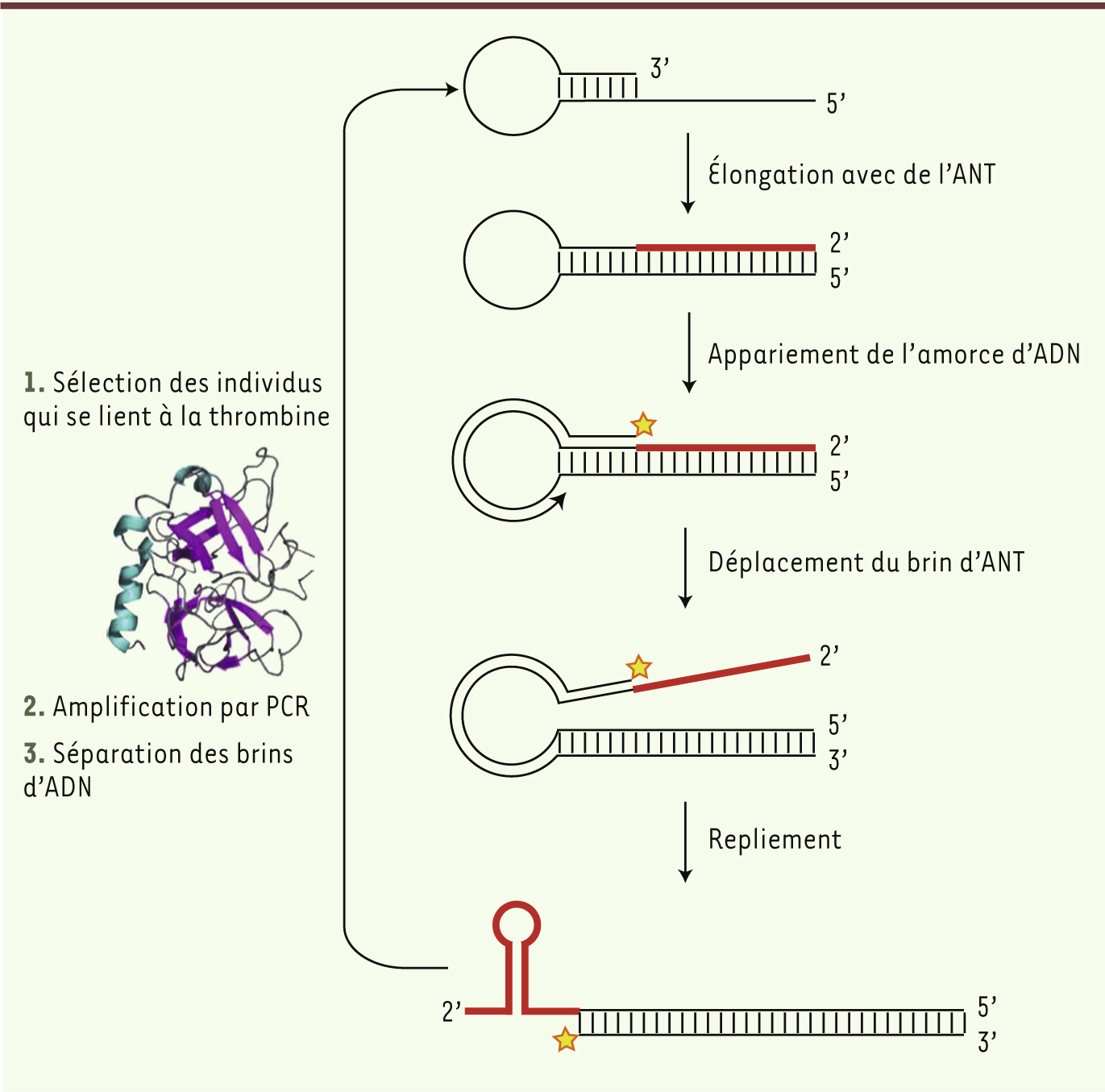
Mise au point d’une technique de sélection in vitro de molécules d’ANT fonctionnelles Afin de déterminer si une molécule d’ANT peut avoir une activité biologique comme celle des molécules d’ADN ou d’ARN, une sélection in vitro a été réalisée de manière à enrichir la librairie d’ANT obtenue précédemment en éléments ayant une activité de récepteur pour une cible biologique spécifique. Pour ce faire, une librairie d’ADN simple brin L2 (la plus efficace pour l’étape de transcription) liée par une structure tige-boucle à une amorce d’ADN qui lui est complémentaire a été construite (Figure 3). L’amorce d’ADN a ensuite été élonguée avec des résidus d’ANT, ce qui fournit une structure chimérique en épingle à cheveux, composée d’ANT et d’ADN. Le brin d’ANT apparié à son brin d’ADN complémentaire a ensuite été déplacé en ajoutant une autre amorce d’ADN s’appariant à l’ADN de la structure tige-boucle de départ. Par cette approche, la molécule d’ANT qui correspond au phénotype, c’est-à-dire aux propriétés de la molécule, est physiquement liée à son ADN double brin complémentaire qui, lui, correspond au génotype, c’est-à-dire à l’information génétique de départ. La librairie de molécules d’ANT et d’ADN fusionnées a ensuite été mise en présence d’une cible biologique spécifique, la thrombine humaine. Les molécules ayant une activité de récepteur pour la thrombine s’y sont fixées puis ont été récupérées et amplifiées par PCR (polymerase chain reaction) pour fournir une nouvelle librairie d’ADN simple brin liée à une amorce d’ADN par une structure tige-boucle. La nouvelle librairie ainsi réalisée constitue l’élément de départ pour un second tour de sélection. Après chaque tour de sélection, la librairie obtenue est plus enrichie en molécules capables de se fixer à la thrombine par rapport au cycle précédent. Cette méthode est particulièrement ingénieuse, car la liaison physique entre la molécule d’ANT et son ADN complémentaire permet d’éviter une étape de transcription réverse pour reconvertir les molécules d’ANT en ADN après l’étape de sélection. Par cette approche, plusieurs tours de sélection et d’amplification ont pu être réalisés sur la librairie L2. En outre, le fait que les polymères d’ANT ainsi produits ne contiennent pas de résidus de cytidine n’était pas un souci majeur, car il est tout à fait probable que ces derniers n’existaient pas dans le premier matériel génétique.
 | Figure 3.
Stratégie de sélection in vitro mise au point pour identifier des molécules d’ANT capables de reconnaître la thrombine humaine [ 9]. |
|
Étude de l’activité des molécules sélectionnées Après trois tours de sélection, l’ADN de la librairie enrichie en molécules présentant une affinité pour la thrombine a été séquencé afin d’étudier la diversité des molécules sélectionnées. Dix séquences représentatives de cette librairie ont été synthétisées individuellement par élongation d’une amorce d’ADN appariée à un brin d’ADN synthétique avec des résidus d’ANT suivie d’une purification par électrophorèse sur gel dénaturant. La mesure de leur affinité pour la thrombine par électrophorèse par capillarité démontre que ces molécules d’ANT ont une activité similaire à celle des molécules d’ADN et d’ARN sélectionnées de la même manière. Afin de déterminer quelle partie de la molécule d’ANT est responsable de l’interaction avec la thrombine, des études plus approfondies ont été menées sur la molécule présentant la plus grande affinité pour la thrombine parmi les dix sélectionnées. Des variantes de cette molécule d’ANT ont été synthétisées, chacune ayant subi la délétion d’une région de la séquence originale. La comparaison de l’affinité de chacune de ces variantes pour la thrombine a permis d’identifier une région de 41 nucléotides responsable de l’interaction avec la thrombine. Afin de confirmer ces résultats, une série de contrôles ont été menés. Le premier contrôle consiste à fixer la molécule d’ANT sélectionnée à une autre séquence d’ADN que celle qui lui est complémentaire. Dans ce cas, le composé obtenu présente la même affinité pour la thrombine que la molécule originale, ce qui démontre que c’est la partie composée d’ANT qui est responsable de l’interaction avec la thrombine. De plus, aucune activité n’a été observée pour des molécules aléatoires d’ANT n’ayant pas subi l’étape de sélection, ce qui veut dire que c’est grâce à cette étape de sélection que des molécules actives sont identifiées. Par ailleurs, l’amorce d’ADN ne présente aucune activité lorsqu’elle n’est pas liée à l’ANT et, enfin, la molécule d’ANT n’a pas d’affinité pour d’autres protéines telles que la BSA (bovine serum albumin) ou la streptavidine. Ainsi, la sélection in vitro a permis d’identifier des molécules d’ANT qui se lient à la thrombine avec une grande affinité et de manière spécifique. |
Un hypothétique monde d’ANT ? Le développement d’une méthodologie efficace pour l’évolution in vitro de molécules d’ANT est un processus qui s’est établi sur une décennie. Par conséquent, l’identification d’un système qui soit à la fois structurellement plus simple que l’ARN de manière à ce que sa synthèse soit compatible avec les composants du monde prébiotique, et capable d’évolution darwinienne, est un défi plus qu’impressionnant. Par une méthode de sélection in vitro basée sur le principe de l’évolution darwinienne, il a été établi que des molécules d’ANT peuvent avoir des fonctions aussi complexes que celles de l’ARN, ce qui suggère qu’il puisse être l’ancêtre du système génétique conventionnel basé sur ce dernier. Ainsi, la sélection in vitro s’est révélée être un outil remarquable qui pourra être utilisé dans de futures études visant à renforcer la théorie de l’ANT comme ancêtre de l’ARN dans un hypothétique monde d’ANT. |
L’auteur déclare n’avoir aucun lien d’intérêts concernant les données publiées dans cet article.
|
1.
Gilbert
W.
The RNA world . Nature.
1986; ; 319 : :618.. 2.
Joyce
GF
,
Schwartz
AW
,
Miller
SL
,
Orgel
LE
. The case for an ancestral genetic system involving simple analogues of the nucleotides . Proc Natl Acad Sci USA.
1987; ; 84 : :4398.–4402. 3.
Joyce
GF.
The rise and fall of the RNA world . New Biol.
1991; ; 3 : :399.–407. 4.
Eschenmoser
A.
Chemical etiology of nucleic acid structure . Science.
1999; ; 284 : :2118.–2124. 5.
Benner
SA
,
Ellington
AD
,
Tauer
A.
Modern metabolism as a palimpsest of the RNA world . Proc Natl Acad Sci USA.
1989; ; 86 : :7054.–7058. 6.
Schöning
KU
,
Scholz
P
,
Guntha
S
, et al.
Chemical etiology of nucleic acid structure: the α-L-threofuranosyl-(3’-2’) oligonucleotide system . Science.
2000; ; 290 : :1347.–1351. 7.
Orgel
LE.
A simpler nucleic acid . Science.
2000; ; 290 : :1306.–1307. 8.
Ebert
MO
,
Mang
C
,
Krishnamurthy
R
, et al.
The structure of a TNA-TNA complex in solution : NMR study of the octamer duplex derived from α-L-threofuranosyl-(3’-2’)-CGAATTCG . J Am Chem Soc.
2008; ; 130 : :15105.–15115. 9.
Yu
H
,
Zhang
S
,
Chaput
JC.
Darwinian evolution of an alternative genetic system provides support for TNA as an RNA progenitor . Nat Chem.
2012; ; 4 : :183.–187. 10.
Horhota
A
,
Zou
K
,
Ichida
JK
, et al.
Kinetic analysis of an efficient DNA-dependent TNA polymerase . J Am Chem Soc.
2005; ; 127 : :7427.–7434. 11.
Ichida
JK
,
Zou
K
,
Horhota
A
, et al.
An in vitro selection system for TNA . J Am Chem Soc.
2005; ; 127 : :2802.–2803. 12.
Ichida
JK
,
Horhota
A
,
Zou
K
, et al.
High fidelity TNA synthesis by therminator polymerase . Nucleic Acids Res.
2005; ; 33 : :5219.–5225. |