| |
| Med Sci (Paris). 2012 April; 28(4): 435–437. Published online 2012 April 25. doi: 10.1051/medsci/2012284023.Chroniques génomiques The sound of silence Bertrand Jordan1* 1CoReBio PACA, case 901, parc scientifique de Luminy, 13288Marseille Cedex 9, France MeSH keywords: Cartographie chromosomique, méthodes, Maladie, génétique, Évolution moléculaire, Exons, Génie génétique, effets indésirables, Étude d'association pangénomique, Humains, Modèles biologiques, Mutagenèse, physiologie, Cadres ouverts de lecture, Mutation ponctuelle, Polymorphisme de nucléotide simple |
Il ne s’agit pas ici, naturellement, du silence de la belle chanson de Simon et Garfunkel1, mais d’un ensemble de données récentes indiquant que les « mutations silencieuses » ne sont pas si silencieuses que cela, et que nombre d’entre elles pourraient jouer un rôle dans diverses pathologies humaines. Ce thème a été abordé dans un récent numéro de Nature Medicine [1], et je souhaite l’exposer ici sous l’angle de ses retombées en génétique médicale. |
Les surprises de l’optimisation Il est devenu courant, en biotechnologie, d’« optimiser » la séquence d’ADN que l’on veut faire exprimer par un microorganisme en vue de la production industrielle d’une protéine. Optimiser, c’est-à-dire modifier la séquence de l’ADN introduit afin qu’elle comporte, parmi les différents codons synonymes possibles, ceux qui sont les plus fréquemment utilisés par l’hôte. Cette approche s’est généralisée pour la production de protéines, et est également employée en thérapie génique, avec toujours le même but : favoriser l’obtention de la quantité la plus grande possible de la protéine thérapeutique. Mais à l’occasion, on découvre certaines bizarreries. Un des premiers exemples, publié en 2007 [2], concerne la protéine MDR1 (multidrug resistance protein 1), une glycoprotéine (de la superfamille des transporteurs ABC, ATP binding cassette) impliquée dans la résistance aux médicaments. Elle a été exprimée après transfection à partir d’une séquence nucléotidique contenant dans l’exon 26 soit ATC (version sauvage), soit ATT, deux codons correspondant au même acide aminé, l’isoleucine. Les niveaux d’ARN messagers obtenus sont comparables dans les deux cas, mais les propriétés de la protéine obtenue diffèrent en termes de sensibilité à différents inhibiteurs et de transport de drogues. L’explication à laquelle arrivent les auteurs est que le rythme de la traduction de l’ARN messager codant pour MDR1 est modifié pour la séquence « mutante » en raison d’une abondance très différente des ARN de transfert correspondant à ces deux codons. Ainsi, la protéine codée par la séquence contenant un ATT (le mutant synonyme et censément silencieux) n’est pas correctement repliée et donc ne présente pas une activité normale. Un autre exemple publié peu avant [3] concerne le gène COMT (catechol-O-methyltransferase), codant pour un enzyme de dégradation des catécholamines. Ici, une substitution « silencieuse » aboutit à une très importante réduction du niveau d’expression de la protéine, qui baisse d’un facteur vingt. Les auteurs montrent que cela est presque certainement dû à un changement de structure secondaire de l’ARN messager qui favorise sa dégradation et/ou le rend moins facilement traduisible. À partir de ces deux cas, qui ont été suivis de nombreux autres, il devient clair que « synonyme » ne veut pas forcément dire « silencieux ». |
Cas isolés, ou phénomène général ? Ces deux exemples sont certes intéressants, mais quelle est la fréquence de tels phénomènes ? Lors de la recherche des causes d’une pathologie, on tend généralement à ne tenir compte que des mutations qui modifient la séquence de la protéine, mutations non-sens ou faux-sens. Faut-il maintenant tenir aussi compte des mutations synonymes ? Il semble bien que la réponse soit oui. Je vais résumer dans ce but deux articles récents. L’un d’eux [4] décrit les résultats d’une analyse fondée sur la construction d’une base de données incluant toutes les associations publiées entre maladies humaines et snip (quel qu’en soit le type). Pour ce faire, les auteurs ont dépouillé plus de deux mille publications, et ont répertorié 21 429 mutations associées de manière significative à diverses affections. Ils se sont limités ensuite aux snip pour lesquels le rapport des chances d’association (odds ratio) a été publié et qui, de plus, sont répertoriés dans le projet HapMap 3 [5] : plus de cinq mille mutations ont été extraites. Ils ont pu alors examiner quelles catégories de snip s’avèrent être associées avec une pathologie. Sans surprise, les snip les plus fréquemment retrouvés sont ceux qui provoquent l’apparition d’un codon de terminaison : 2,77 % de tous les snip catalogués dans HapMap ont déjà été associés avec une maladie. Les snip faux-sens, eux, sont impliqués à 1,46 % ; mais surtout, les snip synonymes s’avèrent associés à 1,26 % (Tableau I).
Table I.
| Type de snip codant |
Répertoriés dans HapMap [5] |
Associés à une pathologie [4] |
| Non-sens |
613 |
17 (2,77 %) |
| Faux-sens |
30 256 |
442 (1,46 %) |
| Synonyme |
21 813 |
257 (1,26 %) |
Nombre de mutations dans les régions codantes associées à une pathologie, en fonction du type de mutation. Tableau établi à partir des données rapportées par Chen et al. [ 4]. Les snip présentés sont ceux répertoriés dans HapMap [ 5]. |
Si l’on en croit ces chiffres, les mutations « silencieuses » deviennent un acteur important dans le domaine des pathologies génétiques. Il faut certes noter que les travaux qui ont été dépouillés pour constituer ce catalogue rapportent des associations entre snip et maladie, et non (en général) des cas où il a été démontré directement que le variant était la cause de la maladie. On pourrait donc imaginer que certains des snip synonymes qui apparaissent dans le Tableau I ne soient en fait que des marqueurs, en déséquilibre de liaison avec la vraie mutation causale située à proximité sur le génome. Les auteurs avancent plusieurs arguments assez convaincants à l’encontre de cette interprétation [4], notamment le fait que leur échantillon résulte d’une analyse détaillée (curation) de chacune des deux mille publications prises en compte, et que si les auteurs de ces articles avaient repéré des associations significatives impliquant des snip faux-sens, ils les auraient sûrement rapportées de préférence aux snip synonymes. D’autres arguments plus complexes vont dans le même sens et, d’ailleurs, selon l’article de Katsnelson [1], plus de cinquante affections ont maintenant été liées à des mutations synonymes. Il semble donc bien que l’on ne puisse vraiment plus éliminer ce type d’altération dans la recherche des bases génétiques d’une maladie. |
Les mécanismes et les conséquences Un autre article publié à l’automne 2011 dans Nature Reviews Genetics [6] passe en revue cette question en s’intéressant plus particulièrement aux mécanismes par lesquels les mutations « silencieuses » exercent leur effet. C’est d’ailleurs de cette revue qu’est extraite l’affirmation précédemment citée : les cinquante affections en cause sont répertoriées dans un tableau du supplément accessible via Internet. Je présente ci-dessous (Tableau II) les quinze premières, avec quelques indications très résumées.
Table II.
| Affection |
Gène |
Mécanisme |
| Dégénérescence maculaire |
CFHR5
|
Épissage |
| Amyotrophie spinale infantile liée à l’X |
UBE1
|
Méthylation exon 15 |
| Myasthénie |
CHRNE
|
Épissage |
| Asthme |
SLC6A7
|
Épissage |
| Sarcoïdose pulmonaire |
CARD15
|
? |
| Tuberculose |
TIRAP
|
? |
| Mucoviscidose |
CFTR
|
Repliement de l’ARNm |
| Hyperlipidémie de type III |
LPL
|
? |
| Déficience en prékallicréine |
PK
|
Épissage ? |
| Hémophilie B |
F9
|
? |
| Syndrome de Crouzon |
FGFR2
|
Épissage |
| Ostéoporose |
CD44
|
Enhancer de l’épissage |
| Syndrome de Treacher-Collins |
TCOF1
|
Épissage |
| Cancer du fumeur |
NBS1
|
? |
| Cancer colorectal |
ERCC1
|
? |
Quinze affections pour lesquelles une mutation « silencieuse » peut provoquer la maladie ou modifier ses caractéristiques. Le gène touché est indiqué dans la deuxième colonne, et le type de mécanisme invoqué (ou parfois prouvé) est donné dans la troisième colonne (tableau établi à partir des données du tableau 1 du supplément de [ 6]). |
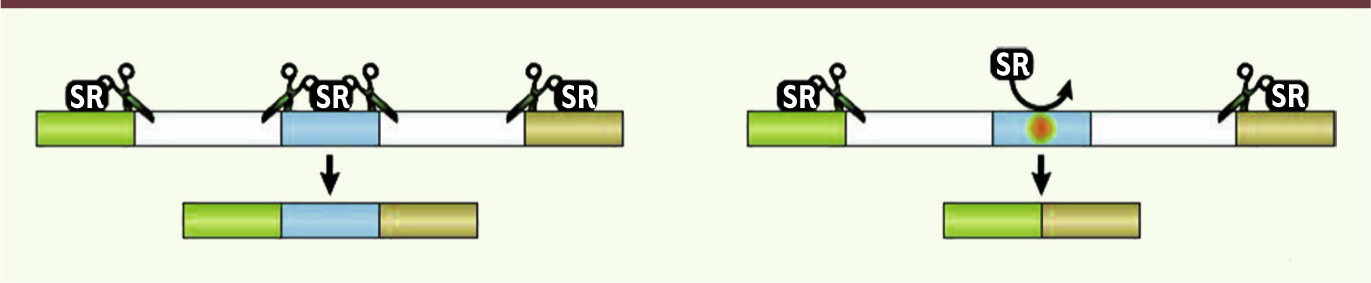
Rappelons qu’il s’agit à chaque fois d’un snip situé dans un exon dont les deux versions aboutissent au codage du même acide aminé, et que la liaison entre ce polymorphisme et l’affection est solidement établie. En revanche, la démonstration du mécanisme reste souvent incomplète, plus proche de l’hypothèse que de la conclusion. On voit en tout cas qu’un défaut d’épissage est évoqué dans la majorité des cas : précisons bien qu’il ne s’agit pas de la modification d’un site d’épissage proprement dit, mais d’une altération à l’intérieur d’un exon qui modifie le site de reconnaissance d’une protéine de régulation de l’épissage et, du coup, provoque un saut d’exon (Figure 1).
 | Figure 1.Effet d’une mutation « silencieuse » sur l’épissage d’un pré-ARNm. À gauche, la protéine de régulation (SR : splicing regulator) reconnaît son site dans l’exon central et dirige correctement l’épissage. À droite, la mutation synonyme détruit le site de reconnaissance et l’épissage est incorrect (schéma inspiré d’une partie de la figure 1 de [ 6]). |
Bien d’autres mécanismes sont possibles et ont été démontrés dans certains cas : altération de la stabilité de l’ARN messager, qui devient instable (vite dégradé) ou au contraire trop stable, avec une structure secondaire qui freine sa traduction ; modification de la vitesse de déplacement des ribosomes, qui peut influer sur le repliement de la chaîne protéique. |
La morale de l’histoire…. … c’est notamment que l’interprétation faite jusqu’ici des associations génétiques à grande échelle (GWAS) devrait être revue en évitant d’écarter a priori les snip synonymes sous prétexte qu’ils correspondent à des mutations silencieuses. Les données de Chen et al. [4] montrent bien que ce n’est pas le cas. Et l’optimisation des protéines thérapeutiques ou, plus encore, des constructions destinées à la thérapie génique - grâce à l’introduction de codons synonymes censés augmenter l’expression de la protéine - devrait être envisagée avec une très grande prudence. Décidément, « Rien n’est simple, tout se complique » [7]. |
L’auteur déclare n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Footnotes |
1. Katsnelson A. Breaking the silence . Nat Med. 2011; ; 17 : :1536.–1538. 2. Kimchi-Sarfaty C, Oh JM, Kim IW, et al. A « silent » polymorphism in the MDR1 gene changes substrate specificity . Science. 2007; ; 315 : :525.–528. 3. Nackley AG, Shabalina SA, Tchivileva IE, et al. Human catechol-O-methyltransferase haplotypes modulate protein expression by altering mRNA secondary structure . Science. 2006; ; 314 : :1930.–1933. 4. Chen R, Davydov EV, Sirota M, Butte AJ. Non-synonymous and synonymous coding SNPs show similar likelihood and effect size of human disease association . PLoS One. 2010; ; 5 : :e13574.. 5. International HapMap Consortium. , Frazer KA, Ballinger DG, Cox DR, et al. A second generation human haplotype map of over 3.1 million SNPs . Nature. 2007; ; 449 : :851.–861. 6. Sauna ZE, Kimchi-Sarfaty C. Understanding the contribution of synonymous mutations to human disease . Nat Rev Genet. 2011; ; 12 : :683.–691. 7. Sempé JJ. Rien n’est simple . Paris: : Denoël; 1962. |