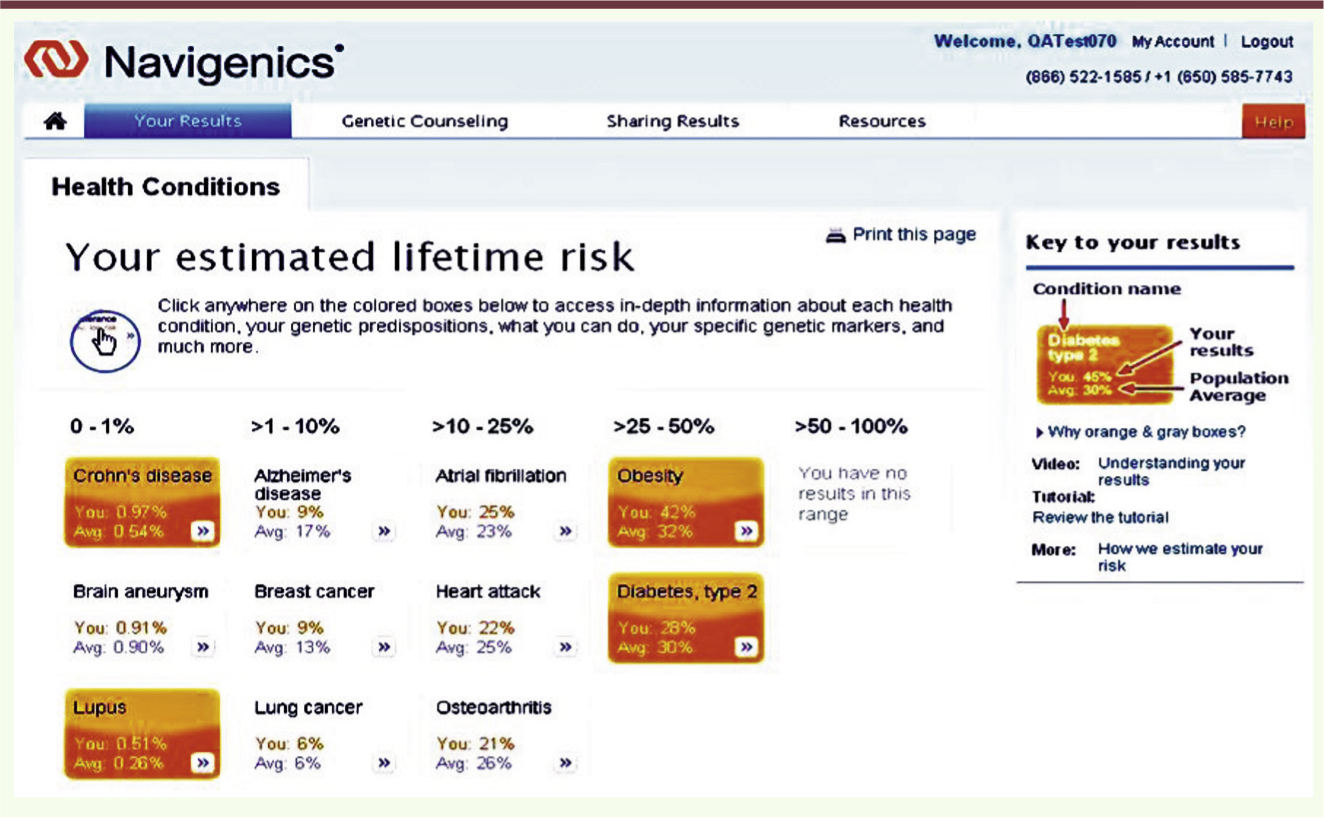
Parmi les 23 affections pour lesquelles Navigenics donne une évaluation du risque, 16 font partie des 51 retenues pour le questionnaire médical. C’est donc sur ces 16 maladies que va porter l’étude comparative. Dans un premier temps, les auteurs ont recherché sur cet échantillon si les résultats du Navigenics Health Compass étaient corrélés avec l’histoire médicale familiale ou personnelle des participants. Vu la taille de l’échantillon (plus de 3 000 personnes), on peut valablement faire une telle étude. Si elle n’a aucun sens au niveau individuel, comme je l’avais relevé précédemment [1] (ce n’est pas parce qu’on a un risque élevé pour une affection que l’on en sera forcément atteint), elle devient pertinente si le nombre de cas est suffisant, comme pour le diabète de type 2 : la prévalence publiée est de 6,7 % et, de fait, dans l’échantillon, près de la moitié des participants ont au moins un membre atteint dans leur famille2 et près de 3 % (soit une centaine de personnes) souffrent effectivement de cette maladie. Parmi les 16 affections étudiées et présentées dans l’ordre décroissant d’héritabilité sur le Tableau I, toutes permettent ainsi une étude sur l’association avec l’histoire familiale, et la moitié environ sont suffisamment fréquentes pour autoriser l’étude de la relation avec l’histoire médicale personnelle. De fait, une corrélation très significative entre les indications de Navigenics et l’histoire médicale familiale est trouvée pour cinq affections (Tableau I) et, pour trois d’entre elles (maladie de Graves, diabète de type 2 et syndrome des jambes sans repos), avec l’histoire médicale personnelle. Ces cas de corrélation positive correspondent tous à des affections relativement fréquentes (donc statistiquement évaluables au sein de l’échantillon) et présentant une forte héritabilité (de 0,6 à 0,7). Au total, concluent les auteurs de l’étude, le Navigenics Health Compass donne bien - pour les maladies fortement héritables et relativement fréquentes - des résultats cohérents avec les données de l’histoire médicale du patient et de sa famille. Cela apparaît donc comme une validation partielle du test commercialisé par cette entreprise : il « capture » effectivement une fraction suffisante des déterminants génétiques de l’héritabilité pour fournir une évaluation du risque qui est cohérente (ou du moins corrélée) avec l’histoire familiale et, dans quelques cas, avec l’histoire personnelle des patients. Ce résultat positif n’était pas évident a priori : comme l’ensemble des snip (single nucleotide polymorphism) connus ne rend compte que d’une faible part de l’héritabilité d’une affection [6], la corrélation aurait fort bien pu être inexistante.
Tableau I.
| Affection |
Héritabilité |
Prévalence |
Prévalence familiale
|
Prévalence personnelle
|
| Valeur |
Corrélation v. ELTR (P) |
Valeur |
Corrélation v. ELTP (P) |
| Maladie de Crohn |
0,80 |
0,0020 |
0,0252 |
0,537 |
0,00326 |
nsa |
| Anévrisme aortique |
0,72 |
0,0265 |
0,0643 |
0,159 |
0,00148 |
ns |
| Maladie de Graves |
0,64 |
0,011515 |
0,220 |
< 0,001 |
0,0797 |
< 0,001 |
| Diabète de type 2 |
0,64 |
0,0665 |
0,477 |
< 0,001 |
0,0294 |
0,007 |
| Lupus |
0,62 |
0,00045 |
0,0300 |
0,027 |
0,00416 |
ns |
| Maladie d’Alzheimer |
0,62 |
0,0150 |
0,233 |
< 0,001 |
0,000294 |
ns |
| Syndrome des jambes sans repos |
0,60 |
0,0270 |
0,0794 |
< 0,001 |
0,0507 |
0,058 |
| Crise cardiaque (hommes) |
0,57 |
0,055 |
0,568 |
0,249 |
0,0112 |
0,322 |
| Sclérose en plaques |
0,48 |
0,000583 |
0,0314 |
0,715 |
0,00416 |
ns |
| Cancer de la prostate (hommes) |
0,42 |
0,0150 |
0,170 |
0,107 |
0,0132 |
0,666 |
| Crise cardiaque (femmes) |
0,38 |
0,029 |
0,576 |
0,966 |
0,00166 |
ns |
| Cancer du côlon |
0,35 |
0,0027 |
0,191 |
0,765 |
0,00294 |
ns |
| Cancer du sein (femmes) |
0,27 |
0,0089 |
0,308 |
0,878 |
0,0283 |
0,553 |
| Mélanome |
0,21 |
0,001762 |
0,132 |
0,585 |
0,0165 |
0,476 |
| Cancer du poumon |
0,14 |
0,000884 |
0,227 |
0,484 |
0,00118 |
ns |
| Glaucome |
0,13 |
0,0180 |
0,201 |
0,183 |
0,00884 |
ns |
Corrélation entre les indications du Navigenics Health Compass, l’histoire médicale familiale et l’histoire médicale personnelle. Pour chaque affection sont données les valeurs de l’héritabilité (colonne 2) et de la prévalence dans la population générale (colonne 3). Les deux colonnes suivantes indiquent la prévalence familiale, comptée à partir du moment où un membre au moins de la famille (au sens de la note 2) est atteint – la valeur est donc nettement plus forte que la précédente, et le degré de corrélation (P- value) avec le risque évalué par Navigenics (ELTR, estimated lifetime risk, voir Figure 1) pour chaque personne. Les deux dernières colonnes montrent la prévalence personnelle (proportion des volontaires de l’étude présentant l’affection en cause) et son degré de corrélation avec l’ELTR. On voit que le test Navigenics est corrélé avec l’histoire familiale pour cinq affections, et « prédit » l’histoire personnelle pour trois d’entre elles (P inférieur ou égal à 0,05). |
|
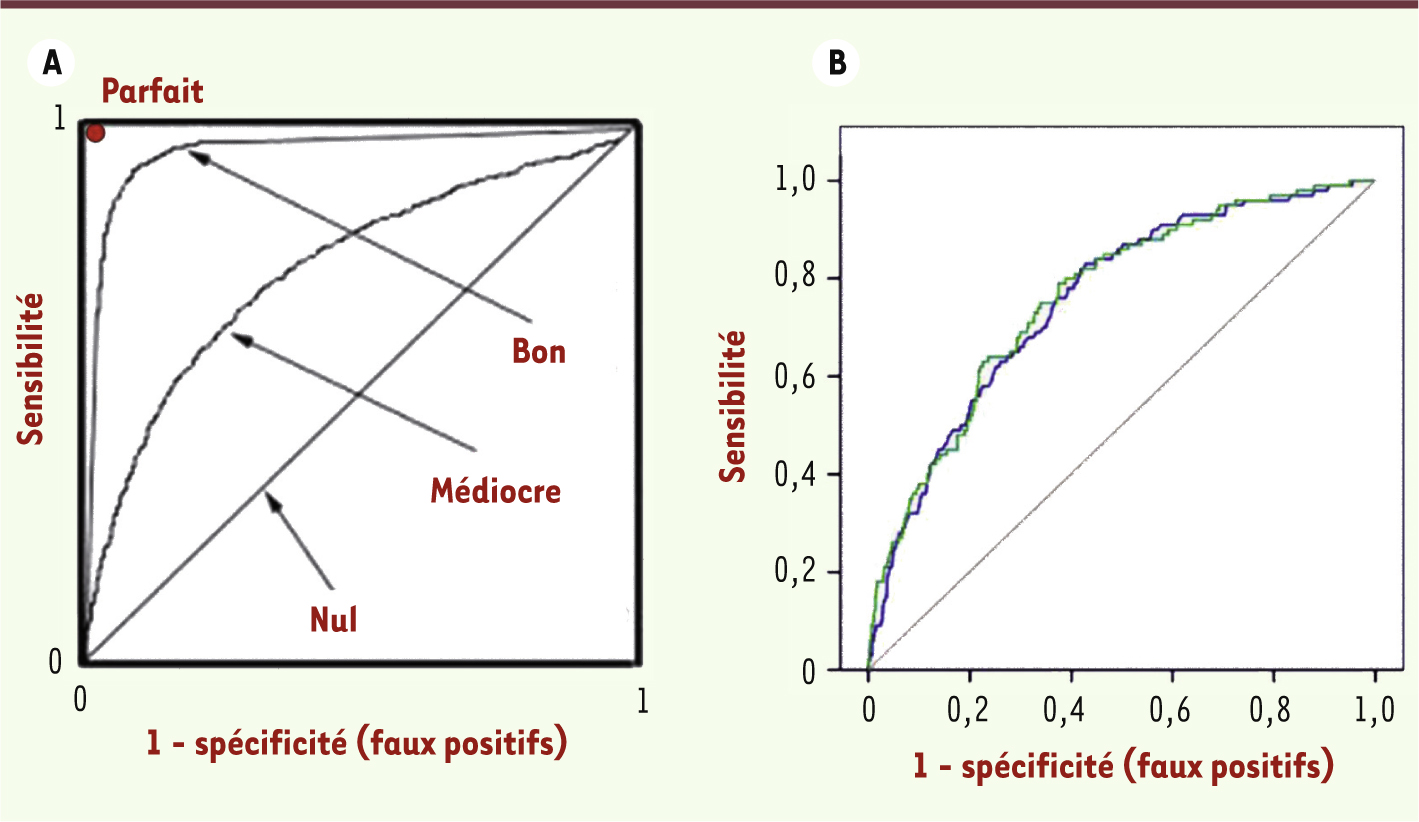
Mais qu’en est-il de la valeur ajoutée ? Mais l’interrogation réellement pertinente est la suivante : le test Navigenics procure-t-il une information additionnelle par rapport à l’histoire familiale ? Cette dernière est après tout très informative [7], facile à obtenir, son coût est très faible sinon nul, et le test génétique ne se justifie réellement que s’il apporte des résultats plus complets et plus précis. Ce point peut être examiné en étudiant la valeur prédictive de l’histoire médicale familiale pour la santé du patient (repérée par son histoire médicale personnelle), et en voyant si l’ajout des résultats du test Navigenics améliore ou non cette prédiction. Ce point est examiné [4] en utilisant le formalisme des courbes ROC (receiver operating characteristic), très courant dans l’industrie du diagnostic mais peut-être moins connu des lecteurs de médecine/sciences - je vais donc en donner le principe [8]. Il s’agit d’un diagramme mis au point à l’origine dans le secteur des télétransmissions (d’où son nom) qui permet de représenter les performances d’un test en termes de sensibilité et de spécificité. Ces deux variables ne sont pas indépendantes : si l’on ajuste les paramètres d’un test (par exemple, la valeur du signal à partir duquel on considère que le résultat est positif) afin d’augmenter la sensibilité, on va du même coup générer plus de faux positifs et donc diminuer la spécificité. Dans un test de pur dépistage (sérologie VIH par exemple), on privilégiera la sensibilité, quitte à avoir quelques faux positifs qui seront éliminés par le test de confirmation plus spécifique (et plus coûteux) que l’on fera passer à tous les positifs en première intention. Dans d’autres cas au contraire, on privilégiera la spécificité. Les performances d’un test peuvent être représentées sur un graphique, appelé « courbe ROC » (Figure 2A), dans lequel un test parfait (parfaitement sensible et parfaitement spécifique) serait représenté par un point (en haut à gauche sur le graphique), un très bon test par une courbe montant très vite jusqu’au maximum, et un test sans valeur (pas différent d’un tirage au hasard) par une diagonale. On peut attribuer une valeur numérique à la qualité d’un diagnostic en calculant la surface comprise sous la courbe (AUC, area under the curve) : pour un test presque parfait, elle est proche de 1, pour un test sans aucun intérêt elle est de 0,5. On considère en général qu’un test clinique doit avoir une AUC au moins égale à 0,8 pour être utilisable.
 | Figure 2.Courbes ROC. A. Principe de la courbe ROC. La diagonale correspond à un test sans valeur (résultats équivalents à un tirage au sort), le point en haut à gauche à un test parfait (aucun faux positif ni faux négatif). B. Courbe ROC pour le risque personnel de diabète (type 2) d’après l’histoire médicale familiale seule (en bleu) ou avec ajout, en plus, des résultats du test de Navigenics (en vert). On voit que le test n’ajoute pas d’information puisque les deux courbes sont pratiquement confondues. (Figure établie d’après des données contenues dans le supplément de [ 4]). |
Les auteurs ont alors étudié les performances d’un test prédisant l’histoire médicale personnelle à partir de l’histoire médicale familiale du même patient, et ont déterminé ses performances sur l’échantillon du projet SGHI. Selon les affections choisies, la valeur d’AUC obtenue oscille entre 0,55 et 0,68, des valeurs modestes mais significatives, confirmant ainsi que l’on peut dans une certaine mesure évaluer le risque d’une personne pour une affection à partir de son histoire médicale familiale. La question est alors : si l’on ajoute aux données familiales les résultats de l’analyse Navigenics, améliore-t-on la qualité de la prévision ? Comme on le voit sur la Figure 2B, la réponse est non : la courbe « sans Navigenics » et celle « avec » sont pratiquement confondues, l’analyse génétique telle qu’elle est pratiquée (et vendue) par cette entreprise n’ajoute donc rien aux informations familiales. La citation rappelée au début de cette chronique se trouve ainsi validée expérimentalement : une bonne histoire familiale vaut bien mieux que les analyses « génome entier » telles qu’elles sont pratiquées actuellement. Le seul cas de figure où le Navigenics Health Compass pourrait présenter un intérêt, selon les auteurs, est celui d’une personne adoptée, puisqu’en principe elle n’a pas accès aux données de sa famille biologique. Convenons que cela restreint considérablement le public concerné. |