| |
| Med Sci (Paris). 2012 March; 28(3): 258–261. Published online 2012 April 6. doi: 10.1051/medsci/2012283010.MITF : une clé génétique du mélanome et du carcinome rénal ? Corine Bertolotto,1,2* Fabienne Lesueur,3,5 and Brigitte Bressac de Paillerets3,4 1Inserm, U895, équipe labélisée Ligue contre le cancer, C3M, 06204Nice, France 2Université Sophia-Antipolis de Nice, UFR médecine, 06204Nice, France 3Service de génétique, Institut de cancérologie Gustave Roussy, 94805Villejuif, France 4Inserm, U946, Variabilité génétique et maladies humaines, 75010Paris, France 5Addresse actuelle : Genetic cancer susceptibility group, international agency for research on cancer, 69372Lyon, France MeSH keywords: Allèles, Facteurs de transcription à motifs basiques hélice-boucle-hélice et à glissière à leucines, génétique, physiologie, Néphrocarcinome, épidémiologie, Différenciation cellulaire, Transformation cellulaire néoplasique, France, Régulation de l'expression des gènes tumoraux, Prédisposition génétique à une maladie, Humains, Tumeurs du rein, Mélanocytes, métabolisme, Mélanome, Facteur de transcription associé à la microphtalmie, Modèles génétiques, Mutation faux-sens, Tumeurs primitives multiples, Syndromes néoplasiques héréditaires, Stress oxydatif, Tumeurs cutanées |
La transformation tumorale est la conséquence d’altérations génétiques et/ou épigénétiques de gènes clés (gènes drivers) contrôlant des processus tels que la prolifération cellulaire, l’apoptose, la sénescence et la réponse aux dommages de l’ADN. Ces altérations vont conduire à la synthèse de protéines activées et modifiées (pour les oncogènes) ou ayant une activité réduite, voire nulle (pour les gènes suppresseurs de tumeurs ou les gènes impliqués dans la réparation de l’ADN). Les modifications de la stœchiométrie et du comportement biologique normal de ces protéines au sein des cellules constituent une étape d’accélération de la progression tumorale. À l’étape de l’initiation tumorale, l’altération génétique peut être constitutionnelle. Quant on sait que le pronostic vital d’un patient est étroitement lié à la précocité de sa prise en charge, l’identification des gènes de prédisposition aux cancers apparaît d’une importance capitale pour identifier et suivre les patients à risque. |
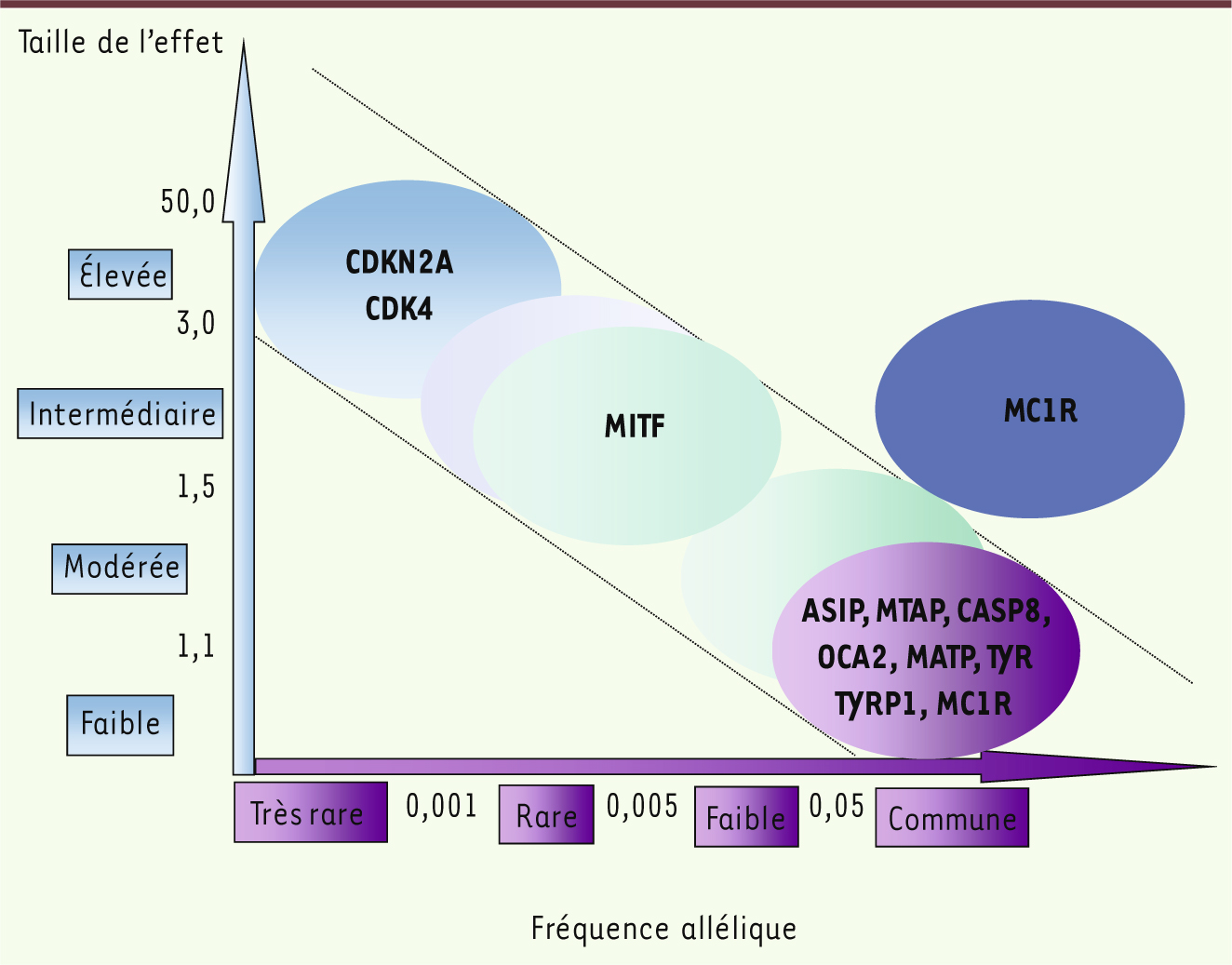
Identification d’une mutation dans le gène MITF prédisposant au mélanome et au cancer du rein Facteurs de risque de développer un mélanome L’incidence du mélanome augmente régulièrement en France. En 2011, ce cancer était classé au 9 e rang par sa fréquence 1. Paradoxalement, la mortalité reste stable, probablement en raison de l’efficacité des mesures de prévention et de dépistage précoce. Ces mesures sont d’autant plus importantes qu’en cas de diagnostic tardif, lorsque le cancer est au stade métastatique, il n’existe pas ou peu de traitements efficaces. Les principaux facteurs de risque du mélanome incluent : un nombre élevé de nævi, l’exposition solaire et la réaction de la peau selon le phototype (couleur de la peau) et des mutations constitutionnelles ou germinales des gènes CDKN2A (cyclin-dependent kinase inhibitor 2A) et CDK4 (cyclin dependent kinase 4). Dans le contexte de familles dont plusieurs sujets sont atteints de mélanome cutané et dans lesquelles une mutation germinale de CDKN2A a été identifiée, les porteurs de la mutation familiale ont un risque d’environ 60 % de développer un mélanome au cours de leur vie [1]. Outre les mutations rares dans les gènes de prédisposition CDKN2A et CDK4 (dits gènes à effet fort ou gènes à haut risque), des polymorphismes nucléotidiques ou SNP (single-nucleotide polymorphisms) fréquents (notamment dans les gènes MC1R [melanocortin receptor 1], ASIP [agouti signaling protein], MTAP [methylthioadenosine phosphorylase], MATP [membrane-associated transporter protein] et CASP8 [caspase 8]) ont également été associés à un risque modéré à faible de développer la tumeur. De plus, certains variants alléliques de MC1R modifient la pénétrance des gènes à haut risque [2]. Identification d’une mutation germinale faux sens dans le gène MITF Récemment, nous avons identifié par séquençage une mutation germinale fauxsens dans le gène MITF (microphthalmia-associated transcription factor) qui est associée à un risque accru de mélanome chez les porteurs [ 3, 4]. MITF code pour un facteur de transcription de la superfamille MYC, de type hélice-boucle-hélice, qui comporte des répétitions de leucines. Il joue un rôle clé dans l’homéostasie mélanocytaire et a été impliqué récemment dans la pathogénie du mélanome. MITF régule la transcription de gènes impliqués dans le contrôle du cycle cellulaire, de la prolifération et de la migration cellulaires. En fonction de son niveau et de ses modifications post-traductionnelles, MITF agirait comme un rhéostat induisant différentes fonctions biologiques [ 5]. Une amplification génique et des mutations somatiques de MITF ont été rapportées dans des échantillons tumoraux de mélanome [ 6, 7]. Pour la première fois, nous démontrons la responsabilité de MITF dans la susceptibilité au développement d’un mélanome. En effet, nous avons identifié une mutation germinale p.E318K qui, en changeant l’acide glutamique en position 318 en lysine, altère un domaine très conservé de la protéine. Dans les populations étudiées (France, Australie, Angleterre), la mutation est retrouvée chez moins d’une personne sur 165. Cependant, elle est deux (Australie et Angleterre) à cinq (France) fois plus fréquente chez les sujets atteints de mélanome (en particulier les sujets avec une histoire familiale de mélanome ou ayant eu des mélanomes primaires multiples) que chez les témoins. Cette mutation augmente au moins d’un facteur deux (étude australienne sur 3 920 sujets) à cinq (étude française sur 2 262 sujets) le risque de développer un mélanome. De plus, cette mutation ségrège dans certaines familles dans lesquelles surviennent des cas multiples de mélanome, mais pas dans toutes [ 3, 4]. Ces observations indiquent que ce variant de MITF est rarement retrouvé dans la population générale et qu’il appartient à la classe des variants génétiques conférant un risque intermédiaire de développer la maladie (Figure 1).
 | Figure 1.
Les variants génétiques dans le mélanome. Fréquence allélique et importance de l’effet génétique (adapté de [ 14]). |
MITF : le premier facteur génétique identifié commun au mélanome et au cancer du rein Des études épidémiologiques ont montré que les patients atteints de mélanome ont un risque accru de développer d’autres types de tumeurs tel qu’un carcinome à cellules rénales (CCR). Inversement, les individus atteints d’un carcinome à cellules rénales ont un risque significativement plus élevé de développer un mélanome. Les facteurs de risque connus de ce carcinome sont le tabagisme, l’obésité et l’hypertension artérielle. Aucun des facteurs de risque ou des facteurs génétiques connus pour ces deux cancers ne permettait d’expliquer le développement d’un mélanome et d’un cancer du rein chez le même patient [ 8]. Bien que le rôle de MITF dans la physiopathologie rénale reste à déterminer, il a été montré qu’il stimulait la transcription, entre autres, du facteur HIF1A ( hypoxia inducible factor) [ 16], une cible plus ou moins directe de tous les gènes de prédisposition au cancer du rein, comme les gènes suppresseurs de tumeur VHL (von Hippel Lindau), FLCN ( folliculin gene), FH ( fumarate hydratase), SDHB ( succinate dehydrogenase complex subunits B), TSC1 ( tuberous sclerosis complex) et TSC2 et l’oncogène MET (codant pour l’ hepatocyte growth factor receptor). MITF contrôle également directement la transcription de MET [ 9]. Il est donc très intéressant de souligner que la mutation germinale p.E318K de MITF a également été détectée chez une série de patients ayant développé un carcinome à cellules rénales et un mélanome malin cutané, ainsi que dans une série clinique de patients à haut risque de développer un carcinome à cellules rénales mais qui ne présentaient aucune des mutations germinales des gènes connus de prédisposition à ce cancer [ 3]. Une fois encore, la fréquence de la mutation était significativement plus élevée dans ces deux séries de patients que dans la population générale. Notre étude propose donc MITF comme étant le premier facteur génétique commun au mélanome et au cancer du rein. |
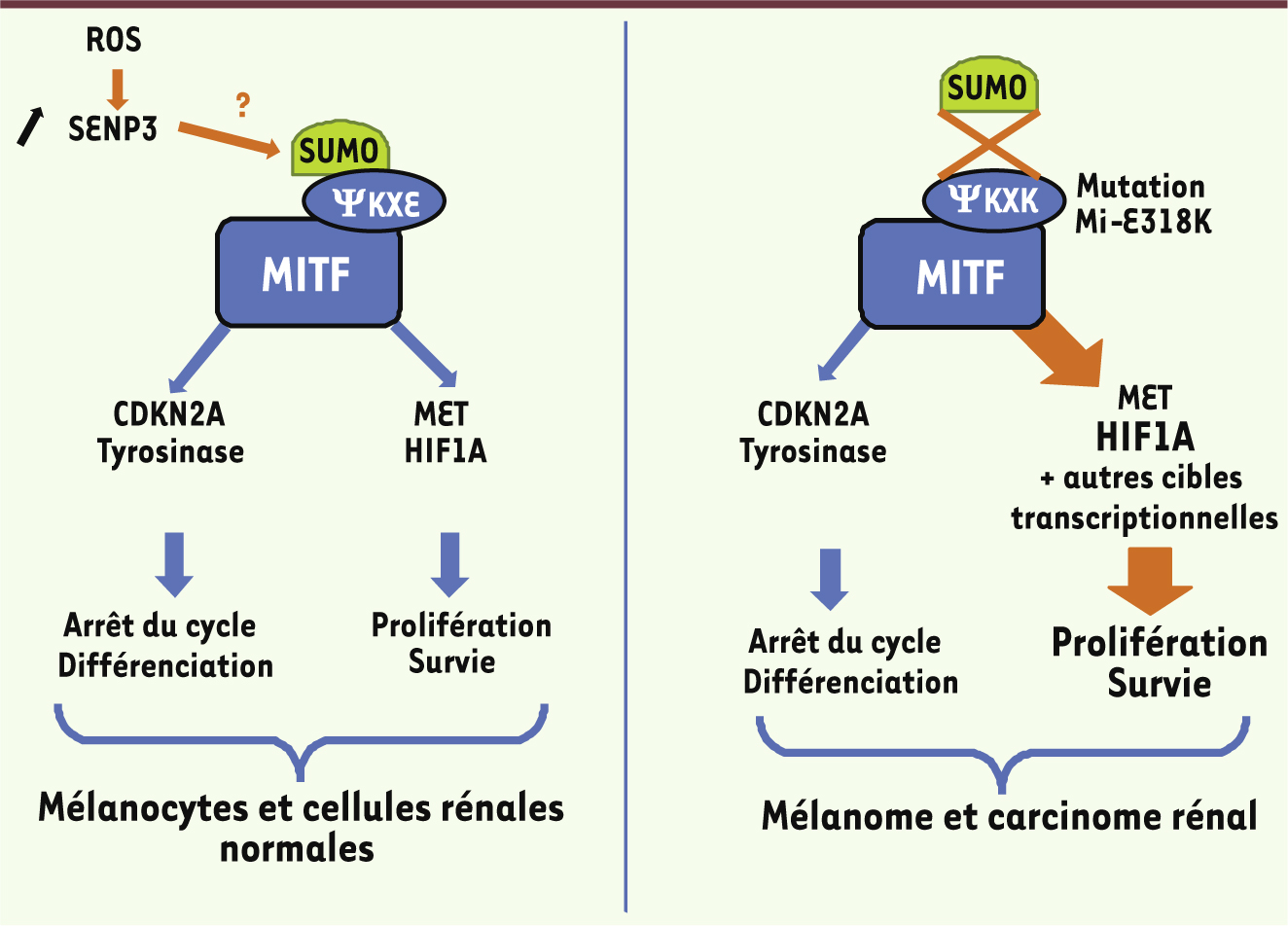
Régulation de la sumoylation et cancer La mutation faux-sens p.E318K de MITF transforme la séquence IKQE en IKQK. Cette séquence correspond à un site consensus de sumoylation (ΨKXE où Ψ représente un acide aminé hydrophobe). La sumoylation est une modification post-traductionnelle aboutissant à la liaison covalente d’une ou plusieurs protéines SUMO sur une lysine acceptrice. Le processus de sumoylation est très proche biochimiquement de l’ubiquitinylation et contrôle la localisation cellulaire des protéines, leur stabilité et leur activité transcriptionnelle. Les protéines SUMO sont essentielles à la fonction des cellules eucaryotes [10]. Une relation entre la dérégulation de la voie de sumoylation et l’apparition de maladies chez l’homme, telles que les maladies neurodégénératives et les maladies cardiaques a été proposée. Ainsi, les protéines impliquées dans les maladies neurologiques telles que les maladies de Huntington (huntingtin), d’Alzheimer (tau, amyloid precursor protein) et de Parkinson (tau, α-synucléine, DJ-1), ainsi que dans les maladies cardiaques (NKX2-5), présentent une altération de leur sumoylation. Des mutations constitutionnelles de la lamine A, dans des cas de cardiomyopathies dilatées, altèrent un site consensus de sumoylation, ce qui entraîne une diminution significative de la liaison de protéines SUMO. Cette réduction de sumoylation s’accompagne d’une modification de la localisation cellulaire de la lamine A, un changement de morphologie nucléaire aboutissant à une mort cellulaire [11]. La sumoylation joue également un rôle dans la cancérogenèse. Le niveau d’expression des enzymes SUMO, UBC9 (une E2 ubiquitin-conjugating enzyme) et PIAS3 (protein inhibitor of activated STAT, une E3 SUMO-protein ligase) est augmenté dans certains cancers et a été associé à de mauvais pronostics cliniques. Une surexpression des protéases SUMO (SENP) est également observée dans les cancers. De plus, plusieurs facteurs tels que p53, pRb, BRCA1 (breast cancer), dont les activités sont dérégulées au cours de la progression tumorale, sont des cibles de la sumoylation [12]. Nos résultats récents apportent un nouvel éclairage sur le rôle essentiel de la sumoylation dans le cancer [3, 4]. En effet, la mutation E318K de MITF abolit un site de conjugaison aux protéines SUMO et altère drastiquement la sumoylation de la protéine. L’analyse de l’occupation du génome par la forme sauvage et mutante de MITF a révélé une régulation d’un ensemble de gènes distincts avec notamment un groupe de régions fortement liées par le mutant. Nos résultats renforcent donc la notion que la sumoylation est impliquée dans la répression génique. Enfin, l’analyse du transcriptome a révélé que la forme hyposumoylée de MITF, qui favorise la migration et l’invasion cellulaires, est associée à des réponses cellulaires de type prolifération, hypoxie et inflammation. Les mécanismes moléculaires par lesquels MITF agit dans la phase d’initiation de la transformation restent à déterminer, mais une des hypothèses serait que la mutation E318K altère la réponse des mélanocytes et des cellules rénales au stress oxydant (Figure 2).
 | Figure 2.
La mutation E318K de MITF altèrerait la réponse au stress oxydant. Les rayonnements ultraviolets et l’hypoxie, qui sont impliqués dans le développement du mélanome et du cancer du rein respectivement, conduisent à la formation d’espèces réactives de l’oxygène (ROS). Les ROS stabilisent la désumoylase SENP3 ( SUMO1/sentrin/SMT3 specific peptidase 3) [ 15] et induisent la transactivation de HIF1A. Dans le système mélanocytaire où MITF régule HIF1A, la mutation E318K de MITF, entraînant son hyposumoylation, mimerait une stabilisation de SENP3 induisant de façon soutenue la transcription de HIF1A. Ainsi, MITF hyposumoylée pourrait empêcher l’adaptation des cellules au stress et induire la transformation cellulaire. |
|
MITF appartient à la famille des facteurs de transcription de type Microphthalmia (MiT) comprenant également TFE3 et TFEB (transcription factor EB), déjà impliqués dans des cancers, en particulier celui du rein [13]. Notre étude révèle donc l’implication d’un troisième membre de la famille MiT dans la tumorigenèse rénale (Tableau I).
Tableau I.
| Cancers →Protéines ↓ |
Mélanomes |
Carcinome à cellules rénales |
Sarcomes |
|
Mutation germinale |
Mutation germinale |
Mutations somatiques (fusion de gènes) |
|
E318K |
E318K |
EWS-ATF1 t(12;22)(q13;q12) |
| MITF |
Mutations somatiques |
|
|
|
E87R, 4TΔ2B, L135V , L142F, A/T (-3),
G244R, D380N |
|
|
|
Amplification génomique 3p14 |
|
|
|
|
|
Mutations somatiques (fusion de gènes) |
Mutations somatiques (fusion de gènes) |
|
|
PRCC-TFE3 : t(X;1)(p11.2;q21) |
ASPL-TFE3: t (X;17)(p11.2;q25) |
| TFE3 |
|
ASPSCR1-TFE3 : t(X;17)(p11.2;q25) |
|
|
|
CLTC-TFE3 : t(X;17)(p11.2;q23) |
|
|
|
NONO-TFE3 : inv(X)(p11;q12) |
|
|
|
PSF-TFE3 : t(X;1)(p11.2;p34) |
|
|
| TFEB |
|
Mutations somatiques (fusion de gènes) |
|
|
|
Alpha : t(6;11)(p21.1;q12) |
|
Altérations des facteurs de transcription de la famille MiT (MITF, TFE3, TFEB) dans les cancers chez l’homme. L’activité de MITF est dérégulée par mutations, amplification et fusion de gènes. Des mutations somatiques dans différents domaines de MITF (7 % des mélanomes primaires et 16 % des mélanomes métastatiques) ont été détectées. Certaines de ces mutations modifient l’activité transcriptionnelle du facteur MITF. Des mutations germinales ont été identifiées dans environ 3 % des cas de mélanomes et/ou de carcinomes à cellules rénales (CCR) et 8 % des cas de mélanomes plus CCR. Une amplification génomique de MITF a été identifiée dans 10 à 20 % des cas de mélanome. La fusion entre EWS (Ewing sarcoma breakpoint region 1) et ATF1 (activating transcription factor 1) conduit à une stimulation de l’expression de MITF. L’activité de TFE3 est dérégulée par fusion de gènes et celle de TFEB par échange de promoteur. |
Les données génétiques et fonctionnelles démontrent que la substitution faux sens E318K de MITF, présente à l’état constitutionnel chez des patients ayant développé un CCR et/ou un mélanome, confère un pouvoir oncogénique au facteur de transcription. Une telle co-occurence pourrait représenter un nouveau syndrome tumoral. La mutation E318K de MITF pourrait être un événement initiateur de la transformation du mélanocyte et de la cellule rénale. L’identification de la mutation de MITF chez les sujets à risque pourrait permettre aux individus de bénéficier d’une surveillance accrue, ciblée sur la détection précoce de lésions potentiellement dangereuses. Cette découverte ouvre aussi des perspectives de chimioprévention, notamment par l’utilisation d’antioxydants. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Les auteurs remercient Jacob Seeler (Inserm U579, Institut Pasteur, Paris) pour la relecture critique de ce manuscrit.
|
Footnotes |
1. Bressac de Paillerets B, Avril MF, Chompret A, Demenais F. Genetic and environmental factors in cutaneous malignant melanoma . Biochimie. 2002; ; 84 : :67.–74. 2. Demenais F, Mohamdi H, Chaudru V, et al. Association of MC1R variants and host phenotypes with melanoma risk in CDKN2A mutation carriers: a GenoMEL study . J Natl Cancer Inst. 2010; ; 102 : :1568.–1583. 3. Bertolotto C, Lesueur F, Giuliano S, et al. A SUMOylation-defective MITF germline mutation predisposes to melanoma and renal carcinoma . Nature. 2011; ; 480 : :94.–98. 4. Yokoyama S, Woods SL, Boyle GM, et al. A novel recurrent mutation in MITF predisposes to familial and sporadic melanoma . Nature. 2011; ; 480 : :99.–103. 5. Carreira S, Goodall J, Denat L, et al. Mitf regulation of Dia1 controls melanoma proliferation and invasiveness . Genes Dev. 2006; ; 20 : :3426.–3439. 6. Cronin JC, Wunderlich J, Loftus SK, et al. Frequent mutations in the MITF pathway in melanoma . Pigment Cell Melanoma Res. 2009; ; 22 : :435.–444. 7. Garraway LA, Widlund HR, Rubin MA, et al. Integrative genomic analyses identify MITF as a lineage survival oncogene amplified in malignant melanoma . Nature. 2005; ; 436 : :117.–122. 8. Maubec E, Chaudru V, Mohamdi H, et al. Characteristics of the coexistence of melanoma and renal cell carcinoma . Cancer. 2010; ; 116 : :5716.–5724. 9. Cheli Y, Ohanna M, Ballotti R, Bertolotto C. Fifteen-year quest for microphthalmia-associated transcription factor target genes . Pigment Cell Melanoma Res. 2010; ; 23 : :27.–40. 10. Sarge KD, Park-Sarge OK. SUMO and its role in human diseases . Int Rev Cell Mol Biol. 2011; ; 288 : :167.–183. 11. Zhang YQ, Sarge KD. Sumoylation regulates lamin A function and is lost in lamin A mutants associated with familial cardiomyopathies . J Cell Biol. 2008; ; 182 : :35.–39. 12. Bettermann K, Benesch M, Weis S, Haybaeck J. SUMOylation in carcinogenesis . Cancer Lett. 2012; ; 316 : :113.–125. 13. Haq R, Fisher DE. Biology and clinical relevance of the micropthalmia family of transcription factors in human cancer . J Clin Oncol. 2011; ; 29 : :3474.–3482. 14. Manolio TA, Collins FS, Cox NJ, et al. Finding the missing heritability of complex diseases . Nature. 2009; ; 461 : :747.–753. 15. Huang C, Han Y, Wang Y, et al. SENP3 is responsible for HIF-1 transactivation under mild oxidative stress via p300 de-SUMOylation . EMBO J. 2009; ; 28 : :2748.–2762. 16. Buscà R, Berra E, Pouysségur J, Ballotti R. HIF1α est une nouvelle cible du facteur de transcription MITF : implication de la cascade AMPc-MITF-HIF1α dans le développement des mélanomes . Med Sci (Paris). 2006; ; 22 : :10.–13. |