| |
| Med Sci (Paris). 2012 February; 28(2): 136–138. Published online 2012 February 27. doi: 10.1051/medsci/2012282006.RIG-I Un commutateur moléculaire détecteur d’ARN viral Eva Kowalinski,1 Jade Louber,2 Denis Gerlier,2* and Stephen Cusack1 1Antenne de Grenoble de l’European Molecular Biology Laboratory, biologie structurale des interactions entre virus et cellule hôte, UJF-EMBL-CNRS, UMI 3265, 6, rue Jules Horowitz, BP181, 38042Grenoble Cedex 9, France 2Virologie humaine, Inserm U758, École normale supérieure de Lyon, université Lyon 1, 21, avenue Tony Garnier, 69007Lyon, France MeSH keywords: Gènes switch, physiologie, Humains, Modèles biologiques, Modèles moléculaires, Liaison aux protéines, Motifs et domaines d'intéraction protéique, RNA helicases, composition chimique, métabolisme, ARN viral, agonistes, Récepteurs à l'acide rétinoïque, génétique |
Les récepteurs spécifiques de l’immunité innée Afin de lutter contre les infections virales, les eucaryotes supérieurs ont développé de multiples mécanismes de défense regroupés en deux types de réponse immunitaire, l’innée et l’adaptative. La première est immédiate mais aspécifique ; la seconde est différée mais très spécifique. Ces deux réponses sont complémentaires et permettent une prise en charge efficace de la plupart des infections virales avec guérison à la clef. La réponse innée est déclenchée par l’activation de récepteurs spécifiques, les PRR (pattern recognition receptors), capables de détecter des motifs moléculaires associés aux microbes, les MAMP (microbe-associated molecular patterns). Il existe trois types de PRR : les récepteurs membranaires de type Toll (Toll-like receptors, TLR), dont la découverte a conduit à l’attribution du Prix Nobel de physiologie et de médecine en 2011 [12], les récepteurs de type Nod (Nod-like receptors, NLR), et les récepteurs cytoplasmiques de type RIG-I (RIG-I-like receptors) ou RLR. Les RLR reconnaissent des ARN viraux issus d’une infection par des virus le plus souvent à génome ARN comme ceux de la grippe, la rougeole, la rage, la poliomyélite, les virus Ebola, Marburg et Nipah, etc. Cela conduit à la sécrétion de cytokines pro-inflammatoires et à l’activation de l’interféron (IFN), le messager soluble. L’IFN alerte les cellules environnantes pour qu’elles activent en interne un ensemble d’effecteurs antiviraux et se protègent de l’infection [1]. Ces récepteurs sont indispensables au contrôle très précoce de l’infection, gage d’une bonne chance de guérison [2]. La famille des RLR comprend trois hélicases à ARN de la superfamille des hélicases 2 (SF2) : RIG-I (retinoic acid-inducible gene I), MDA5 (melanoma differentiation-associated gene 5) et LGP2 (laboratory of genetics and physiology 2). La structure de RIG-I (et de MDA5) comprend deux domaines CARD (caspase activation and recruitment domains) à l’extrémité amino-terminale, un domaine hélicase central comportant plusieurs motifs conservés, et un domaine carboxy-terminal (CTD). RIG-I reconnaît des ARN dont la longueur est comprise entre 0,3 et 1kb associant deux motifs : une structure double brin (ARNdb) et une extrémité 5’ triphosphate (5’ppp). Ces deux motifs permettent à RIG-I de discriminer les ARN viraux des ARN cellulaires qui n’arborent jamais d’extrémité 5’ppp libre dans le cytoplasme [2, 3]. |
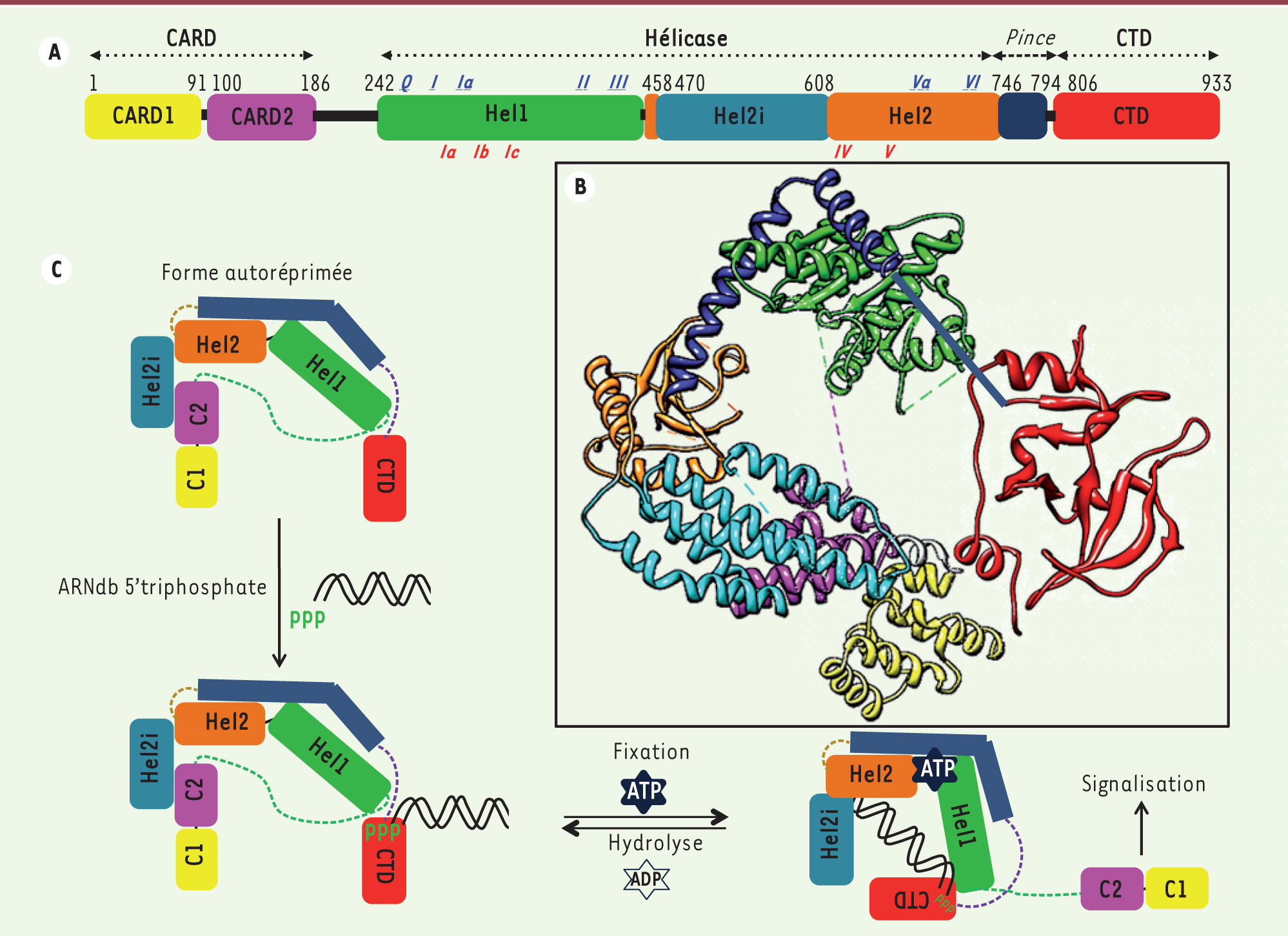
Structure du domaine hélicase de RIG-I et interaction avec les ARN agonistes Les structures tridimensionnelles de la protéine RIG-I entière de canard [4] et de formes tronquées comportant les domaines CARD-hélicase, hélicase, et hélicase-CTD, complexées ou non avec de l’ARNdb, viennent d’être simultanément résolues à l’automne 2011 par quatre équipes, dont la nôtre, travaillant sur le RIG-I de canard, humain ou de la souris [4–7]. Les informations obtenues sont complémentaires. Elles convergent vers un modèle cohérent des changements de conformation responsables de la commutation d’un état autoréprimé inactif (ou dormant) vers un état activé de RIG-I lors de la détection et fixation de son ligand ARN ou agoniste. Le domaine hélicase comporte trois sous-domaines structuraux, deux domaines de type hélicase RecA (Hel1 et Hel2, chacun composé d’un feuillet β et de plusieurs hélices α) et un troisième domaine atypique, constitué de cinq hélices α, inséré au tout début de Hel2 et appelé Hel2i. En absence de ligand, cette structure est ouverte et très flexible avec le domaine Hel2 peu ordonné. Entre Hel2 et le CTD, un domaine hélicoïdal, composé de 2 longues hélices α coudées à 65° - 80° en forme de V, réalise un pontage mécanique et pince ensemble les domaines Hel2 et Hel1 [4–7]. La première hélice α remonte vers Hel1 qui est prise en sandwich par la seconde hélice α. Cette pince moléculaire établit donc une connexion mécanique entre Hel1, Hel2, et le CTD, et son intégrité est nécessaire à la transmission du signal à la manière d’un arbre à came [5]. Au sein de la protéine RIG-I en mode autoréprimé (sans ARNdb), le lien Hel2-CTD est flexible et le CTD est « libre », car la structure de CTD n’est pas « vue » pour cause de mobilité dans le cristal de RIG-I examiné par diffraction aux rayons X [4]. La comparaison de la structure de RIG-I avec des domaines CARD mais sans ligand et de la structure de RIG-I complexée à une molécule d’ARNdb et à un analogue non hydrolysable de l’ATP révèle un changement de conformation majeur (Figure 1). Les domaines Hel1 et Hel2 pivotent par rotation croisée convergente de plusieurs dizaines de degrés l’un vers l’autre avec structuration complémentaire de Hel2. Cela conduit à la formation simultanée de deux sites « matures » pour la liaison coopérative de l’ARNdb, d’une part, et de l’ATP, d’autre part, selon une disposition proche, mais distincte, de celle adoptée par d’autres hélicases via des motifs conservés chez la plupart d’entre elles. À l’intérieur, les motifs Ia, Ib et Ic de Hel1 se lient à l’extrémité 3’ du brin 3’ et le motif IIa interagit avec le brin 5’ de l’ARN. À l’extérieur, les motifs Q, I, Ia, II (Walker B), Va et VI lient l’ATP. En outre, Hel2i interagit aussi avec les brins 5’ et 3’ de l’ARN via, en particulier, une liaison hydrogène entre la glutamine 511 et le brin 3’, dont la rupture par mutation affaiblit l’activité de RIG-I [5]. La conservation de ce résidu parmi les RLR suggère son implication dans la reconnaissance des ARN par les RLR en général. Ce mode de liaison du domaine hélicase sur une longueur de neuf nucléotides est parfaitement complémentaire avec le mode de liaison du CTD à l’ARNdb 5’ppp précédemment caractérisé [8] ; les domaines Hel1, Hel2, Hel2i et CTD enferment ainsi l’ARNdb dans un canal presque circulaire dont la stabilité est assurée par la liaison de l’ATP qui vient, tel une clef de voûte amovible, bloquer par l’extérieur l’orientation relative des structures secondaires d’Hel2 [5]. L’activité ATPase du site assure la réversibilité de la liaison hélicase-ARNdb.
 | Figure 1. Structure et modèle de commutation du récepteur RIG-I. A. RIG-I est composé de deux domaines CARD (CARD1 en jaune et CARD2 en fuchsia), d’un domaine hélicase central qui comprend 3 sous-domaines (Hel1 en vert, Hel2 en orange et Hel2i, inséré dans Hel2, en bleu clair), et d’un domaine carboxy-terminal (CTD en rouge). Un domaine « pince » (en bleu foncé) relie les domaines Hel2 et CTD. Les motifs hélicase impliqués dans la liaison de l’ATP et de l’ARNdb sont indiqués (en italique souligné) respectivement en bleu (face supérieure) et en rouge (face inférieure). B. Structure cristallographique de la forme autoréprimée de RIG-I. Les domaines CARD, liés à l’hélicase au niveau du sous-domaine Hel1, interagissent avec le sous-domaine Hel2i via des interactions CARD2-Hel2i. Le domaine « pince », constitué de deux hélices α enveloppant le sous-domaine Hel1, établit une connexion mécanique entre Hel1, Hel2 et le CTD. L’emplacement de ce dernier domaine est modélisé car trop mobile dans le cristal pour être défini. Les structures sont issues de la banque de données des protéines (PDB) sous les références : 4A2Q pour le complexe CARD-hélicase et 4A2V pour le CTD. C. Modèle d’activation de RIG-I. RIG-I est présent dans le cytosol sous sa forme autoréprimée, les domaines CARD étant masqués par l’interaction entre les domaines Hel2i et CARD2. Lors d’une infection, le CTD, lié de façon flexible à l’hélicase, détecte l’extrémité 5’ triphosphate d’un ARNdb viral. Le rapprochement qui s’en suit permet à l’ARNdb d’entrer en compétition favorable avec le domaine CARD2 pour la liaison à l’hélicase. La double fixation d’ARN et d’ATP provoque un changement de conformation aboutissant au dévoilement des domaines CARD. Ces derniers recrutent alors le(s) partenaire(s) moléculaire(s) assurant la transmission du signal. |
|
Compétition des domaines CARD pour la liaison du domaine hélicase à l’ARNdb La résolution de la structure tridimensionnelle de RIG-I entière et d’une forme tronquée du CTD complexé à l’ARNdb [4] nous a permis pour la première fois de voir une structure de deux domaines CARD contigus et leur orientation dans la molécule. Les domaines CARD1 et CARD2 comprennent respectivement 7 et 6 hélices α. Ils sont reliés de manière assez rigide et forment une seule unité fonctionnelle responsable de la transmission du signal [9]. À l’état autoréprimé, le domaine CARD2 interagit avec Hel2i, ce qui empêche une liaison du domaine hélicase directement avec l’ARNdb. À l’état activé, la liaison de l’ARNdb, stabilisée par la liaison d’une molécule d’ATP au site ATPase, libère totalement les domaines CARD, les rendant ainsi capables de recruter la machinerie de signalisation conduisant à la réponse IFN. |
Modèle d’activation de RIG-I par commutation structurale Nous proposons [4] le modèle d’activation suivant : en l’absence de stimulus ARN, RIG-I est présent dans le cytoplasme sous forme autoréprimée. Les domaines CARD sont masqués par les interactions entre CARD2 et Hel2i et ne peuvent donc pas recruter leurs partenaires protéiques de signalisation. Aucun signal ne peut donc être transmis. Ce masquage des CARD est prouvé a contrario par le phénotype constitutivement actif de RIG-I après rupture de la liaison entre Hel2i et CARD2 (substitution de la phénylalanine 539 en alanine ou en aspartate). En revanche, dans cette forme autoréprimée de RIG-I, le CTD est lié de façon flexible à l’hélicase, sans interaction forte avec le reste de la protéine. Il est donc capable de détecter et lier un ARNdb 5’triphosphate avec une très forte affinité. La reconnaissance de ces motifs par le CTD conduit à une proximité d’ARNdb favorable à la compétition avec le domaine CARD2 pour la liaison à l’hélicase en présence d’ATP. Cette double liaison provoque un changement de conformation au cours duquel, comme décrit plus haut, les domaines Hel1 et Hel2 se replient l’un sur l’autre selon un mouvement torsadé avec un repositionnement du CTD et une libération des domaines CARD. Le démasquage des CARD conduit au recrutement de la cascade de signalisation gouvernant la sécrétion d’IFN et de cytokines [10, 11]. La conservation de résidus clés et l’homologie des structures déjà résolues entre les RIG-I de différentes espèces et entre RIG-I, MDA5 et LGP2 laissent présager une stratégie de commutation structurale commune probablement réversible et hautement dynamique grâce à l’activité ATPase. Par ailleurs, ces travaux illustrent l’intérêt majeur d’études comparatives du fonctionnement biologique de plusieurs espèces (exemple : le RIG-I humain entier a été réfractaire à la cristallisation [4]), et la puissance de l’analyse structurale conjuguée à l’épreuve fonctionnelle comme apport cognitif de mécanismes biologiques fondamentaux. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article. |
1. Loo YM, Gale M Jr. Immune signaling by RIG-I-like receptors . Immunity. 2011; ; 34 : :680.–692. 2. Gerlier D, Lyles DS. Interplay between innate immunity and negative strand RNA viruses: towards a rational model . Microbiol Mol Biol Rev. 2011; ; 75 : :468.–490. 3. Schlee M, Hartmann G. The chase for the RIG-I ligand: recent advances . Mol Ther. 2010; ; 18 : :1254.–1262. 4. Kowalinski E, Lunardi T, McCarthy AA, et al. Structural basis for the activation of innate immune pattern-recognition receptor RIG-I by viral RNA . Cell. 2011; ; 147 : :423.–435. 5. Luo D, Ding SC, Vela A, et al. Structural insights into RNA recognition by RIG-I . Cell. 2011; ; 147 : :409.–422. 6. Jiang F, Ramanathan A, Miller MT, et al. Structural basis of RNA recognition and activation by innate immune receptor RIG-I . Nature. 2011; ; 479 : :423.–427. 7. Civril F, Bennett M, Moldt M, et al. The RIG-I ATPase domain structure reveals insights into ATP-dependent antiviral signalling . EMBO Rep. 2011; ; 12 : :1127.–1134. 8. Lu C, Xu H, Ranjith-Kumar CT, et al. The structural basis of 5’ triphosphate double-stranded RNA recognition by RIG-I C-terminal domain . Structure. 2010; ; 18 : :1032.–1043. 9. Saito T, Hirai R, Loo YM, et al. Regulation of innate antiviral defenses through a shared repressor domain in RIG-I and LGP2 . Proc Natl Acad Sci USA. 2007; ; 104 : :582.–587. 10. Belgnaoui SM, Paz S, Hiscott J. Orchestrating the interferon antiviral response through the mitochondrial antiviral signaling (MAVS) adapter . Curr Opin Immunol. 2011; ; 23 : :564.–572. 11. Loo YM, Gale MJ. Immune signaling by RIG-I-like receptors . Immunity. 2011; ; 34 : :680.–692. 12. Imler JL, Ferrandon D. Le printemps de l’immunité innée couronné à Stockholm. Prix Nobel de Médecine 2011 . Med Sci (Paris). 2011; ; 27 : :1019.–1024. |