| |
| Med Sci (Paris). 2011 August; 27(8-9): 781–786. Published online 2011 August 31. doi: 10.1051/medsci/2011278022.Chroniques génomiques L’insoutenable complexité des cancers Bertrand Jordan1* 1CoReBio PACA, case 901, Parc scientifique de Luminy, 13288Marseille Cedex 9, France MeSH keywords: Animaux, Tumeurs du sein, génétique, Analyse de mutations d'ADN, ADN tumoral, Femelle, Gènes tumoraux, Hétérogénéité génétique, Génome humain, Humains, Mâle, Myélome multiple, Mutation, Protéines tumorales, Tumeurs, épidémiologie, Analyse de séquence d'ADN |
Si l’on en croit les magazines et les émissions de télévision - même celles qui sont programmées sur les chaînes dites « sérieuses » -, une incidence sans cesse croissante des cancers, due aux additifs alimentaires, à la pollution, au stress, aux lignes électriques à haute tension, aux centrales nucléaires, aux OGM, etc. est en passe d’aboutir à une situation sanitaire catastrophique pour nous et plus encore pour nos enfants. Les lecteurs de médecine/sciences savent qu’il n’en est rien, et que l’augmentation bien réelle du nombre des cas est essentiellement liée au vieillissement de la population. Les courbes d’incidence corrigées pour l’âge (c’est-à-dire relatives à un ensemble de personnes dont l’âge moyen est le même pour les différentes périodes considérées) sont en fait - si l’on en croit les dernières statistiques du National cancer institute et son tout récent « Rapport à la nation » (Annual report to the nation on the status of cancer
2
) -, en légère décroissance pour les hommes, en plateau pour les femmes. La différence entre les sexes est essentiellement due au cancer du poumon, dont l’incidence a diminué de plus de 25 % pour les uns et doublé pour les autres - il s’agit là bien sûr de l’influence du tabac et du décalage temporel entre hommes et femmes dans le pic de consommation de ce cancérigène avéré. Face à l’image populaire et catastrophiste d’une « épidémie de cancers », la communauté scientifique et médicale, elle, diffuse souvent une vision optimiste, selon laquelle la génomique découvre les mécanismes sous-jacents à ces affections et débouche sur la mise au point de traitements ciblés plus efficaces et moins lourds que les chimiothérapies traditionnelles. La « médecine personnalisée » deviendrait ainsi une réalité, autorisant un ajustement précis du traitement à chaque patient, avec une amélioration continue des résultats permettant d’espérer soit la guérison, soit au moins la transformation du cancer en une maladie chronique permettant une longue survie avec une qualité de vie acceptable. Il faut cependant reconnaître que cette révolution génomique n’en est qu’à ses débuts, que les thérapies traditionnelles (chirurgie, chimiothérapie et radiothérapie) restent très largement dominantes, et que le pourcentage de tumeurs effectivement analysées en biologie moléculaire est encore faible, même dans les centres les plus performants. En dehors des pesanteurs sociologiques et corporatistes, et de réels problèmes financiers, cela traduit le fait que le catalogue des mutations oncogéniques reste très incomplet et que la panoplie d’agents spécifiques utilisable est encore restreinte. |
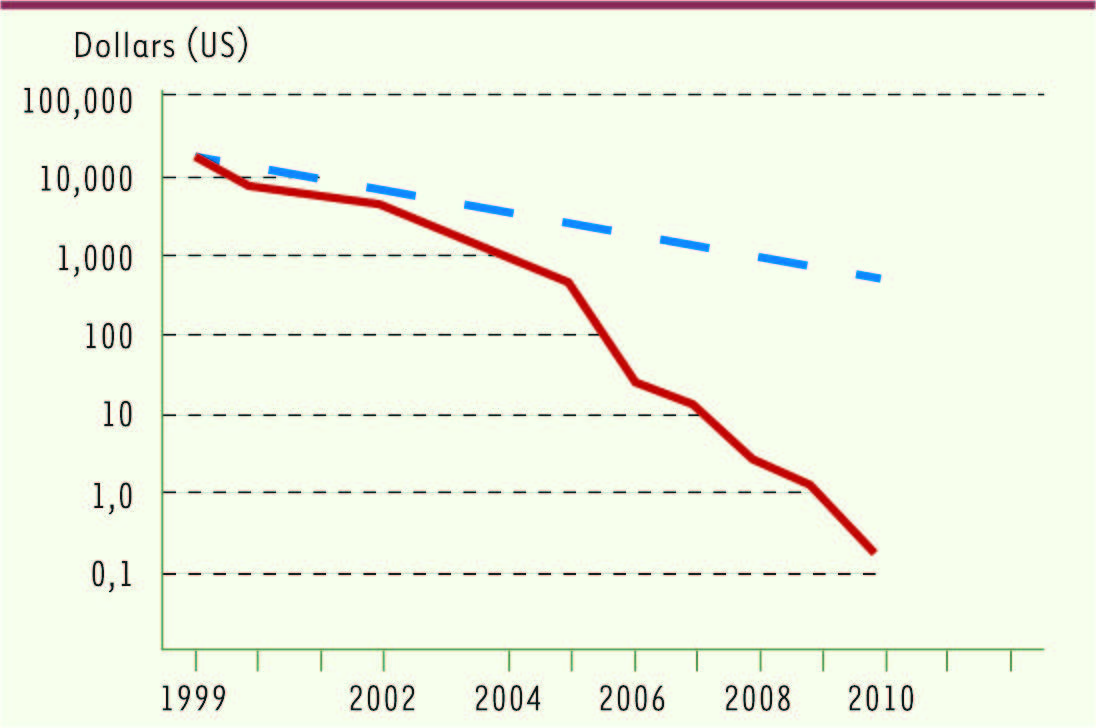
Le séquençage à la rescousse L’incroyable accélération des possibilités de séquençage au cours des cinq dernières années - le coût par mégabase séquencée est tombé d’un facteur supérieur à 1 000 au cours de cet intervalle [
1] (Figure 1) - offre a priori de grands espoirs. Après toutes ces années où la vision moléculaire des changements survenus dans la cellule cancéreuse était forcément incomplète et grossière, le séquençage intégral de l’ADN d’une tumeur et du tissu normal correspondant devrait fournir enfin un catalogue complet des événements survenus lors de l’oncogenèse et révéler les cibles vers lesquelles diriger les traitements à venir. On peut même rêver à la mise en œuvre d’un « séquençage clinique », grâce auquel l’évolution des cellules tumorales au cours de la maladie serait suivie par séquençage périodique, permettant ainsi l’adaptation continue des traitements afin d’atteindre une efficacité optimale.
 | Figure 1.
Évolution du coût du séquençage. Le prix est exprimé en dollars US par mégabase de séquence brute (courbe en trait plein) et comparé au coût de calcul (ligne pointillée correspondant à la loi de Moore, soit un facteur 2 tous les dix-huit mois) (d’après les données diffusées par le Broad Institute). |
Mais il a fallu déchanter : les premières séquences globales de tumeurs, publiées en 2008 et 2009 (voir la revue de Wooster et Bachman [
2] qui fait le point sur les travaux jusqu’à début 2010), ont surtout révélé l’incroyable diversité des mutations présentes dans l’ADN des cellules tumorales. Qu’il s’agisse de leucémies, de cancer du poumon, de mélanome ou de cancer du sein, les altérations par rapport à l’ADN normal se comptent par milliers. Citons, par exemple, les 22 901 substitutions révélées dans une lignée de cancer du poumon à petites cellules, « une mutation pour chaque quinzaine de cigarettes fumées » selon les auteurs3 [
3], ou encore les 33 345 mutations, 983 insertions/délétions, et 37 translocations trouvées dans un mélanome [
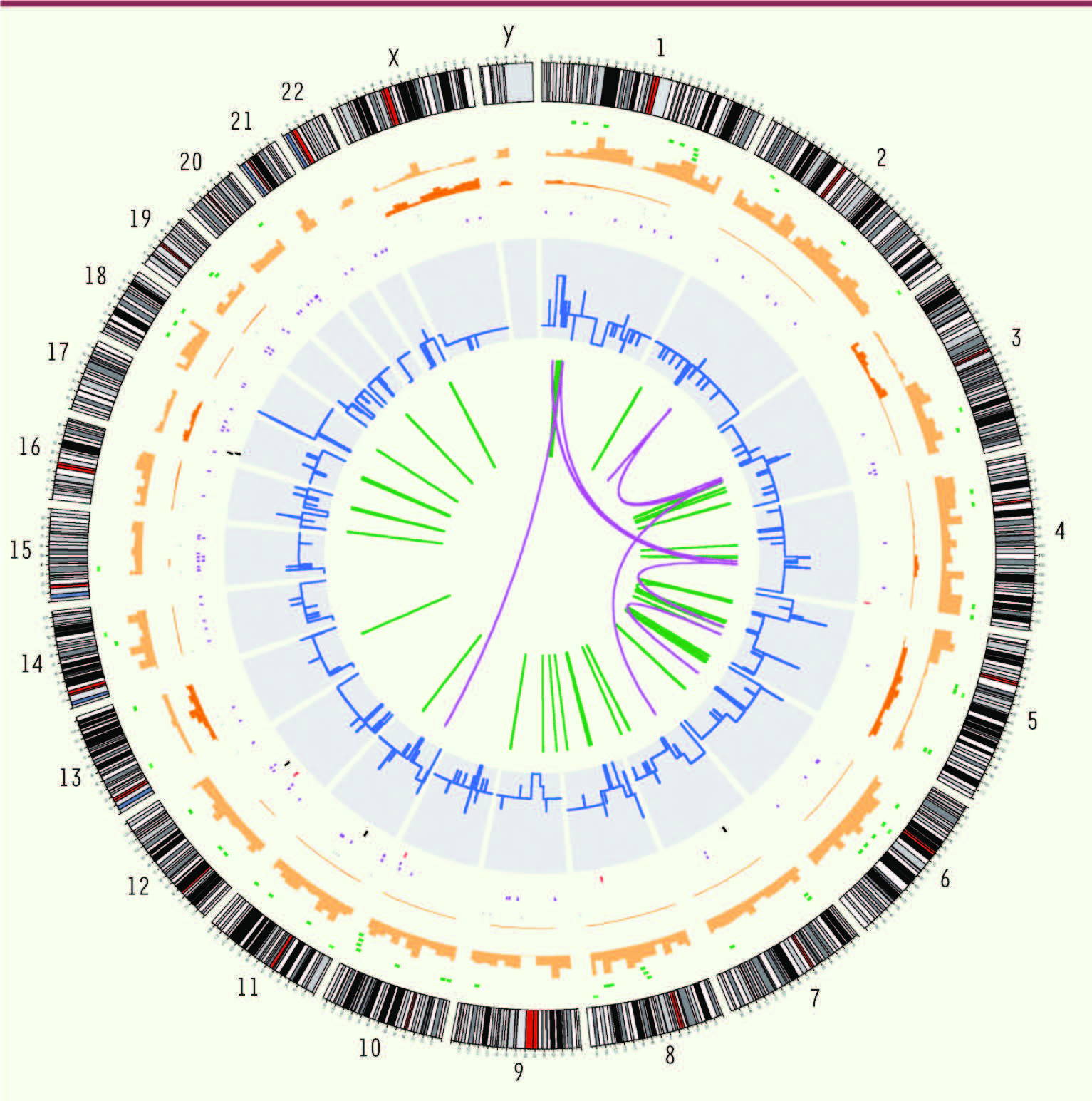
4] (Figure 2).
 | Figure 2.
L’extrême variété des changements observés dans l’ADN d’un cancer du poumon (par rapport au tissu normal). Les chromosomes sont figurés à la périphérie du cercle. En allant vers l’intérieur, on trouve les insertions (vert clair), les délétions (vert foncé), les substitutions hétérozygotes (orange clair) et homozygotes (orange foncé) repérées par leur densité par intervalle de dix mégabases, les substitutions dans des régions codantes : silencieuses (gris), faux-sens (pourpre), nonsens (rouge) et dans les sites d’épissage (noir). Les variations de nombre de copies sont indiquées en bleu, les zones de perte d’hétérozygotie en rouge, les réarrangements intra-chromosomiques en vert et inter-chromosomiques en pourpre (figure aimablement donnée par Peter Campbell, Wellcome Trust Sanger Institute). |
Bien entendu, si l’on se limite aux mutations qui affectent les régions codantes des gènes et modifient la séquence des protéines correspondantes (faux-sens et non-sens), les chiffres deviennent moins effrayants : 187 substitutions de ce type, par exemple, pour le mélanome [4]. Cela fait encore beaucoup, d’autant plus que des substitutions situées en dehors des exons peuvent tout à fait modifier l’expression d’un ou plusieurs gènes et donc être elles aussi impliquées dans l’oncogenèse. Un examen attentif des gènes touchés par ces mutations (et surtout, bien sûr, par celles qui entraînent des codons stop) permet d’en retrouver certains dont l’implication dans le cancer était déjà connue, ce qui est rassurant. Quant aux autres, plusieurs appartiennent aux mêmes circuits (pathways) que les précédents, ce qui renforce la probabilité de leur implication - il reste que beaucoup des altérations constatées ne sont probablement pas causales : ce sont des « passagers » apparus en raison de l’instabilité générale de la cellule cancéreuse, mais qui ne sont pas impliqués dans sa genèse. Il est donc essentiel d’arriver à distinguer les mutations causales (drivers) des autres (passengers) - et l’on ne dispose guère pour cela que d’un moyen : la répétition, c’est-à-dire, en pratique, le séquençage itératif de nombreuses tumeurs. |
Des milliers de génomes ? On peut en principe procéder à cette opération de manière ciblée : selon une stratégie qui s’apparente à celle du séquençage des exomes, choisir quelques dizaines de gènes candidats, sélectionner l’ADN correspondant dans celui de nombreuses tumeurs et procéder à son séquençage. On peut aussi séquencer l’ensemble de l’exome (examinant ainsi tous les gènes, mais faisant l’impasse sur les régions non codantes), ou bien encore lire carrément le génome dans son intégralité. Cette dernière approche présente l’avantage d’être exhaustive, et, comme elle devient de plus en plus abordable, elle semble être actuellement privilégiée. Je vais donc, dans le cadre de cette chronique, me limiter à elle, tout en rappelant que d’autres techniques (et notamment l’étude du transcriptome par RNA-seq) peuvent, elles aussi, apporter des informations utiles. Exemple 1 : séquençage en masse du génome de cellules de myélome multiple En fait le séquençage en masse de génomes de cancer est surtout actuellement à l’état de work in progress, et les publications sont peu nombreuses. Parmi les opérations en cours, il faut évidemment citer le Cancer Genome Atlas
4, qui prévoit à terme de séquencer 50 000 couples tumeur/tissu normal, et en a déjà programmé 3 000 pour cette année. Il est vrai que - du moins pour le moment - il s’agit le plus souvent d’exomes (seuls 10 % des échantillons sont intégralement séquencés), mais la moisson de données promet d’être considérable. Au rythme actuel, cela représente déjà plus de 20 téraoctets de données par mois, dont le stockage, le transfert et surtout l’analyse posent de redoutables problèmes logistiques. En attendant l’arrivée des résultats de ces grands projets, on peut déjà s’intéresser à une étude publiée, et à une autre qui l’est « presque ». La première, parue fin mars 2011 dans Nature [
5], rapporte le séquençage de l’ADN des cellules de 38 tumeurs de type myélome multiple (plus les témoins correspondants), effectué en intégral pour 23 d’entre elles et « en exome » pour les autres. En moyenne, les auteurs trouvent pour chaque tumeur plus de 7 000 mutations ponctuelles, dont 30 à 40 substitutions faux-sens et non-sens, et une bonne vingtaine de réarrangements chromosomiques ayant le même effet. Et surtout, rien de vraiment systématique à première vue. Grâce à une analyse plus approfondie, ils parviennent à sélectionner dix gènes « significativement mutés » ( significantly mutated genes), c’est-à-dire dans lesquels des mutations sont retrouvées de manière statistiquement très significative par rapport au bruit de fond. Mais, à deux exceptions près ( KRAS et NRAS), ces gènes ne sont altérés que dans 5 à 10 % des tumeurs séquencées. KRAS et NRAS étaient déjà connus en tant que gènes mutés dans le myélome multiple, tout comme TP53 qui fait également partie du lot de dix, ainsi que CCND1 (gène codant pour la cycline D1) , fréquemment affecté par des translocations dans cette affection. On retrouve donc trois ou quatre gènes déjà impliqués dans ce cancer, ce qui, une fois encore, rassure sur la validité de l’approche. Les nouveaux gènes « significatifs », eux, fournissent autant de pistes pour une meilleure compréhension de l’oncogenèse. Certains constitueront sans doute, à terme, des cibles pour des thérapies spécifiques, mais les espoirs que l’on pouvait encore nourrir de ramener cette affection à un petit nombre d’altérations géniques s’éloignent. Exemple 2 : séquençage en masse du génome de cellules de cancer du sein Le deuxième de ces projets de séquençage à grande échelle ne nous est pour le moment connu que par un résumé présenté à la réunion d’avril 2011 de l’AACR ( American Association for Cancer Research
5) et par une nouvelle publiée en ligne dans la revue Nature [
6]. Il s’agit du séquençage de 50 tumeurs mammaires (et du tissu normal correspondant). La catégorie choisie est celle des cancers de type luminal, un sous-type fréquent qui peut être soit sensible, soit résistant au traitement par les estrogènes [
7], et ce séquençage a été organisé dans le cadre d’un essai clinique, ce qui garantit un suivi de qualité du devenir des malades. Les auteurs avaient fait porter leur étude sur ces deux catégories (24 tumeurs sensibles, 26 résistantes) dans l’espoir, bien sûr, d’observer des spectres de mutation différents et de pouvoir en tirer des indications pronostiques et thérapeutiques. Au terme de ce travail, ils indiquent avoir lu en tout «10 trillions de bases 6, » et on sent bien, du moins dans leurs commentaires, leur effarement devant les résultats obtenus : « Les résultats sont complexes et un peu alarmants, ils vous obligent à repenser ce que peut être le cancer du sein », dit à peu près Matthew Ellis, le responsable de l’équipe impliquée 7. En fait, 1 700 mutations au total ont été repérées dans ces tumeurs, mais la plupart n’apparaissent que dans un seul échantillon, seules trois sont présentes dans 10 % ou plus des tumeurs. Il n’émerge pas non plus de distinction claire entre tumeurs sensibles et résistantes, à part le fait que le nombre de mutations faux-sens tend à être plus important dans la deuxième catégorie. Les trois gènes les plus fréquemment mutés sont PIK3CA (dans 43 % des tumeurs), TP53 (15 %) et MAP3K1 (9 %) - ce dernier constituant une nouveauté et semblant caractéristique des tumeurs de type luminal (par rapport au type basal, l’autre grande catégorie pour le cancer du sein). Les auteurs annoncent (dans leur résumé de l’AACR) une analyse détaillée montrant que nombre des gènes mutés (à faible fréquence) appartiennent à quelques circuits particuliers, et formulent l’espoir qu’une analyse en termes de voies d’activation oncogénique permette de regrouper à chaque fois plusieurs gènes, de mettre un peu d’ordre dans cette multitude d’évènements mutationnels observés - et d’en tirer des indications pronostiques et thérapeutiques. Et, dans ce but, ils ont d’ores et déjà engagé un nouveau projet, qui vise cette fois à séquencer 1 000 tumeurs. |
Cet acharnement à séquencer encore et encore fait un peu penser à ce qu’écrivait Ivan Illich, prophète aujourd’hui bien oublié de la « convivialité ». Dans les années 1970, il qualifiait de contre-productive la tendance des sociétés technologiques à traiter les problèmes causés par une innovation en amplifiant cette même approche [
8] : des autoroutes pour répondre aux nuisances des voitures, des transports à grande vitesse qui en réalité font perdre du temps, une hyper-communication que plus personne n’écoute vraiment. La réponse recherchée ici (une meilleure compréhension de la cellule cancéreuse débouchant sur une réelle amélioration des traitements) découlera-t-elle automatiquement de l’accumulation des données de séquence ? Il est permis d’en douter, il faudra y ajouter beaucoup d’intelligence et de renseignements fonctionnels. Les chercheurs en sont bien sûr conscients, et les efforts déjà mentionnés d’interprétation en termes de circuits vont certainement dans la bonne direction. La combinaison des investigations avec des essais cliniques est également importante pour se donner les meilleures chances de relier la séquence et la pathologie. Et, devant la complexité de la situation au niveau génomique, on peut se demander si finalement les profils d’expression, un peu passés de mode aujourd’hui, ne méritent pas une attention plus soutenue ? Ils renseignent en effet sur les principales activités en cours à l’intérieur de la cellule cancéreuse, sans s’égarer dans la comptabilité d’innombrables substitutions dont seules quelques-unes sont réellement importantes et causales. Les nouvelles machines à séquencer permettent aussi d’établir des spectres d’expression « digitaux », bien plus précis que ceux que fournissent les puces à ADN, et il serait sans doute indiqué d’amplifier les efforts dans cette direction. En tout cas, force est de constater que l’irruption du séquençage massif en cancérologie, pour positive qu’elle soit et prometteuse à terme, ne semble pas être l’arme absolue qui permettrait de gagner enfin cette « guerre contre le cancer » déclarée en 1971 sous la présidence de Richard Nixon. Conflit d’intérêts
L’auteur déclare n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Nous remercions Peter Campbell (Wellcome Trust Sanger Institute, Hinxton, Cambridge, United Kingdom) pour nous avoir permis d'utiliser la Figure 2.
|
Footnotes |
1.
Jordan
B
. Le boom des séquenceurs nouvelle génération . Med Sci (Paris).
2010; ; 26 : :325.-327. 2.
Wooster
R
,
Bachman
KE
. Catalogue, cause, complexity and cure; the many uses of cancer genome sequence . Curr Opin Genet Dev.
2010; ; 20 : :336.-341. 3.
Pleasance
ED
,
Stephens
PJ
,
O’Meara
S
, et al.
A small-cell lung cancer genome with complex signatures of tobacco exposure . Nature.
2010; ; 463 : :184.-192. 4.
Pleasance
ED
,
Cheetham
RK
,
Stephens
PJ
, et al.
A comprehensive catalogue of somatic mutations from a human cancer genome . Nature.
2010; ; 463 : :191.-196. 5.
Chapman
MA
,
Lawrence
MS
,
Keats
JJ
, et al.
Initial genome sequencing and analysis of multiple myeloma . Nature.
2011; ; 471 : :467.-472. 6.
Wadman
M
. Fifty genome sequences reveal breast cancer’s complexity . Nature News.
2011, 2 avril (online) doi: 10.1038/news.2011.203.. 7.
Bertucci
F
,
Finetti
P
,
Cervera
N
,
Birnbaum
D
. Classification pronostique du cancer du sein et profils d’expression génique sur puces à ADN . Med Sci (Paris).
2008; ; 24 : :599.-606. 8.
Ivan
Illich
. Libérer l’avenir . Paris: : Seuil; , 1971
(titre original : Celebration of awareness).. |