| |
| Med Sci (Paris). 2011 December; 27(12): 1127–1130. Published online 2011 December 23. doi: 10.1051/medsci/20112712020.Chroniques génomiques Séquençage de nouvelle génération : déjà en clinique ? Bertrand Jordan1* 1CoReBio PACA, case 901, Parc scientifique de Luminy, 13288Marseille Cedex 9, France MeSH keywords: Interprétation statistique de données, Femelle, Dépistage génétique, économie, éthique, normes, tendances, Séquençage nucléotidique à haut débit, utilisation, Humains, Nouveau-né, Tumeurs, diagnostic, génétique, Périnatologie, méthodes, Types de pratiques des médecins, Grossesse, Contrôle de qualité, Analyse de séquence d'ADN |
Précisons tout d’abord le sujet de cette chronique : il ne s’agit pas ici de détailler les nombreuses applications du séquençage ultra-rapide en recherche médicale – nous en avons déjà parlé plusieurs fois – mais bien de faire un point sur l’utilisation effective de cette technique dans la pratique clinique. Et le séquençage à visée diagnostique d’un ou deux gènes, dont l’exemple typique est le fructueux business construit par Myriad Genetics autour des gènes BRCA1 et BRCA2, n’entre pas non plus dans le cadre de cet article. En fait, je souhaite décrire ici les cas où l’obtention de quelques gigabases de séquence brute1 s’intègre effectivement dans une pratique de soins – ou va le faire dans un futur proche. |
Commençons par un rapide panorama des possibilités actuelles de la technologie NGS (new generation sequencing). Le sujet est éminemment évolutif [
1] et, compte tenu de la concurrence effrénée que se livrent les principaux intervenants (Illumina, Life Technologies et Complete Genomics, Roche/454 étant désormais un peu marginalisé en raison de son coût bien plus élevé malgré l’avantage de lectures plus longues), il est parfois difficile de distinguer ce qui est réellement disponible aujourd’hui des projections plus ou moins optimistes à un, deux ou trois ans. Disons, pour fixer les idées, que la séquence complète d’un génome humain est aujourd’hui offerte à un tarif de l’ordre de cinq mille dollars, celle d’un exome à un peu plus de mille (Figure 1). Encore faut-il savoir de quelle séquence on parle : le coût dépend énormément de la qualité2, et la « qualité recherche » qui correspond aux prix indiqués ci-dessus n’est pas forcément suffisante pour une utilisation clinique. Et, par ailleurs, l’interprétation des résultats peut s’avérer longue et coûteuse [
2]. La précision annoncée par Complete Genomics pour la séquence complète (grâce à une redondance de cinquante environ) est de un sur cent mille – le chiffre paraît impressionnant, mais cela signifie que les six gigabases du génome obtenu peuvent contenir soixante mille erreurs, ce qui rend délicate la détection d’éventuelles mutations. Pour le séquençage d’exomes, la redondance adoptée est en général de l’ordre de cent, ce qui améliore nettement la qualité mais laisse encore une bonne marge d’incertitude. Il faut savoir, de plus, que la valeur annoncée est celle de la redondance moyenne : en fait, certaines zones seront lues cent ou deux cents fois mais d’autres n’auront bénéficié que de dix ou vingt lectures. Tout cela montre à l’évidence que la séquence obtenue doit faire l’objet d’une évaluation précise, et que sa qualité doit être adaptée à l’usage prévu. En ce qui concerne l’interprétation, une utilisation clinique suppose un traitement informatique rodé et automatisé des données, aboutissant à une réponse binaire ou du moins quantifiée, directement utilisable par le clinicien : il n’est pas question de faire travailler durant six mois une équipe de bioinformaticiens pour éplucher les résultats, comme ce fut le cas pour une publication récente [
3]. Et bien sûr, tout cela doit aller vite, avec un délai de rendu de l’ordre de la semaine : en clinique, on a toujours besoin des résultats « pour avant-hier ».
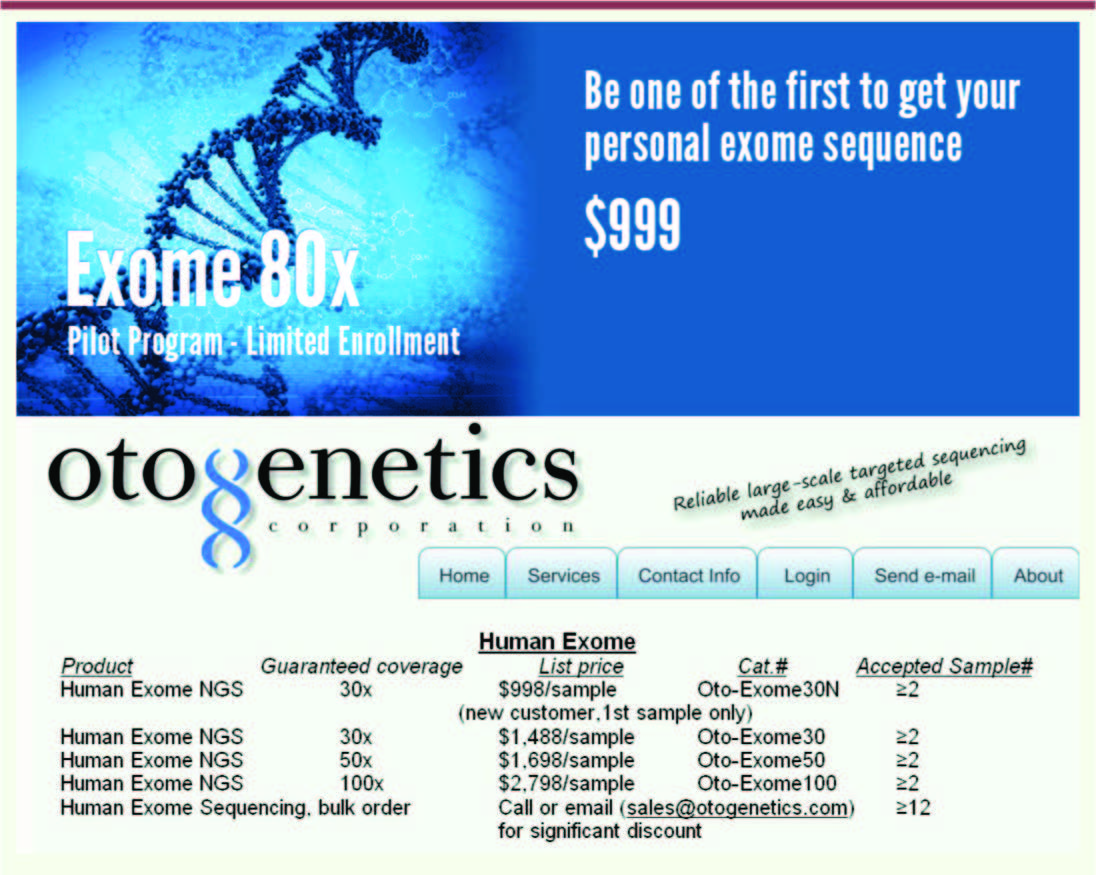
 | Figure 1.
Deux offres commerciales de séquençage d’exomes. En haut, celle de 23andMe (résultats fournis bruts, sans interprétation), en bas celle de l’entreprise Otogenetics (Tucker, États-Unis) qui est plus détaillée. Noter la variation des tarifs en fonction de la redondance désirée. Dans le cas d’Otogenetics, une analyse assez basique est comprise, l’analyse plus détaillée est facturée en sus (« prix sur demande »). Noter que dans les deux cas il s’agit de tarifs promotionnels. |
Rappelons encore, pour fixer les idées, qu’à l’heure actuelle le marché du séquençage est assez segmenté avec d’une part les machines haut de gamme (Illumina HiSeq 2000, Life Technologies SOLiD 5500) qui produisent plusieurs centaines de gigabases en une session d’une semaine, d’autre part les (nouveaux) « séquenceurs personnels » (Ion Torrent/Life Technologies personal genome machine, ou Illumina MiSeq), fournissant cent fois moins de séquences en quelques heures mais à un tarif (par base) nettement plus élevé, et qui sont peut-être mieux adaptés à un environnement clinique, et enfin les usines à séquencer comme Complete Genomics ou le chinois BGI, qui revendique avoir déterminé dix mille séquences humaines. |
Des applications effectives : du préconceptionnel au périnatal Malgré les limites rappelées ci-dessus, il existe déjà des mises en œuvre réelles du séquençage NGS en clinique. Une chronique récente a présenté une application prometteuse, la détection des porteurs hétérozygotes de mutations récessives [
4]. Rappelons qu’il s’agit de faire un large inventaire, en situation préconceptionnelle, des mutations récessives présentes chez chaque membre d’un couple afin, en cas de risque avéré, de prendre les mesures nécessaires pour éviter la naissance d’un enfant atteint (par exemple en passant par la fécondation in vitro et un diagnostic préimplantatoire). En pratique, on procède au séquençage d’un exome restreint à quelques centaines de gènes, puisque la quasi-totalité des mutations pathogènes connues est située dans la partie exonique des gènes. Un service commercial fondé sur l’emploi d’un microarray est déjà proposé3,, mais je présenterai ici un test plus évolué, mis au point dans un cadre académico-hospitalier et fondé sur le séquençage4 [
5]. Développé par le Children’s Mercy Hospital à Kansas City (États-Unis) et actuellement en fin de validation, ce test consiste en une analyse portant sur 592 maladies, qui sera utilisée en interne avant la fin de l’année puis offerte à l’extérieur courant 2012. Le coût, 600 dollars US, est remarquablement bas lorsqu’on songe que l’examen d’un seul gène est couramment facturé 500 à 1 000 dollars, sans parler du séquençage de BRCA1 et 2 qui coûte, lui, plus de 3 000 dollars. En résumé, l’exome à séquencer est sélectionné grâce au système Agilent sure select (hybridation en solution), puis lu sur une machine Illumina HiSeq 2000 en multiplexant jusqu’à 96 échantillons. Dans sa version actuelle, le test aurait une sensibilité de 95 % et une spécificité de 99,99 %, ce qui est assez remarquable, et la version suivante, déjà annoncée pour 2013, testerait 1 200 gènes pour 300 dollars. Le délai est actuellement de quatre semaines. En fait, ce test est actuellement utilisé comme diagnostic pour les enfants présentant un problème d’origine encore inconnue, et permet de balayer en une fois toute une gamme d’étiologies possibles, ce qui limite l’errance diagnostique (diagnostic odyssey) tout en réduisant considérablement les coûts. L’expérience acquise dans cet emploi permettra ensuite de le proposer en situation préconceptionnelle. Restent quelques problèmes non résolus : le fait que beaucoup des gènes examinés sont couverts par des brevets, ce qui en principe impose l’obtention de nombreuses licences ; et la question des variants inconnus détectés, et dont on ignore l’impact physiologique : qu’en faire, faut-il ou non les signaler au médecin, au malade ? |
De la « séquence utile » pour les cancers Une autre application du séquençage à haut débit se situe en cancérologie. Malgré l’extrême et décourageante complexité révélée par l’analyse détaillée de nombreuses tumeurs [
6], le séquençage semble y trouver sa place aujourd’hui. Il ne s’agit plus de faire l’inventaire de toutes les altérations présentes dans l’ADN d’une cellule tumorale, mais de s’intéresser uniquement aux gènes dont l’implication dans le processus d’oncogenèse est connue et pour lesquels existe (ou a des chances d’exister bientôt) une thérapie spécifique5. On peut alors définir une liste de 100 à 200 gènes, isoler l’ADN correspondant et le séquencer à une redondance élevée (100 fois, par exemple) pour repérer mutations et délétions dans tous les gènes d’intérêt. Il n’est pas nécessaire, du coup, d’utiliser un panel spécialisé pour chaque type de tumeur, la liste est suffisamment large pour couvrir l’ensemble – d’autant plus que nombre d’altérations initialement considérées comme spécifiques d’un type tumoral ont depuis été retrouvées dans plusieurs autres. Les tests qui sont actuellement en cours de validation examinent 176 gènes pour celui que met au point l’entreprise Foundation Medicine (Cambridge, États-Unis) [
7], ou 128 pour celui qui est développé au Dana-Farber Cancer Institute (Boston, États-Unis) [
8]. La sélection de l’ADN à séquencer est faite, une fois encore, grâce à l’hybridation en solution, et le séquençage peut être fait soit sur une machine haut de gamme (Illumina HiSeq) en multiplexant les échantillons, soit, pour les cas urgents, sur un des nouveaux systèmes cliniques (Ion Torrent/LifeTechnologies ou Illumina MiSeq) qui donnent des résultats plus rapidement – mais à un coût plus élevé. Les résultats des études pilotes sont encourageants, avec un délai de réponse inférieur à un mois, la détection fréquente de mutations déjà connues et une amorce de piste thérapeutique dans presque chaque cas6. Les choses sont donc assez avancées ; d’ailleurs, en Grande-Bretagne, le National Health Service a lancé un appel pour de tels tests, avec un plafond de coût fixé à 300 livres sterling, soit 350 euros environ. Notons que dans ces projets (qui sont maintenant plus que des projets) l’éventail des gènes étudiés est volontairement restreint, et pas seulement pour des raisons financières : il ne s’agit plus de découverte, mais réellement d’application clinique, et l’on n’a que faire, dans ce cadre, de mutations que l’on ne sait pas interpréter. Évidemment, pour l’une comme pour l’autre de ces applications qui font déjà partie d’une réalité clinique, les impératifs règlementaires sont importants : au minimum, le laboratoire comme les procédures d’interprétation doivent être certifiés (CLIA7 aux États-Unis). Comme déjà mentionné, la question des brevets se pose : Myriad va-t-il attaquer ceux qui proposent un test général incluant, comme c’est logique, les gènes BRCA ? Et le délai de rendu des résultats est critique, d’autant plus que la logique économique pousserait à un multiplexage intensif (mais chronophage) afin de rentabiliser ces machines très performantes, capables aujourd’hui de produire plusieurs centaines de gigabases par session. Notons enfin que l’approche par les exomes semble s’imposer. J’étais assez sceptique à son sujet à l’origine, mais l’efficacité surprenante des techniques de purification du type Agilent sure select (qui parviennent, ce qui est remarquable, à purifier à 50 % environ la fraction exome qui ne représente que 1 % à 2 % de l’ADN au départ), et les impératifs cliniques (simplifier l’interprétation et ne pas s’encombrer d’altérations dont on ne connaît pas le corrélat physiologique) expliquent sans doute ce succès [
9]. |
D’autres entrées pour le séquençage clinique Au-delà de ces application déjà bien développées et entrées dans la pratique clinique, au moins pour une phase d’essai, bien d’autres approches faisant intervenir le séquençage de quelques mégabases d’ADN et les machines à haut débit sont actuellement étudiées. On peut mentionner à cet égard les travaux en cours dans le cadre de l’agence du cancer de la Colombie britannique sur la stratification de malades souffrant de leucémies myéloïdes aiguës. Il s’agit là de procéder, grâce à un séquençage simultané d’exomes et du transcriptome, à un examen plus approfondi que l’analyse cytogénétique classique pour décider comment orienter les malades vers une chimiothérapie ou une greffe de moelle osseuse – on attend surtout du séquençage qu’il clarifie le cas de ceux qui, du point de vue cytogénétique, se situent dans une catégorie intermédiaire. Bien que le nombre actuellement connu de mutations, de fusions géniques et de CNV (copy number variation) soit relativement limité, le séquençage permet d’emmagasiner l’ensemble des informations et de les interpréter à l’avenir à la lumière de nouvelles découvertes. L’étude est actuellement menée de manière rétrospective sur 184 échantillons, et sera suivie d’une étude prospective sur une centaine de patients avant que le test ne soit introduit en pratique clinique. Les performances des séquenceurs de nouvelle génération (y compris les « petites » machines comme celle de Ion Torrent/Life Technologies) permettent aussi de revisiter les analyses de pathogènes et d’en proposer des versions beaucoup plus complètes à un tarif accessible. Par exemple, selon une équipe canadienne utilisant le système Roche/454, le séquençage d’ADN viral dans des taches de sang séché permet d’évaluer les risques de résistance aux antiviraux chez les patients séropositifs [
10] ; quant à l’entreprise Pathogenica (Cambridge, États-Unis), elle annonce le lancement d’un test de typage des papillomavirus (HPV) plus sensible et plus fiable que les tests actuels, employant la machine commercialisée par Life Technologies. Bien entendu, comme il s’agit ici d’analyses relativement simples (en termes de quantité de séquence par patient), un multiplexage massif est nécessaire pour que le coût soit concurrentiel. |
Cette chronique déjà longue est loin d’épuiser le sujet, ou même d’en évoquer toutes les facettes. Trois aspects importants devraient encore être évoqués : les critères de qualité auxquels doit répondre une séquence à usage clinique, la question de l’interprétation de la séquence par les entreprises qui proposent déjà un tel service, et, enfin, inévitablement, les questions éthiques : quid du consentement éclairé dans ce cadre, que faire des données inattendues dont la signification est actuellement incertaine mais qui peuvent prendre un sens à l’avenir compte tenu des avancées de la recherche ? Tout cela justifiera sans doute une prochaine chronique… Conflit d’intérêts
L’auteur déclare n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Footnotes |
1.
Jordan
B
. Séquence : le juste prix . Med Sci (Paris).
2010; ; 25 : :988.–989. 2.
Jordan
B
. La séquence… et après ?
Med Sci (Paris).
2010; ; 26 : :655.–658. 3.
Ashley
EA
,
Butte
AJ
,
Wheeler
MT
, et al.
Clinical assessment incorporating a personal genome . Lancet.
2010; ; 375 : :1525.–1535. 4.
Jordan
B
. Le véritable « génome personnel » . Med Sci (Paris).
2011; ; 27 : :553.–558. 5.
Bell
CJ
,
Dinwiddie
DL
,
Miller
NA
, et al.
Carrier testing for severe childhood recessive diseases by next-generation sequencing . Sci Transl Med.
2011; ; 12 : :3.–65ra4. 6.
Jordan
B
. L’insoutenable complexité des cancers . Med Sci (Paris).
2011; ; 27 : :781.–786. 9.
Jordan
B. Exomes
, le retour… . Med Sci (Paris).
2011; ; 27 : :220.–222. 10.
Ji
H
,
Li
Y
,
Graham
M
, et al.
Next-generation sequencing of dried blood spot specimens: a novel approach to HIV drug-resistance surveillance . Antivir Ther.
2011; ; 16 : :871.–878. |