| |
| Med Sci (Paris). 2011 December; 27(12): 1089–1095. Published online 2011 December 23. doi: 10.1051/medsci/20112712015.ATAD3, une ATPase membranaire mitochondriale vitale impliquée dans la progression tumorale Shuijie Li1 and Denis Rousseau1* 1Laboratoire de bioénergétique fondamentale et appliquée, Inserm U1055, Université Joseph Fourier, 2280, rue de la Piscine, BP 53, 38041Grenoble Cedex 9, France |
ATAD3 (ATPase family AAA domain-containing protein 3) est une ATPase découverte en 2003, identifiée d’abord parce que son gène est une cible de c-MYC [
1] puis comme protéine de la membrane mitochondriale interne [
2]. Elle appartient à la très grande famille des AAA-ATPases (ATPases associated with diverse cellular activities). Cette famille regroupe des protéines à activité ATPasique impliquées dans de nombreux processus cellulaires comme la transcription, la réplication, la traduction, la protéolyse et le transport vésiculaire [
3]. Cependant, la fonction d’ATAD3 n’est pas encore connue bien que l’ensemble des études lui reconnaissent un rôle important. En effet, son expression est ubiquitaire, et elle est essentielle à l’embryogenèse, à la structuration du réseau mitochondrial et aux interactions fonctionnelles entre mitochondries et réticulum endoplasmique [
4–
7]. ATAD3 participe donc à une fonction mitochondriale vitale qui reste à découvrir. |
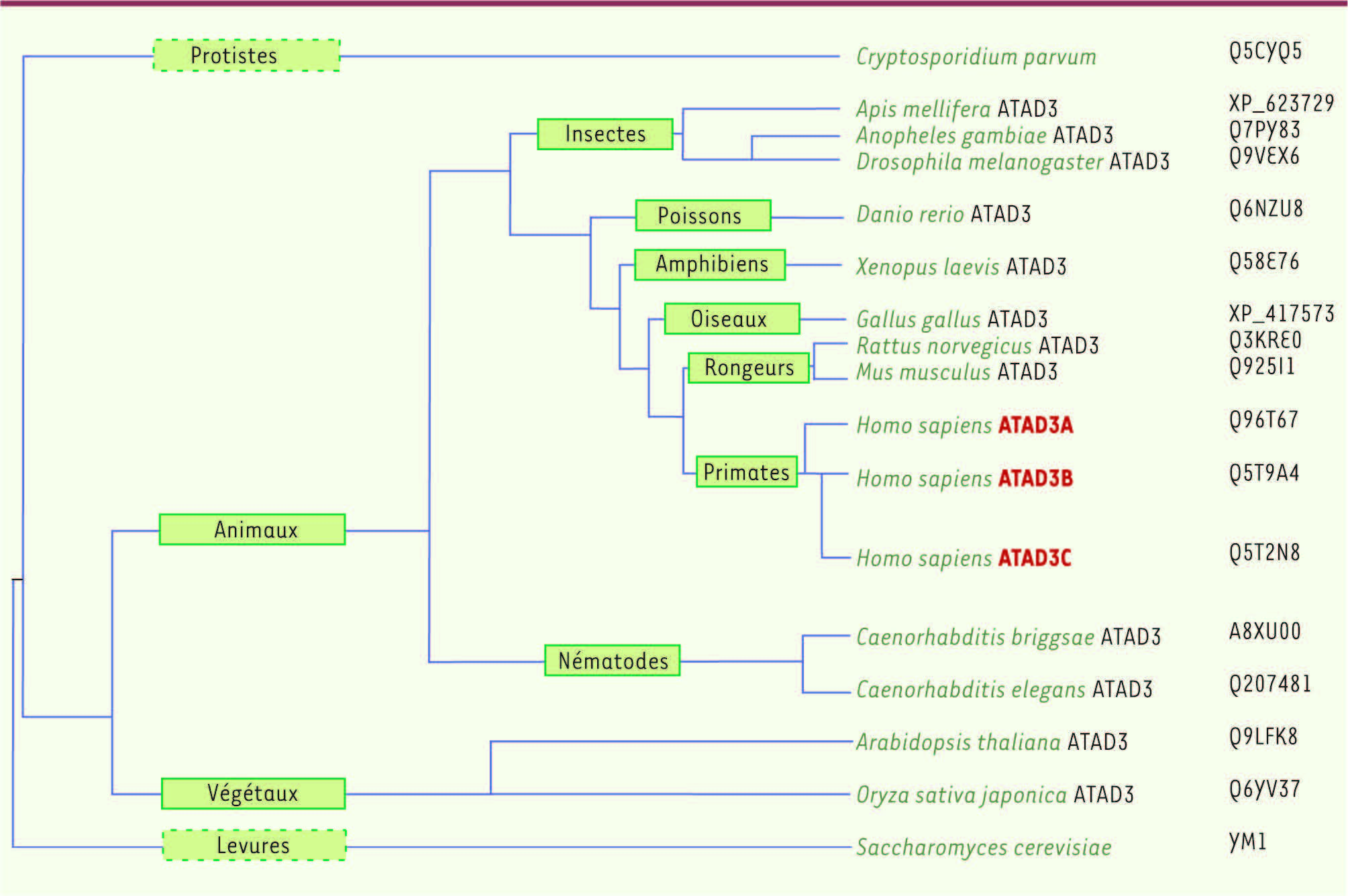
Aucun orthologue d’ATAD3 n’existe chez les organismes procaryotes ni chez la levure. Un orthologue très éloigné est présent chez les protistes et les levures, mais il s’agit d’une protéase dont le domaine caractéristique n’est pas présent dans ATAD3. Le gène ATAD3 apparaît réellement chez les organismes eucaryotes supérieurs : chez les plantes (Arabidopsis thaliana), les nématodes (Caenorhabditis elegans), les insectes (Drosophila melanogaster), les mammifères, le singe macaque et l’homme (Figure 1). Une seule copie de ce gène est présente dans les génomes haploïdes de ces organismes eucaryotes supérieurs, sauf chez les primates et l’homme où trois gènes différents existent. Chez les plantes, le gène ATAD3 est présent en trois exemplaires identiques par la simple conséquence de la polyploïdie. Chez les primates et l’homme, ATAD3 a été dupliqué deux fois (duplications contiguës sur le chromosome 1 et mutations). Ces duplications, tardives dans l’évolution des espèces, sont propres aux primates et aux hommes.
 | Figure 1.
Arbre phylogénétique simplifié d’ATAD3. Un homologue très éloigné est repéré chez les protozoaires et la levure mais il s’agit d’une ATPase ayant fonction de protéase. Ce domaine protéasique n’est pas présent dans les protéines ATAD3. (http://www.treefam.org/cgi-bin/ ou http://phylofacts.berkeley.edu/phyloscope/). |
En tant que forme ancestrale, l’un des gènes est nommé ATAD3A. Le deuxième gène est un paralogue très proche, nommé ATAD3B et le troisième gène, ATAD3C, présente une délétion de la séquence codante d’ATAD3A, produisant ainsi une partie amino-terminale raccourcie. Ces trois gènes se situent côte à côte à l’extrémité du bras court du chromosome 1 au locus 1p36.33. Le gène ATAD3B est très similaire à ATAD3A : le nombre d’exons est identique (16) et la longueur des introns ne varie que très peu. La partie codante engendre une séquence protéique dont l’identité stricte est de 93 %. ATAD3A et ATAD3B diffèrent essentiellement dans la structure de leur promoteur (voir plus loin) et au niveau du dernier exon : dans ATAD3B, le codon stop présent originellement dans ATAD3A a subi une mutation permettant le prolongement de la traduction. Il en résulte une extension carboxy-terminale de 62 acides aminés de la protéine, avec quelques sites potentiels de phosphorylation (http://www.phosphosite.org) et une séquence transmembranaire putative. Le troisième gène, ATAD3C, ne possède que 12 exons au lieu de 16 pour ATAD3A et ATAD3B. Les exons codant pour les 70 premiers acides aminés d’ATAD3A/B ne sont pas traduits, car le site d’initiation de la traduction est modifié et non utilisé, l’initiation ayant lieu en aval (+210). Pour le reste de sa structure, ATAD3C possède une identité de 87 % par rapport à ATAD3A. Enfin, les gènes codant ATAD3 semblent présenter des processus d’excision-épissage différentiels : les variants d’épissage des gènes ATAD3A et ATAD3B (ATAD3Al et ATAD3Bs) présentent deux exons supplémentaires codant pour une séquence à potentiel transmembranaire de type hélice alpha hydrophobe en amino-terminal (aa101-120 pour ATAD3Al) (Figure 2).
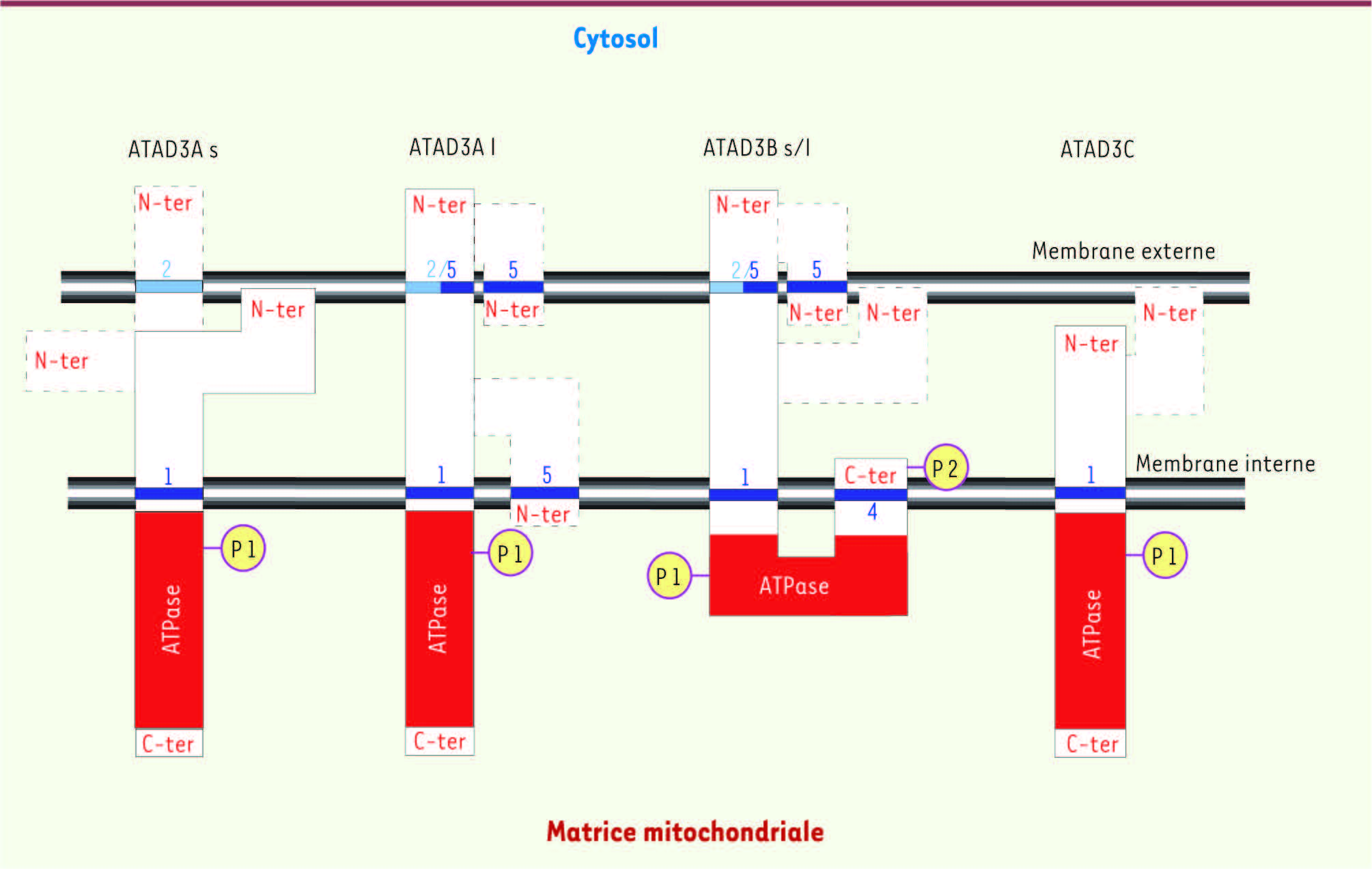
 | Figure 2.
Modèles d’insertion membranaire d’ATAD3 et de ses variants. Selon les données actuelles, la partie ATPasique d’ATAD3 se situe dans la matrice mitochondriale quelles que soient les isoformes concernées. Pour ATAD3As, qui pourrait posséder une, deux, voire trois hélices, la partie amino-terminale est soit dans le cytosol, soit dans l’espace intermembranaire, soit ancrée dans la membrane externe. Pour ATAD3Al, qui possède clairement une deuxième hélice transmembranaire (excision-épissage différentiel), la partie amino-terminale est soit dans le cytosol, soit dans l’espace intermembranaire, soit renvoyée dans la matrice. Pour ATAD3B, le cas est similaire à ATAD3As/l mais la partie carboxy-terminale devrait revenir dans l’espace intermembranaire. Enfin, pour ATAD3C, il est très probable que la partie amino-terminale soit dans l’espace intermembranaire ou ancrée dans la membrane externe. Les séquences transmembranaires en bleu foncé correspondent à des séquences identifiées par tous les moteurs de recherche (DAS, TOPpred, TMpred et HMMTOP, n° 1, 4 et 5). Les séquences transmembranaires en bleu clair correspondent à des séquences identifiées par un ou quelques moteurs de recherche (TMpred et HMMTOP, n° 2 et 3). Ces dernières zones d’hydrophobicité pourraient correspondre à des formes transitoires d’insertion membranaire. Les sites putatifs de phosphorylation par la PKC sont indiqués par P1 [
23] et le site de phosphorylation spécifique aux isoformes d’ATAD3Bs/l est représenté par P2. |
La séquence de la protéine ATAD3A est donc très conservée au cours de l’évolution : 39 % d’identité entre la protéine ATAD3A humaine et ses orthologues chez les plantes. ATAD3A a été détectée dans tous les modèles cellulaires et tous les tissus étudiés à ce jour. L’invalidation d’ATAD3 chez le nématode C. elegans est létale précocement, à un stade où la biosynthèse massive de mitochondries est nécessaire [4,
5]. De même, l’invalidation d’ATAD3 chez la drosophile inhibe le développement embryonnaire au stade prépupal [
6]. |
La séquence primaire d’ATAD3 permet de distinguer deux domaines fonctionnels majeurs correspondant aux deux moitiés, amino- et carboxy-terminales de la protéine. La présence ou l’absence de deux exons épissés pour ATAD3A et ATAD3B a amené à définir une nomenclature basée sur la taille respective des protéines à savoir s et l pour small et long
1. La partie carboxy-terminale de la protéine constitue le domaine ATPasique : on y retrouve les motifs clés impliqués dans la fixation et l’hydrolyse de l’ATP : sensor 1, sensor 2, doigt arginine (Arg), Walker A (WA) et Walker B (WB) [3]. La région amino-terminale constitue le domaine spécifique d’ATAD3. Il contient un, ou des, domaine(s) transmembranaire(s) (certains logiciels en trouvent jusqu’à trois,
Figure 2
[
8]), une région flexible riche en proline (a.a 18-27 de ATAD3As), et une région de type coiled coil (a.a. 80-220 de ATAD3As) possiblement impliquée dans l’oligomérisation des monomères d’ATAD3 et/ou l’interaction avec des partenaires [6]. On sait par ailleurs que les membres de la famille des AAA-ATPases dont la structure a été précisée forment des hexamères arrangés en anneaux [3]. D’après l’analyse in silico de la séquence primaire, aucune des isoformes d’ATAD3A/B/C ne possède de signal d’adressage mitochondrial connu de type extrémité amino-terminale clivable. L’adressage d’ATAD3 à la mitochondrie dépend d’une large séquence centrale comprenant la séquence transmembranaire majeure [6, 7]. Concernant les modifications post-traductionnelles prédictibles, il est possible de détecter plusieurs sites de phosphorylation putatifs pour des kinases. |
Localisation cellulaire et topologie membranaire La localisation mitochondriale d’ATAD3 a d’abord été mise en évidence par une approche d’exploration protéomique de la membrane interne de la mitochondrie [2]. Ce résultat a été confirmé par immunofluorescence dans des cellules Hela [
9]. Différentes approches de biochimie ont permis ensuite de montrer qu’ATAD3 est fortement ancrée dans la membrane interne de la mitochondrie [6, 7,
10]. Par ailleurs, ATAD3 a été trouvée dans de larges complexes multimoléculaires associés à l’ADN mitochondrial [
11, 10]. L’extrémité amino-terminale d’ATAD3 n’est toutefois pas située dans la matrice mitochondriale (Figure 2) [11–
13]. Nous avons effectivement montré que la partie amino-terminale d’ATAD3A est exposée à l’extérieur de la membrane interne, sa localisation précise dans le cytosol, l’espace intermembranaire ou la membrane externe étant encore incertaine. La partie carboxy-terminale est, quant à elle, bien située dans la matrice (Figure 2) [13]. ATAD3Al, ATAD3Bl et ATAD3C, qui contiennent une partie de ces 50 acides aminés amino-terminaux, pourraient aussi partager cette insertion membranaire, mais pas ATAD3s qui ne contient pas cette séquence. Le problème se complique légèrement si l’on considère les variants d’épissage d’ATAD3A et d’ATAD3B [8]. ATAD3Al et ATAD3Bs possèdent ainsi un domaine structural supplémentaire susceptible de former une hélice alpha transmembranaire. Ce segment pourrait permettre à la protéine d’accéder au cytosol, ou de retraverser la membrane interne. Il présente de plus des sites de phosphorylation (Figure 2). Enfin, les formes s et l de ATAD3B, contrairement à celles de ATAD3A, possèdent une séquence transmembranaire potentielle à l’extrémité carboxy-terminale qui pourrait contribuer à faire revenir la protéine dans l’espace intermembranaire (Figure 2). Cependant, on ne peut exclure que ces régions hydrophobes à potentiel transmembranaire jouent un rôle dans l’homopolymérisation d’ATAD3, dans l’interaction avec des partenaires ou dans les mécanismes d’insertion membranaire d’ATAD3, sans pour autant constituer in fine un domaine transmembranaire. Une meilleure connaissance de la topologie membranaire d’ATAD3 et de ses isoformes est aujourd’hui nécessaire à la poursuite d’autres investigations, en particulier la recherche de partenaires. La microscopie électronique semble être aujourd’hui une étape incontournable pour atteindre ce but. |
Expression d’ATAD3 chez l’homme Chez les primates et l’homme, la forme ancestrale du gène codant pour ATAD3, nommée ATAD3A, a été dupliquée deux fois et mutée pour engendrer deux paralogues nommés ATAD3B et ATAD3C. Ces trois gènes sont contigus à l’extrémité du chromosome 1 (locus 1p36.33). Comme nous l’avons vu, le gène ATAD3B est très similaire à ATAD3A (93 % d’homologie par rapport à ATAD3As), mais avec une extension de son extrémité carboxy-terminale (voir ci-dessus). La protéine ATAD3C (87 % d’homologie avec ATAD3As), ne possède pas les premiers 70 a.a., ce qui lui fait perdre la structure coiled coil ; elle diffère aussi dans la partie carboxy-terminale en lui conférant de nouveaux sites potentiels de phosphorylation. Ces trois protéines pourraient donc partager une même fonction tout en gardant des spécificités de fonctionnement (localisation, régulation, partenaires…). Une observation notable concerne les promoteurs de ces gènes (http://www.bimas.cit.nih.gov). Le promoteur putatif du gène ancestral, ATAD3A, contient des sites de fixation pour de nombreux éléments de régulation de la croissance cellulaire comme par exemple C/EBP (CCAAT/enhancer binding protein, 2 sites de fixation), CBF/IRP/CREB (core binding factor et iron regulatoty protein et cAMP response element binding protein, 2 sites), GATA-1 (3 sites), Oct-1 (octamer-binding protein, 1 site) et TFIID (1 site). Le promoteur d’ATAD3B contient ces mêmes sites (CREB, GATA-1, Oct-1 et TFIID), mais contient, en plus, 5 domaines de liaison à Pit1 (growth hormone factor 1), 2 domaines de liaison à Myc et un domaine de liaison à la myogénine. Le facteur transcriptionnel Pit1 est exprimé au cours de l’embryogenèse et dans l’hypophyse chez l’adulte [
14], et la myogénine est également exprimée au cours de l’embryogenèse [
15–
17]. En conséquence, ces deux facteurs pourraient contribuer à l’expression spécifique d’ATAD3Bl/s dans des tissus embryonnaires et en particulier dans les tissus musculaires. Nous avons montré que l’expression relative de ces deux gènes ATAD3A et ATAD3B varie significativement chez l’homme [8] : le premier est exprimé de manière ubiquitaire, et en particulier dans le cerveau ; le second n’est pas exprimé dans le cerveau adulte normal mais dans des tissus à fort potentiel prolifératif (embryon et zones germinatives adultes comme l’hypophyse adulte) [8–10]. Concernant l’expression d’ATAD3C, aucune donnée expérimentale n’existe actuellement. |
Plusieurs articles ont mis en évidence un lien entre la surexpression d’ATAD3As ou d’ATAD3Bl, la progression tumorale et une résistance accrue à l’apoptose (chimiorésistance) [8, 9,
18,
19]. La localisation chromosomique des gènes humains ATAD3A, ATAD3B et ATAD3C (1p36-33) a attiré notre attention, car dans cette région sont également localisés des gènes suppresseurs de tumeur et de chimiorésistance. Or les remaniements de cette région chromosomique permettent de discriminer deux types de tumeurs gliales : les astrocytomes et les oligodendrogliomes [
20]. Ces derniers présentent une délétion récurrente du bras court du chromosome 1 (1p), absente des astrocytomes. Les oligodendrogliomes étant des tumeurs de bon pronostic, c’est-à-dire peu agressives et chimiosensibles, contrairement aux astrocytomes, il devenait intéressant de voir si ATAD3A et ATAD3B pouvaient se comporter, dans des cellules de type oligodendroglial, comme des anti-oncogènes ou des gènes de chiomorésistance. Dans nos études [8], ATAD3Bl était indétectable dans des lignées dérivées d’oligodendrogliomes, alors qu’ATAD3As était légèrement sous exprimée par rapport à des lignées dérivées d’astrocytomes. Nous avons ensuite montré, en étudiant une lignée d’oligodendrogliome (HS683), qu’en plus de la large délétion 1p, le deuxième gène ATAD3B avait disparu par délétion, ne laissant, sur les quatre gènes ATAD3A/B, qu’un seul exemplaire d’ATAD3A. Ces délétions pourraient indiquer un processus de perte d’hétérozygotie d’un gène suppresseur de tumeur. Puisqu’ATAD3Bl est détectable dans des lignées cellulaires dérivées d’astrocytomes mais indétectable dans celles dérivées d’oligodendrogliomes, il est possible qu’elle puisse être un marqueur positif des astrocytomes par rapport aux oligodendrogliomes et du tissu sain bordant les résections chirurgicales, ou, inversement, un marqueur négatif des oligodendrogliomes. L’analyse de l’expression d’ATAD3Bl dans des biopsies de tumeurs gliales humaines pourrait valider cette hypothèse.
In vitro, l’expression ectopique d’ATAD3As et d’ATAD3Bl dans des cellules dérivées de gliomes induit une inhibition notable de la croissance cellulaire en allongeant la phase G1 du cycle cellulaire (Tableau I) [8]. ATAD3 pourrait donc avoir des propriétés d’anti-oncogène dans ces lignées cellulaires. À l’inverse, le traitement par des siARN dirigés contre ATAD3 des cellules de la lignée U373 dérivée d’un astrocytome, stimule leur prolifération. Cette observation ne peut cependant pas être généralisée à tous les types cellulaires : en effet, ces siARN induisent une apoptose associée à un dysfonctionnement de la cytokinèse et une endoréplication dans les cellules GHD-1 (carcinome de l’hypopharynx) [9], et la surexpression d’ATAD3As dans les cellules HeLa a un effet proapoptotique [2]. Il est possible que selon le contexte cellulaire, dans des lignées tumorales différentes, l’expression d’ATAD3 puisse être soit facultative soit indispensable. Dans les cellules HeLa et GHD-1, la surexpression d’ATAD3Bs/l n’induit pas de ralentissement notable du cycle cellulaire mais ici la surexpression relative d’ATAD3 n’a pas été mesurée [9].
Tableau I.
| Modèles cellulaires |
Techniques Isoformes |
Effets |
Références |
|
COS
|
Surexpression transitoire |
↘ Prolifération |
[8] |
| Cellule de rein-SV40/singe |
ATAD3As/Bl |
|
|
HS683
|
Surexpression transitoire/stable |
↘ Prolifération |
[8] |
| Oligodendrogliome/homme |
ATAD3As/Bl |
↘ Apoptose |
|
|
U373
|
Surexpression transitoire/stable |
↘ Prolifération |
[8] |
| Astrocytome/homme |
ATAD3As/Bl |
|
|
U373
|
SiARN |
↗ Prolifération |
[8] |
| Astrocytome/homme |
ATAD3As/Bl |
↗ Apoptose |
|
|
GHD-1
|
SiARN |
↗ Apoptose |
[9] |
| Carcinome hypopharynx/homme |
ATAD3Bs/Bl |
|
|
GHD-1
|
Surexpression transitoire |
Pas d’effet |
[9] |
| Carcinome hypopharynx/homme |
ATAD3Bs/BI |
|
|
HeLa
|
Surexpression transitoire |
↗ Apoptose |
[2] |
| Cancer cervical/homme |
ATAD3As |
|
|
A549
|
SiARN |
↘ Chimiorésistance |
[18] |
| Cancer poumon/homme |
ATAD3As |
|
|
H838
|
SiARN |
↘ Chimiorésistance |
[18] |
| Cancer poumon/homme |
ATAD3As |
|
|
U373
|
Surexpression |
↗ Chimiorésistance |
[8] |
| Astrocytome/homme |
ATAD3As/BI |
|
|
HS683
|
Surexpression |
↗ Chimiorésistance |
[8] |
| Oligodendrogliome/homme |
ATAD3As/BI |
|
|
RAJI
|
Protéomique différentielle |
↗ Radiorésistance |
[19] |
| Lymphome/homme |
ATAD3Bs/BI |
Tableau récapitulatif des effets de la sur- ou sous-expression d’ATAD3 sur la prolifération et la chimiorésistance pour différents modèles de lignées cancéreuses.
|
Nous avons également montré que l’expression ectopique d’ATAD3As et d’ATAD3Bl dans une lignée issue d’oligodendrogliome chimiosensible induit une résistance à court et à long terme envers différents agents génotoxiques [8] - peut-être liée à l’efficacité de l’arrêt du cycle cellulaire et de la réparation de l’ADN. À l’inverse, la diminution d’expression d’ATAD3As et 3Bl rend chimiosensible une lignée issue d’astrocytome chimiorésistante. Le mécanisme de cette résistance à court terme fait intervenir l’élimination rapide (perméabilité, modification chimique, dégradation) de l’agent génotoxique. En conséquence, en plus d’être un marqueur discriminant l’oligodendrogliome de l’astrocytome, ATAD3 pourrait être une cible thérapeutique pour optimiser le traitement chimiothérapeutique de l’astrocytome. Il a été par ailleurs montré récemment qu’ATAD3A constituait un marqueur antiapoptotique dans les tumeurs du poumon [18] et un marqueur de radiorésistance dans les lymphomes non hodgkiniens [
21]. |
Quelle fonction pour ATAD3 ? Aujourd’hui, les données concernant ATAD3 sont encore peu nombreuses et si sa localisation et sa topologie membranaire sont maintenant mieux établies, certains aspects restent contradictoires. ATAD3 a été initialement impliquée dans des réponses auto-immunes chez des patients cancéreux [
22, 23]. Ces résultats impliquent qu’ATAD3 puisse être accessible au système immunitaire et aux anticorps. Or, elle n’a été visualisée que dans les mitochondries même si sa topologie membranaire mitochondriale n’est pas définitivement élucidée (voir ci-dessus). On ne peut exclure sa relocalisation dans le compartiment extracellulaire à la suite d’un clivage ou d’un processus d’excision/épissage différentiel. Elle peut aussi être exposée par des cellules mortes ou endommagées par les traitements antitumoraux ce d’autant qu’elle est abondante et relativement stable. ATAD3A a été montrée comme pouvant s’associer à la frataxine [
24], qui intervient dans l’assemblage des noyaux fer-soufre essentiels au fonctionnement des complexes de la chaîne respiratoire, et dont les mutations sont impliquées dans l’ataxie de Friedreich, une maladie neurodégénérative. Pour terminer, nous insistons sur le rôle que pourrait avoir ATAD3 dans les interactions entre le réticulum et la mitochondrie et, dans cette dernière, entre membrane interne et membrane externe. En effet, ATAD3 semble être localisée aux points de contact où se juxtaposent la membrane externe et la membrane interne des mitochondries, et ces points de contact sont en rapport avec les zones de contact entre le réticulum endoplasmique et les mitochondries [6, 7, 18]. Ces zones de contact sont reconnues aujourd’hui pour être des lieux de transit de nombreuses molécules comme des protéines néosynthétisées, le calcium et des phospholipides tels que la phosphatidylsérine et le cholestérol. Ainsi, l’invalidation d’ATAD3, qui induit une fragmentation du réseau mitochondrial et une perte d’interaction entre les mitochondries et le réticulum, induit une réduction majeure des flux lipidiques de cholestérol et de phosphatidylsérine entre ces compartiments [5, 6, 7,
25]. En participant à la constitution de ces structures d’échange, ATAD3 pourrait donc, de manière plus générale, contribuer à l’homéostasie mitochondriale en participant à un mécanisme de recyclage/néosynthèse des constituants mitochondriaux et ceci en lien avec le réticulum endoplasmique [19,
26,
27]. Pour autant, nous ne sommes pas certains qu’il existe une causalité directe entre l’invalidation d’ATAD3 et la déstructuration du réseau mitochondrial. Ce processus pourrait être indirect et associé à un autre dysfonctionnement de la mitochondrie. La fonction précise de cette ATPase mitochondriale reste encore inconnue bien qu’elle se révèle essentielle au bon fonctionnement du réseau mitochondrial. Son abondance et son expression ubiquitaire reflètent certainement un rôle important dans la cellule. La recherche de ses partenaires est une étape essentielle - qui peut être rendue difficile par l’abondance cellulaire d’ATAD3, sa localisation membranaire, ainsi que son pHi très basique (pHi=10) -, de même que l’étude de modèles de différenciation, permettant l’analyse des effets cellulaires de son invalidation ou de sa surexpression. ‡ |
L’auteur déclare n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Nous sommes très reconnaissants au Dr Cécile Batandier pour son aide à la rédaction de ce manuscrit.
|
Footnotes |
1.
Zeller
KI
,
Jegga
AG
,
Aronow
BJ
, et al.
An integrated database of genes responsive to the Myc oncogenic transcription factor: identification of direct genomic targets . Genome Biol.
2003; ; 4 : :R69.. 2.
Da Cruz
S
,
Xenarios
I
,
Langridge
J
, et al.
Proteomic analysis of the mouse liver mitochondrial inner membrane . J Biol Chem.
2003; ; 278 : :41566.–41571. 3.
Frickey
T
,
Lupas
AN
. Phylogenitic analysis of AAA proteins . J Struct Biol.
2004; ; 146 : :2.–10. 4.
Kamath
RS
,
Ahringer
J
. Genome-wide RNAi screening in Caenorhabditis elegans . Methods.
2003; ; 30 : :313.–321. 5.
Hoffmann
M
,
Bellance
N
,
Rossignol
R
, et al.
C. elegans ATAD-3 is essential for mitochondrial activity and development . PLoS One.
2009; ; 4 : :e7644.. 6.
Gilquin
B
,
Taillebourg
E
,
Cherradi
N
, et al.
The AAA+ ATPase ATAD3A controls mitochondrial dynamics at the interface of the inner and outer membrane . Mol Cell Biol.
2010; ; 30 : :1984.–1996. 8.
Hubstenberger
A
,
Labourdette
G
,
Baudier
J
,
Rousseau
D
. ATAD3A and ATAD3B are distal 1p-located genes differentially expressed in human glioma cell lines and present in vitro anti-oncogenic and chemoresistant properties . Exp Cell Res.
2008; ; 314 : :2870.–2883. 9.
Schaffrik
M
,
Mack
B
,
Matthias
C
, et al.
Molecular characterization of the tumor-associated antigen AAA-TOB3 . Cell Mol Life Sci.
2006; ; 63 : :2162.–2174. 10.
He
J
,
Mao
CC
,
Reyes
A
, et al.
The AAA+ protein ATAD3 has displacement loop binding properties and is involved in mitochondrial nucleoid organization . J Cell Biol.
2007; ; 176 : :141.–146. 11.
Wang
Y
,
Bogenhagen
DF
. Human mitochondrial DNA nucleoids are linked to protein folding machinery and metabolic enzymes at the mitochondrial inner membrane . J Biol Chem.
2006; ; 281 : :25791.–25802. 12.
Bogenhagen
DF
,
Rousseau
D
,
Burke
S
. The layered structure of human mitochondrial DNA nucleoids . J Biol Chem.
2008; ; 283 : :3665.–3667. 13.
Hubstenberger
A
,
Merle
N
,
Charton
R
, et al.
Topological analysis of ATAD3A insertion in purified human mitochondria . J Bioenerg Biomembr.
2010; ; 42 : :143.–150. 14.
Simmons
DM
,
Voss
JW
,
Ingraham
HA
, et al.
Pituitary cell phenotypes involve cell-specific Pit-1 mRNA translation and synergistic interactions with other classes of transcription factors . Genes Dev.
1990; ; 4 : :695.–711. 15.
Sassoon
D
,
Lyons
G
,
Wright
WE
, et al.
Expression of two myogenic regulatory factors myogenin and MyoD1 during mouse embryogenesis . Nature.
1989; ; 341 : :303.–307. 16.
Hinterberger
TJ
,
Sassoon
DA
,
Rhodes
SJ
,
Konieczny
SF
. Expression of the muscle regulatory factor MRF4 during somite and skeletal myofiber development . Dev Biol.
1991; ; 147 : :144.–156. 17.
Pownall
ME
,
Gustafsson
MK
,
Emerson
CP
Jr
. Myogenic regulatory factors and the specification of muscle progenitors in vertebrate embryons . Annu Rev Cell Dev Biol.
2002; ; 18 : :747.–783. 18.
Fang
HY
,
Chang
CL
,
Hsu
SH
, et al.
ATPase family AAA domain-containing 3A is a novel anti-apoptotic factor in lung adenocarcinoma cells . J Cell Sci.
2010; ; 123 : :1171.–1180. 19.
Hayashi
T
,
Rizzuto
R
,
Hajnoczky
G
,
Su
TP
. MAM: more than just a housekeeper . Trends Cell Biol.
2009; ; 19 : :81.–88. 20.
Smith
JB
,
Alderete
B
,
Minn
Y
, et al.
Localization of common deletion regions on 1p and 19q in human glioma and their association with histological subtype . Oncogene.
1999; ; 18 : :4144.–4152. 21.
Jiang
Y
,
Liu
X
,
Fang
X
,
Wang
X
. Proteomic analysis of mitochondria in Raji cells following exposure to radiation: implications for radiotherapy response . Protein Pept Lett.
2009; ; 16 : :1350.–1359. 22.
Gires
O
,
Münz
M
,
Schaffrik
M
, et al.
Profile identification of disease-asociated humoral antigens using Amida, a novel proteomics-based technology . Cell Mol Life Sci.
2004; ; 61 : :1198.–1207. 23.
Geuijen
CA
,
Bijl
N
,
Smit
RC
, et al.
A proteomic approach to tumour target identification using phage display, affinity purification and mass spectrometry . Eur J Cancer.
2005; ; 41 : :178.–187. 24.
Correia
AR
,
Pastore
C
,
Adinolfi
S
, et al.
Dynamics, stability and iron-binding activity of frataxin clinical mutants . FEBS J.
2008; ; 275 : :3680.–3690. 25.
Gilquin
B
,
Cannon
BR
,
Hubstenberger
A
, et al.
The calcium-dependent interaction between S100B and the mitochondrial AAA-ATPase ATAD3A and the role of this complex in the cytoplasmic processing of ATAD3A . Mol Cell Biol.
2010; ; 30 : :2724.–2736. 26.
Kornmann
B
. Le complexe Ermes : une connexion haut-débit entre le réticulum endoplasmique et les mitochondries . Med Sci (Paris).
2010; ; 26 : :145.–146. 27.
Kornmann
B
,
Walter
P
. ERMES-mediated ER-mitochondria contacts: molecular hubs for the regulation of mitochondrial biology . J Cell Sci.
2010; ; 123 : :1389.–1393. |