| |
| Med Sci (Paris). 2011 June; 27(6-7): 569–571. Published online 2011 July 1. doi: 10.1051/medsci/2011276002.« Se manger soi-même » pour survivre Régulation coordonnée de l’autophagie par les nutriments Benoit Viollet1,2* and Marc Foretz1,2 1Inserm, U1016, Institut Cochin, 24, rue du Faubourg Saint-Jacques, 75014Paris, France 2Université Paris Descartes, Paris, France MeSH keywords: Adenylate kinase, physiologie, Animaux, Autophagie, Survie cellulaire, Cellules cultivées, Aliments, Hépatocytes, métabolisme, Homéostasie, Protéines et peptides de signalisation intracellulaire, Mitochondries du foie, Phosphorylation, Protein kinases, Maturation post-traductionnelle des protéines, Saccharomyces cerevisiae, cytologie, Protéines de Saccharomyces cerevisiae, Transduction du signal, Facteurs de transcription |
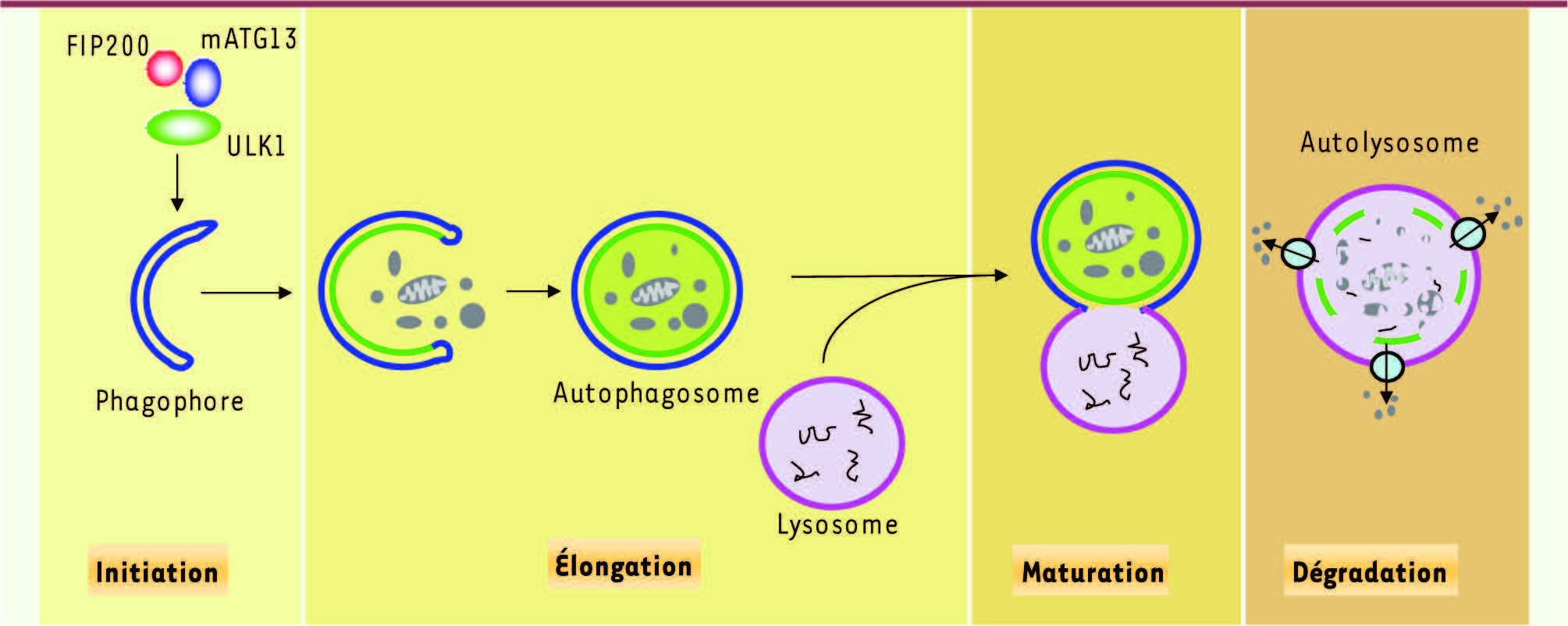
Autophagie : se manger soi-même pour survivre L’autophagie est un processus cellulaire catabolique hautement conservé au cours de l’évolution permettant de maintenir l’homéostasie cellulaire par la dégradation et le recyclage d’organites superflus ou endommagés ainsi que des protéines agrégées ou mal conformées. Des portions du cytoplasme sont alors séquestrées dans des vésicules appelées autophagosomes qui sont dégradées après fusion avec les lysosomes (Figure 1). L’autophagie est un mécanisme hautement inductible et finement régulé qui joue un rôle important dans de nombreux processus physiologiques et pathologiques comme le cancer, les myopathies, la dégénérescence neuronale, la réponse immune et le vieillissement [
1,
12,
13]. L’autophagie permet aux cellules ainsi qu’à l’organisme entier de résister à des situations de stress comme un apport limité en nutriments, en énergie ou en oxygène. Cependant, les mécanismes moléculaires impliqués dans l’induction du processus autophagique en réponse à ces différents stimulus et la coordination des différentes voies de signalisation concourant à sa régulation sont encore mal compris.
 | Figure 1
Schéma du processus d’autophagie. L’autophagie est initiée par la formation du phagosome par le complexe ULK1/mATG13/FIP200 et se poursuit par une série d’étapes comprenant l’élongation du phagosome, la formation de l’autophagosome, la maturation de l’autophagosome par fusion avec le lysosome et la dégradation de l’autolysosome et le recyclage des macromolécules hydrolysées par les enzymes lysosomales à l’aide de perméases (d’après [ 1]). |
|
Les acteurs dans l’induction de l’autophagie Initialement, des études génétiques chez la levure Saccharomyces cerevisiae ont permis d’identifier les principaux acteurs moléculaires des différentes étapes de l’autophagie [1]. En particulier, la protéine kinase ATG1 (autophagy-related genes 1), identifiée par un criblage génétique chez la levure, est un acteur essentiel dans l’induction de l’autophagie. ATG1 forme un complexe avec les protéines ATG13 et ATG17 en réponse à un signal de carence nutritionnelle. Cette induction de l’autophagie est rendue possible grâce à une inhibition de la voie de signalisation TOR (target of rapamycin), la déphosphorylation de ATG13, une interaction accrue entre ATG13 et ATG1 et une augmentation de l’activité kinase de ATG1 et de la phosphorylation de ses cibles. Les cellules de mammifères possèdent deux homologues pour la protéine ATG1, ULK1 (Unc-51-like kinase 1) et ULK2. Alors qu’ULK1 joue un rôle essentiel dans la régulation de l’autophagie, la fonction d’ULK2 n’a pas été clairement définie. Les homologues chez les mammifères des partenaires d’interaction ATG13 et ATG17 sont les protéines mATG13 et FIP200 (focal adhesion kinase family interacting protein of 200 kD). Dans ce complexe, mATG13 permet l’interaction entre ULK1 et FIP200 et est nécessaire à la phosphorylation de FIP200 par ULK1 en condition de carence nutritionnelle. Cependant, les mécanismes moléculaires du contrôle d’ULK1 par les nutriments restent encore largement méconnus. Plusieurs voies de signalisation impliquées dans la détection des niveaux nutritionnels et la gestion des stocks énergétiques ont récemment été identifiées mais leurs interactions dans le contrôle de l’autophagie restent encore mal connues. Une série de publications récentes [
2-
4] a permis d’établir une régulation coordonnée de l’initiation de l’autophagie par phosphorylation d’ULK1 en fonction de la disponibilité en nutriments par les voies de signalisation mTORC1 (mammalian target of rapamycin complex 1) [
14] et AMPK (AMP-activated protein kinase). |
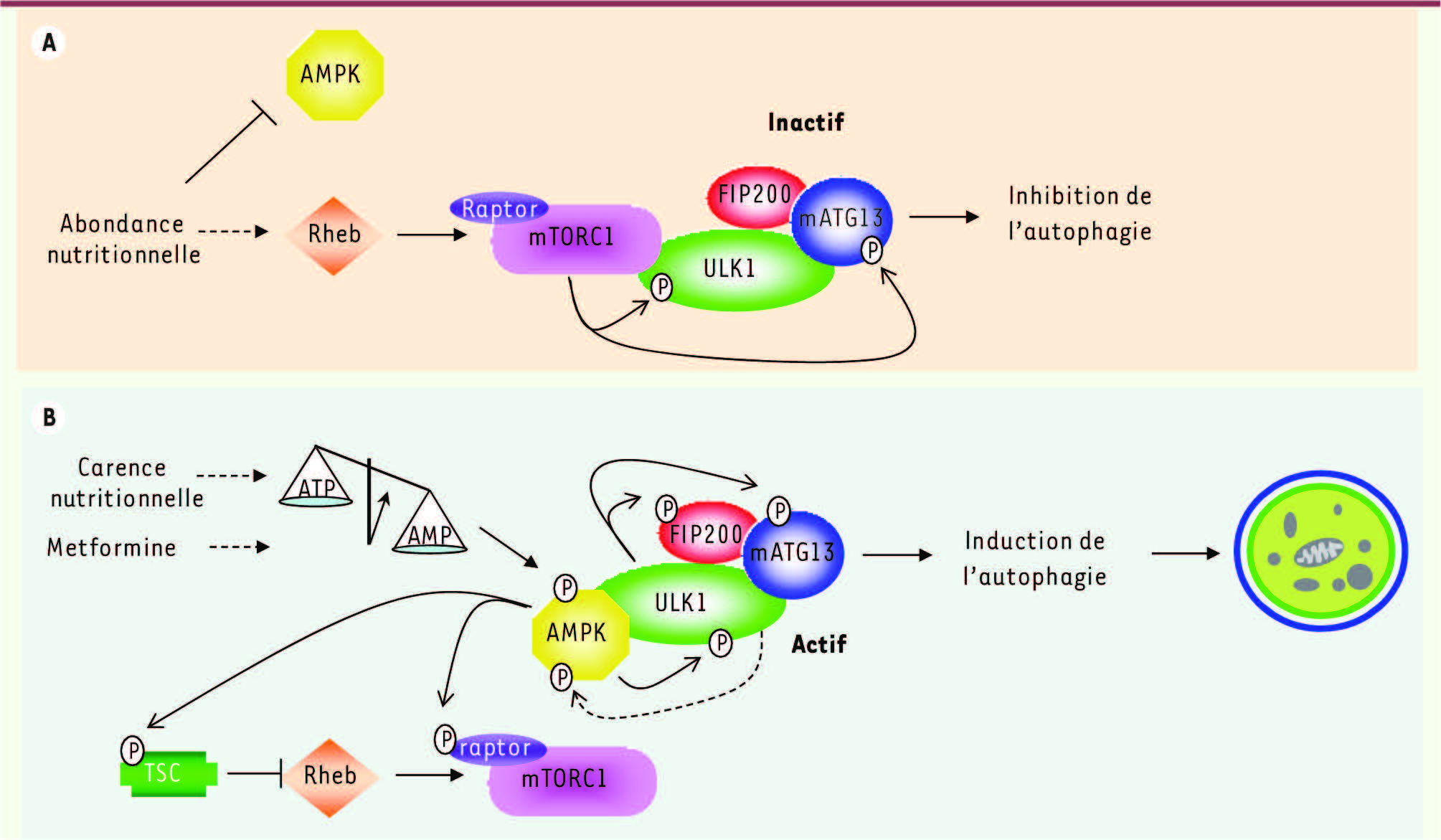
Phosphorylation coordonnée de ULK1 par mTORC1 et AMPK En présence de quantités suffisantes de nutriments, l’autophagie est maintenue à un état basal avec une fonction principalement de ménage (dégradation des protéines à durée de vie longue et renouvellement des organites endommagés). Dans cette situation, la voie de signalisation mTORC1 est fortement activée et bloque l’autophagie par la phosphorylation et l’inactivation des protéines ULK1 et ATG13 [
5,
6] (Figure 2A). À l’inverse, en situation de carence nutritionnelle, mTORC1 est inhibée. Cette inhibition permet alors l’activation d’ULK1, la phosphorylation d’ATG13 et FIP200 et l’induction du processus d’autophagie qui est nécessaire pour la survie cellulaire (Figure 2B). Cette induction de l’autophagie peut être reproduite en présence de rapamycine, inhibiteur de mTORC1, même en présence de nutriments. Le complexe mTORC1, composé de mTOR, GβL, PRAS40 et raptor, est inhibé en absence de nutriments par l’activation de l’AMPK (Figure 2). L’AMPK est un senseur énergétique intracellulaire impliqué dans la régulation de l’homéostasie énergétique en réponse aux variations nutritionnelles [
7]. L’AMPK est une cible indirecte de la metformine, un antidiabétique oral utilisé en première intention dans le traitement du diabète de type 2 [7]. Plusieurs études ont permis de relier l’induction de l’autophagie à l’activation de l’AMPK en réponse à un stress énergétique par sa capacité à inhiber la voie de signalisation mTORC1 [4,
8]. En effet, l’AMPK phosphoryle le suppresseur de tumeur TSC2 (tuberous sclerosis complex 2), aboutissant à l’inactivation de Rheb (Ras homolog enriched in brain), qui interagit et active mTORC1 [
9]. De plus, l’AMPK phosphoryle raptor, induisant sa liaison à la protéine 14-3-3 et l’inhibition de mTORC1 [
10]. Cependant, le contrôle de l’autophagie par l’AMPK pourrait également s’effectuer directement au niveau du complexe ULK1/mATG13/FIP200. Il a été montré que l’AMPK interagit directement avec ULK1 et que cette interaction est nécessaire pour l’induction de l’autophagie [2-4]. La liaison entre ULK1 et AMPK est augmentée en présence de rapamycine, suggérant que mTORC1 contrôle négativement cette interaction [
3]. En effet, la région d’interaction de ULK1 avec l’AMPK contient un site de phosphorylation par mTORC1 qui empêche l’interaction entre ces deux partenaires [3]. La phosphorylation d’ULK1 sur ce site est fortement corrélée avec l’augmentation de l’activité mTORC1. Non seulement l’AMPK interagit avec ULK1, mais elle induit également sa phosphorylation sur de multiples résidus, modifications nécessaires à son activation en réponse à un stress énergétique ainsi qu’à des activateurs de l’AMPK comme la metformine et l’AICAR [2, 3]. Ainsi, la phosphorylation d’ULK1 au niveau des résidus cibles de l’AMPK et de mTORC1 est régulée de manière opposée reflétant les conditions où les activités AMPK et mTORC1 sont contraires (carence versus abondance de nutriments).
 | Figure 2
Régulation coordonnée de la phosphorylation de ULK1 par mTORC1 et AMPK. A. En présence de nutriments, l’inhibition de l’AMPK et la phosphorylation de ULK1 et mATG13 par mTORC1 bloquent l’autophagie. B. En situation de carence nutritionnelle, le complexe mTORC1 est inhibé par l’activation de l’AMPK, ce qui permet l’interaction de ULK1 avec l’AMPK, la phosphorylation et l’activation de ULK1, la phosphorylation des protéines mATG13 et FIP200 et l’induction de l’autophagie. Par une boucle directe de rétrocontrôle négatif, l’activation de ULK1 provoque la phosphorylation de l’AMPK et la diminution de son activité, ce qui module l’induction de l’autophagie. |
|
ULK1, AMPK, mitophagie et survie cellulaire L’analyse d’hépatocytes déficients en ULK1 ou AMPK en culture primaire a révélé d’importants défauts dans l’élimination sélective des mitochondries par autophagie (processus défini par le terme de mitophagie) [2]. Une accumulation anormale de mitochondries a été détectée par une analyse en microscopie électronique à transmission dans ces cellules, parallèlement à une augmentation de la protéine p62 impliquée dans la clairance des mitochondries. L’expression d’un mutant non phosphorylable d’ULK1 (au niveau des résidus cibles de l’AMPK) dans des fibroblastes embryonnaires invalidés pour les protéines ULK1 et ULK2 ne permet pas de complémenter, en réponse à une carence nutritionnelle, les défauts du nombre anormal et de la morphologie aberrante des mitochondries, contrairement à l’expression d’une protéine ULK1 sauvage. En absence de nutriments, les cellules déficientes en ULK1 et ULK2 ont une viabilité cellulaire réduite en raison de leur incapacité à induire l’autophagie. Leur survie cellulaire peut être alors restaurée par complémentation avec une protéine ULK1 sauvage mais non avec un mutant non phosphorylable d’ULK1 au niveau des résidus cibles de l’AMPK [2]. Ces résultats suggèrent que la phosphorylation d’ULK1 par l’AMPK est essentielle et suffisante pour assurer la fonction d’ULK1 dans l’induction de l’autophagie en réponse à un stress nutritionnel. |
Ces travaux dévoilent les mécanismes moléculaires concourant à la régulation de l’induction de l’autophagie en réponse aux variations de la disponibilité en nutriments (Figure 2). En présence de quantités suffisantes de nutriments, mTORC1 est activée et phosphoryle ULK1 pour bloquer l’interaction entre ULK1 et son partenaire AMPK, laissant ULK1 inactif. En situation de carence en nutriments, l’AMPK est activée et inhibe mTORC1 en phosphorylant TSC2 et raptor. ULK1, libérée de son interaction et de sa phosphorylation par mTORC1, peut alors interagir avec l’AMPK qui la phosphoryle et permet son activation. ULK1 augmente alors son autophosphorylation et phosphoryle mATG13 et FIP200 pour déclencher le processus d’autophagie. Cette phosphorylation coordonnée d’ULK1 par l’AMPK et mTORC1 permet d’expliquer comment les cellules peuvent mettre en place un programme de recyclage cellulaire finement régulé et préserver leur homéostasie en réponse à différentes situations de stress. Cependant, un excès d’autophagie peut être néfaste pour la cellule, il est donc important de déterminer par quel(s) mécanisme(s) de rétrocontrôle l’AMPK pourrait être inhibée permettant ainsi de contrôler efficacement l’arrêt de l’autophagie. Il a été récemment démontré qu’une fois activée, ULK1 est capable de phosphoryler l’AMPK conduisant à la diminution de son activité [
11]. Ainsi, ULK1 constitue un point de convergence non seulement dans l’induction de l’autophagie mais également dans l’atténuation des signaux qui déclenchent son activation (Figure 2). Conflit d’interets
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
1.
Yang
Z
,
Klionsky
DJ
. Eaten alive: a history of macroautophagy . Nat Cell Biol.
2010; ; 12 : :814.-822. 2.
Egan
DF
,
Shackelford
DB
,
Mihaylova
MM
, et al.
Phosphorylation of ULK1 (hATG1) by AMP-activated protein kinase connects energy sensing to mitophagy . Science.
2010; ; 331 : :456.-461. 3.
Kim
J
,
Kundu
M
,
Viollet
B
, et al.
AMPK and mTOR regulate autophagy through direct phosphorylation of Ulk1 . Nat Cell Biol.
2010; ; 13 : :132.-141. 4.
Lee
JW
,
Park
S
,
Takahashi
Y
, , et al.
The association of AMPK with ULK1 regulates autophagy.
PLoS One.
2010; ; 5 : :e15394.. 5.
Jung
CH
,
Jun
CB
,
Ro
SH
, et al.
ULK-Atg13-FIP200 complexes mediate mTOR signaling to the autophagy machinery . Mol Biol Cell.
2009; ; 20 : :1992.-2003. 6.
Hosokawa
N
,
Hara
T
,
Kaizuka
T
, et al.
Nutrient-dependent mTORC1 association with the ULK1-Atg13-FIP200 complex required for autophagy . Mol Biol Cell.
2009; ; 20 : :1981.-1991. 7.
Foretz
M
,
Taleux
N
,
Guigas
B
, et al.
Régulation du métabolisme énergétique par l’AMPK : une nouvelle voie thérapeutique pour le traitement des maladies métaboliques et cardiaques . Med Sci (Paris).
2006; ; 22 : :381.-388. 8.
Herrero-Martin
G
,
Hoyer-Hansen
M
,
Garcia-Garcia
C
, et al.
TAK1 activates AMPK-dependent cytoprotective autophagy in TRAIL-treated epithelial cells . Embo J.
2009; ; 28 : :677.-685. 9.
Inoki
K
,
Zhu
T
,
Guan
KL
. TSC2 mediates cellular energy response to control cell growth and survival . Cell.
2003; ; 115 : :577.-590. 10.
Gwinn
DM
,
Shackelford
DB
,
Egan
DF
, et al.
AMPK phosphorylation of raptor mediates a metabolic checkpoint . Mol Cell.
2008; ; 30 : :214.-226. 11.
Loffler
AS
. Ulk1-mediated phosphorylation of AMPK constitutes a negative regulatory feedback loop . 2011; ; 7 : sous presse.. 12.
Puyal
J
,
Ginet
V
,
Vaslin
A
,
Clarke
PG
. L’autophagie remplaçant de luxe du protéasome . Med Sci (Paris).
2008; ; 24 : :19.-21. 13.
Joubert
PE
,
Pombo Grégoire
I
,
Meiffren
G
, et al.
Autophagie et pathogènes : « Bon appétit Messieurs ! » . Med Sci (Paris).
2011; ; 27 : :41.-47. 14.
Julien
LA
,
Roux
PP
. mTOR, la cible fonctionnelle de la rapamycine . Med Sci (Paris).
2010; ; 26 : :1056.-1060. |