| |
| Med Sci (Paris). 2011 May; 27(5): 553–558. Published online 2011 May 25. doi: 10.1051/medsci/2011275023.Chroniques génomiques Le véritable « génome personnel » ? Bertrand Jordan1* 1Marseille-Nice Génopole, case 901, Parc scientifique de Luminy, 13288 Marseille Cedex 9, France |
Détecter les porteurs, une approche qui a des précédents Il s’agit en fait d’étendre un concept qui a déjà fait ses preuves à petite échelle dans quelques cas particuliers, le plus frappant étant la lutte contre la maladie de Tay-Sachs au sein de la communauté juive Ashkenaze. Cette gravissime affection neurologique, qui entraîne la mort en bas âge des enfants atteints, a vu son incidence réduite de plus de 90 % grâce à la détection des hétérozygotes et à l’organisation d’une sorte de « bureau central » au sein de cette communauté très soudée. Celui-ci répercute l’information en cas de mariage envisagé, ce qui aboutit, soit à la non réalisation de l’union projetée, soit à un diagnostic prénatal ou, mieux, préimplantatoire. Résultat très positif en termes de quasi-élimination d’une maladie gravissime [
4], mais dont la mise en œuvre évoque immanquablement l’eugénisme. Le dépistage de l’anémie falciforme (sickle cell disease) (Figure 1) a mis en évidence les dérives auxquelles peuvent donner lieu de telles analyses : les porteurs hétérozygotes - qui se trouvent, aux États-Unis, être majoritairement des Noirs - ont fait dans les années 1970 l’objet de discriminations sans aucune justification médicale [
5].
 | Figure 1.
Timbre recommandant le diagnostic de l’anémie falciforme.
United States postal service, 2004. |
L’article qui fait l’objet de cette chronique [
6] montre comment cette approche pourrait être étendue au diagnostic « préconceptionnel » de plusieurs centaines de maladies récessives pour lesquelles le gène concerné et les mutations pathogènes sont connus. Il émane principalement du National center for genome resources (NCGR) à Santa Fe (États-Unis) et comporte une analyse très détaillée de ce qui est nécessaire pour la mise en place d’un tel test, au niveau du laboratoire mais aussi de la bioinformatique et de la logistique. Chemin faisant, on rencontre quelques surprises et notamment le fait que près de 30 % des mutations indiquées dans les bases de données sont fausses - mais la conclusion finale est qu’un test portant sur près de 500 affections est dès maintenant réalisable à un coût très accessible : moins de 500 dollars américains tout compris, soit moins d’un dollar par échantillon et par maladie. |
Les paramètres de l’épure Selon les auteurs, on connaît à l’heure actuelle près de 1 200 affections récessives monogéniques pour lesquelles le gène en cause est identifié. Pour la mise au point du test, le choix a été fait de privilégier les affections très graves, fortement pénétrantes et dont les effets apparaissent dès l’enfance. Les mutations induisant une susceptibilité au cancer ont donc été écartées, tous comme les gènes impliqués dans divers types de retard mental (qui seront inclus dans un deuxième temps). On aboutit ainsi à une liste de 448 maladies correspondant à une large palettes de pathologies, les plus représentées étant neurologiques (122), métaboliques (142), développementales (46) et cutanées (45). Au niveau technique, les auteurs ont d’abord envisagé l’emploi de puces à ADN, mais ont dû l’écarter en raison du nombre élevé de mutations à détecter pour la plupart des affections, de la difficulté de détecter des altérations non ponctuelles par cette approche et (rétrospectivement) de la mauvaise qualité des données disponibles sur les mutations pathogènes (voir plus loin). Ils ont donc choisi une approche fondée sur le séquençage, qui présente l’avantage supplémentaire de pouvoir inclure facilement des nouvelles découvertes. Mais ils ont reculé devant la modalité la plus simple conceptuellement, celle d’un séquençage intégral de chaque ADN ou même de chaque exome global, en raison de son coût qui est encore de plusieurs milliers de dollars, d’autant plus que l’on exige ici une séquence de très bonne qualité puisqu’il s’agit de détecter des mutations. Ils ont donc choisi de purifier l’ADN correspondant aux régions à étudier afin de séquencer ensuite une « cible » relativement limitée. Sans rentrer dans le détail de cet article très complet (il comporte 14 pages plus les suppléments) qui décrit les différentes options et les tests qui ont été effectués, disons que la technique retenue est celle de la capture en solution mise au point par Agilent (et qui semble être devenue dominante à l’heure actuelle dans ce marché). Celle-ci repose sur une purification de la fraction d’ADN choisie grâce à une hybridation en solution avec un jeu ad hoc d’oligonucléotides. Dans le cas présent, la cible est constituée par l’ensemble des exons des gènes choisis, plus leurs jonctions d’épissage ainsi que les séquences introniques et adjacentes dans lesquelles des mutations ont déjà été repérées : ce qui représente au total près de deux millions de nucléotides. Le facteur d’enrichissement obtenu par rapport à l’ADN total est de 300 à 500 fois, et le séquençage (là aussi différentes options ont été testées) se fait sur une machine Illumina HiSeq (la plus performante de celles que commercialise cette entreprise), en mélangeant dans chacune des huit pistes de la cellule de séquençage douze échantillons (repérés par des amorces différentes). En un run de la machine on séquence ainsi 96 ADN ! Pour chacun d’eux, la couverture moyenne (nombre de lectures par nucléotide) est de 160, ce qui permet d’atteindre la fiabilité requise pour une application en diagnostic. L’évaluation des résultats - tant au niveau de la qualité de la séquence obtenue que de son interprétation à la lumière de l’ensemble des données disponibles - fait appel à un arbre de décision complexe incorporant l’accès à de nombreuses bases de données et une bonne dose de bioinformatique. |
Les résultats : des chiffres encourageants… Les chiffres obtenus à l’arrivée, après vérification par la méthode classique de Sanger sur une centaine d’échantillons, paraissent excellents : la présence de mutations (substitutions, insertions et délétions) est détectée avec une sensibilité de 95 % (seulement 5 % de faux négatifs) et une spécificité de 100 % (aucun faux positif). La technique mise au point s’avère donc fiable et semble largement préférable aux tests individuels pratiqués jusqu’ici et dont le coût unitaire (compte tenu de la nécessité de détecter différentes mutations) dépasse souvent le millier de dollars (Tableau I).
Tableau I.
| Maladie |
Prix public |
Remb. Medicare |
| FRAX1 |
272 $ |
31 $ |
| CFTR (43) |
3 522 $ |
1 720 $ |
| Panel Ashkénaze |
1 104 $ |
440 $ |
| SMA |
285 $ |
87 $ |
Prix public (aux États-Unis) et remboursement par le système public Medicare pour quelques tests génétiques. Le test CFTR (mucoviscidose) recherche les 43 mutations les plus fréquentes, le « panel Ashkénaze » examine un total de 22 mutations correspondant à sept maladies (dont celle de Tay-Sachs). FRAX1 : fragile X ; SMA : spinal muscular atrophy.
|
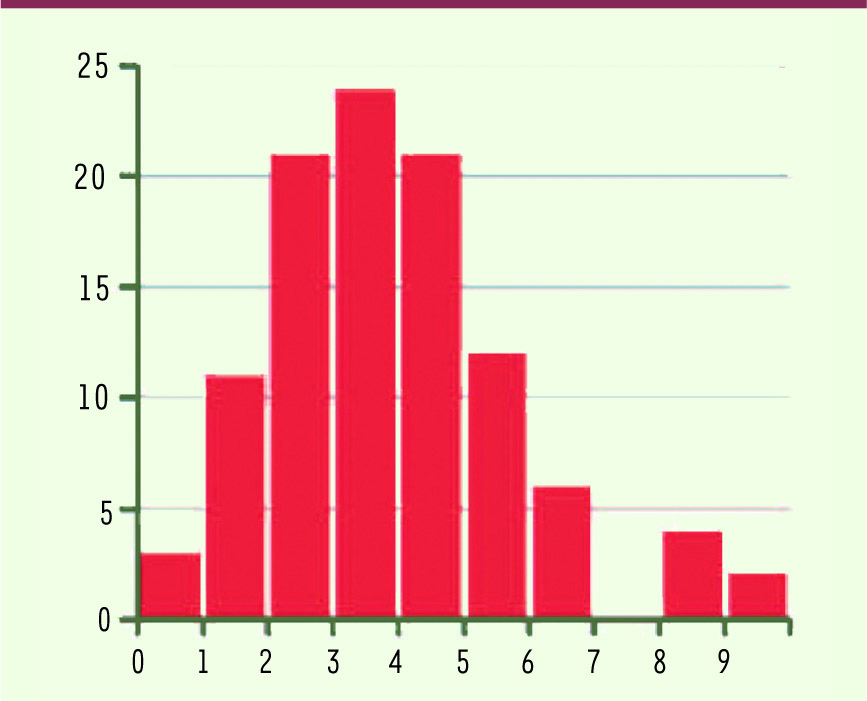
Une mauvaise surprise… Comme déjà mentionné, ces résultats comportent une surprise de taille : les mutations pathogènes répertoriées dans les bases de données sont erronées dans environ 27 % des cas ! L’erreur la plus fréquente consiste en ce que la prétendue mutation pathogène s’avère en fait être un polymorphisme courant, et ceci est facilement détecté dès lors que l’on séquence un certain nombre d’individus. Une mutation responsable (à l’état homozygote) d’une pathologie grave doit obligatoirement être très rare dans la population, or certaines d’entre elles s’avèrent avoir des fréquences supérieures à 5 %. De plus on découvre, pour quelques-unes, des individus homozygotes qui devraient donc être affectés - alors qu’ils sont indemnes. On est donc obligé de conclure que les répertoires officiels fourmillent d’erreurs. Ce n’est pas totalement inattendu dans la mesure où ils rassemblent des données hétéroclites obtenues par de multiples équipes au cours de plusieurs décennies, mais la gravité du problème est surprenante. Cela constitue évidemment un obstacle majeur pour le type de diagnostic multiplex que propose cet article : on ne peut plus se fier aux bases de données existantes pour décider si une variation vue dans l’ADN d’une personne est inoffensive ou pathogène. Il va falloir reconstruire une base de données réellement valable ( authoritative) grâce au séquençage de nombreux échantillons provenant de personnes affectées ou indemnes, gérer ces informations de manière rigoureuse et, idéalement, les mettre à la disposition de la communauté scientifique et médicale. Tout cela est plus facile à dire qu’à faire, mais un consortium de laboratoires publics et privés a déjà commencé à s’y attaquer [
7]. Quelques valeurs utiles Une fois les résultats débarrassés de ces erreurs, on peut noter quelques conclusions générales utiles. Tout d’abord, le nombre de mutations délétères trouvées à l’état hétérozygote est en moyenne d’environ trois par personne, avec une distribution qui va pour l’essentiel de un à six (Figure 2). Notons qu’il ne s’agit pas de la « charge » totale en mutations pathogènes puisque l’on n’a testé « que » 448 affections, mais vu le choix effectué cela doit néanmoins en constituer la majorité. Par ailleurs, les auteurs notent que le nombre total de ces mutations n’est associé ni au sexe, ni à l’ethnicité (Caucasiens ou autres), ce qui montre l’inutilité d’un dépistage ciblé sur certaines populations, contrairement à ce qu’affirment certains partisans d’une médecine ethnique. Comme mentionné dans une chronique récente [
8] à propos du nombre de nouvelles mutations potentiellement pathogènes apparaissant chez chaque nouveau-né (une en moyenne), ces chiffres fondés sur des données réelles (et non sur des estimations plus ou moins contestables) sont fort précieux.
 | Figure 2
Nombre de mutations délétères par personne (abscisse), le nombre d’échantillons est en ordonnée (extrait adapté de la figure 7 de [ 6]). |
|
Et quid des aspects éthiques ? Restent à aborder les questions éthiques que soulèvent ces nouvelles possibilités techniques déjà opérationnelles. De fait, plusieurs tests fondés sur le séquençage et recherchant jusqu’à une centaine de mutations sont actuellement commercialisés aux États-Unis1,, et bientôt en Europe par l’entreprise espagnole Sistemas Genomicos
2. Il s’agit en général d’analyses liées au risque de diverses affections cardiaques, mais le principe est le même et l’extrapolation à l’ensemble des mutations pathogènes connues y est en germe. Nous ne sommes plus en présence d’indications vagues et contestables comme dans la « génétique récréative » pratiquée par 23andMe [
2,
3] mais de données médicales solides et dont les corrélats pathologiques sont bien établis. La question est donc sérieuse. Elle l’est d’autant plus que l’ère du « génome à 1 000 » dollars, le moment où le séquençage intégral d’un ADN deviendra aussi abordable qu’un examen par scanner, s’approche à grands pas. L’interprétation de la séquence fait elle aussi de grands progrès, comme on l’a vu récemment avec le logiciel proposé par Salzberg et Pertea [
9], et ne devrait pas constituer un goulet d’étranglement insurmontable. Et bien sûr, le progrès constant des connaissances va rendre ces données de plus en plus « parlantes ». Il y a donc urgence à mettre en place une bonne gestion de ces informations. L’objectif avancé pour ces études « préconceptionnelles » est d’éviter la naissance d’enfants atteints de maladies « d’une particulière gravité », comme l’on dit chez nous. Dans la pratique, ce but peut être atteint par le choix d’un partenaire qui ne soit pas porteur (à l’état hétérozygote) d’une mutation pathogène détectée (également à l’état hétérozygote) chez l’intéressé. Cela ressemble dangereusement à un « permis de procréer », et l’on peut imaginer que, par exemple, la prise en charge d’un enfant handicapé soit refusée par l’assurance maladie si toutes les « précautions génétiques » n’ont pas été prises. Il est bien sûr possible de contourner le problème en recourant à la fécondation in vitro suivie d’un diagnostic préimplantatoire afin de n’implanter qu’un embryon indemne, mais il s’agit là d’un procédé encore complexe, aléatoire et dont la pratique est beaucoup plus restreinte qu’on ne l’imagine généralement3,. L’organisation mise en place pour la maladie de Tay-Sachs montre comment cet objectif peut être atteint, mais dans des conditions bien particulières. Ce programme a été institué au sein de communautés juives orthodoxes, groupes très cohésifs et dans lesquels la pression sociale est forte. Ses modalités sont les suivantes : tous les élèves du secondaire sont testés, mais le résultat ne leur est pas communiqué, et un code à six chiffres est attribué à chacun d’eux. Si un mariage est ultérieurement envisagé, le couple peut appeler via une hotline un organisme appelé Dor Yeshorim
4, chargé de la gestion de ces informations (Figure 3). Après communication des deux codes, il leur sera alors indiqué s’ils sont ou non compatibles. Apparemment, le mariage ne se fait pas s’il y a incompatibilité - et, dans ce milieu, les conceptions hors mariage sont exceptionnelles. C’est ainsi que l’incidence de la maladie a pu être réduite presque à zéro, sans pour autant stigmatiser a priori des personnes en leur attribuant le statut de porteurs - ce qui pourrait les rendre « inmariables » au sein de la communauté. Ce système a si bien fonctionné qu’il est actuellement étendu à plusieurs maladies génétiques fréquentes chez les Ashkénazes5, c’est le « panel ashkénaze » mentionné dans le Tableau I - on voit pourtant que sa généralisation à une population « tout venant » ne se ferait pas sans difficulté.
 | Figure 3
Formulaire pour être testé dans le cadre du système Dor Yeshorim (spécimen).
|
A contrario, comme nous l’avons déjà mentionné, le dépistage des porteurs de la drépanocytose, aux États-Unis, a donné lieu à une stigmatisation se traduisant notamment par la fermeture de certaines carrières (pilote d’avion par exemple) aux hétérozygotes, sans que cela soit le moins du monde justifié par des données médicales. Il est donc clair que ces informations sont à manier avec beaucoup de précautions. Certes, dans l’état actuel des pratiques et de la législation françaises, le problème n’est pas immédiat, le garde-fou du médecin et/ou du juge étant en principe solide. Mais les choses peuvent changer très vite, bien plus vite que la loi, comme on le voit pour les tests de paternité fondés sur l’ADN : en principe interdits, avec des pénalités dissuasives pour ceux qui y auraient recours, ils sont en fait très largement pratiqués via Internet. « On n’empêchera pas les individus d’accéder à leur moi génétique » [
10]… À tout le moins, un principe essentiel doit être sauvegardé : celui de l’intimité génétique, le fait que de telles analyses ne soient pratiquées qu’à l’initiative explicite de l’individu et que le résultat n’en soit communiqué à des tiers qu’après autorisation expresse et aux seules personnes nommément désignées. Reste que, au-delà des employeurs et des assureurs qui seront naturellement très intéressés par ces données, la pression sociale peut s’exercer sur les individus et les pousser à communiquer des informations qu’ils auraient préféré garder par devers eux - ou que, même, ils n’auraient pas souhaité connaître. Conflit d’intérêts
L’auteur déclare n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Footnotes |
1.
Jordan
B
. À la recherché de l’héritabilité perdue… . Med Sci (Paris).
2010; ; 26 : :541.-543. 2.
Ducournau
P
,
Gourraud
PA
,
Rial-Sebbag
E
, et al.
Tests génétiques en accès libre sur Internet : stratégies commerciales et enjeux éthiques et sociétaux . Med Sci (Paris).
2011; ; 27 : :95.-102. 3.
Jordan
B
. Les tests génétiques grand public en « caméra cachée » . Med Sci (Paris).
2011; ; 27 : :103.-106. 4.
Kaback, Michael M
. Population-based genetic screening for reproductive counseling : the Tay-Sachs disease model . Eur J Pediatr.
2000; ; 159 : :S192.-S195. 5.
Beutler
E
,
Boggs
DR
,
Heller
P
, et al.
Hazards of indiscriminate screening for sickling . N Engl J Med.
1971; ; 285 : :1485.-1486. 6.
Bell
CJ
,
Dinwiddie
DL
,
Miller
NA
, et al.
Carrier testing for severe childhood recessive diseases by next-generation sequencing . Sci Transl Med.
2011; ; 3 : :65ra4.. 7.
Bale
S
,
Devisscher
M
,
Criekinge
WV
, et al.
MutaDatabase : a centralized and standardized DNA variation database . Nat Biotechnol.
2011; ; 29 : :117.-118. 8.
Jordan
B
. Exomes, le retour… . Med Sci (Paris).
2011; ; 27 : :220.-222. 9.
Jordan
B
. Séquence personnelle et tests génétiques : le pavé dans la mare . Med Sci (Paris).
2010; ; 26 : :999.-1001. 10.
Le Coz
P
. On n’empêchera pas les individus d’accéder à leur moi génétique . La Croix. , 9 janvier.
2009. |