| |
| Med Sci (Paris). 2011 April; 27(4): 425–432. Published online 2011 April 28. doi: 10.1051/medsci/2011274020.Représentation en sciences du vivant (7) - Le temps, sculpteur de la cellule ? François Amblard1* and Sylvie Coscoy1* 1Centre de recherche de l’Institut Curie et CNRS, UMR168, 26, rue d’Ulm, 75248Paris, France |
La cellule est aujourd’hui considérée comme l’unité fonctionnelle fondamentale du vivant et sa représentation est multiforme. La microscopie électronique fournit une image géométrique très fine des structures intracellulaires - organites intracellulaires, pores nucléaires, cytosquelette, etc. - et de leur agencement dans l’espace. La vision génétique et biochimique, beaucoup moins géométrique, est celle d’un ensemble de gènes ou de protéines qui interagissent au sein de divers programmes, de façon autonome ou en échangeant des informations avec le milieu extracellulaire. Avec la possibilité plus récente de filmer le monde moléculaire et microscopique, la vision implicitement statique de la microscopie électronique a cédé progressivement la place à une représentation très dynamique de l’organisation cellulaire. De nombreux concepts prennent forme, non seulement dans l’espace mais aussi dans le temps, avec des échelles de temps allant de la picoseconde (10−12 s) jusqu’à la durée de vie de la cellule (Figure 1). Nous proposons ici une réflexion sur les échelles de temps observables et l’importance du contrôle du temps pour le bon fonctionnement cellulaire. Les exemples seront tirés des problèmes posés par le cytosquelette, le transport intracellulaire et les aspects dynamiques de la stabilité collective des cellules dans un tissu.
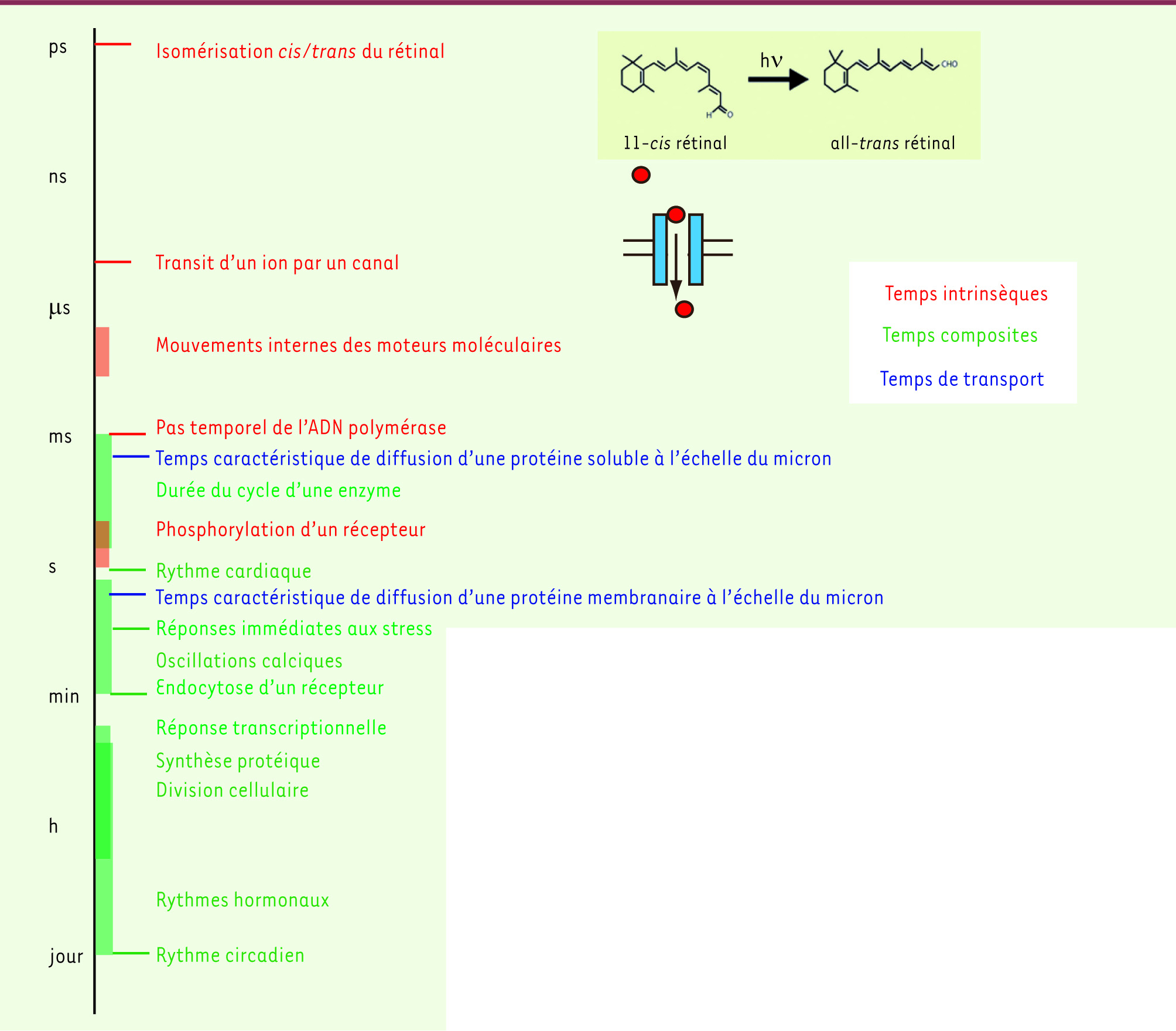
 | Figure 1.
Échelle de quelques temps caractéristiques des processus cellulaires. Les processus les plus rapides sont en général ceux qui sont associés aux modifications induites dans les petites molécules par la lumière ou par les effets d’un champ électrique. L’activité de molécules plus grosses correspond souvent à un changement de conformation dont le temps caractéristique limite la cinétique fonctionnelle. Dans les processus qui mettent en jeu des partenaires mobiles, le temps caractéristique peut dépendre fortement du temps de diffusion, qui varie lui-même suivant la taille des molécules et leur contexte - cytosol ou membrane par exemple. Pour ces processus plus complexes, les temps caractéristiques (indiqués en vert) résultent de la composition des temps nécessaires aux opérations chimiques élémentaires (représentées en rouge) et des temps de transport des multiples participants aux échelles spatiales concernées (indiqués en bleu). Pour mémoire, les petites molécules - ions, métabolites divers, etc. - se dispersent avec des coefficients de diffusion de l’ordre de 103 µm2/s, tandis que les protéines ont des coefficients de l’ordre de quelques 10 µm2/s dans le cytosol et quelques 10-1 µm2/s dans les membranes. |
|
Les temps de la cellule : chimie, biologie et physique Dans la cellule, les processus les plus rapides sont ceux qui mettent en jeu des transformations chimiques induites par l’absorption de lumière, comme celle qui accompagne la détection optique par la rétine, environ 1 ps (Figure 1). En aval de cette échelle de temps s’étend une longue cascade de temps caractéristiques intimement liés aux processus physicochimiques élémentaires et aux échanges d’énergie : mouvements microscopiques de petites molécules (1 ns à 1 µs), passage d’un ion dans un canal (environ 0,1 µs), transitions conformationelles de moteurs moléculaires (10 à 100 µs), pas des ADN polymérases (1 ms). L’agencement complexe de ces phénomènes élémentaires dans les processus physiologiques à l’échelle cellulaire puis systémique conditionne la vitesse avec laquelle la cellule exécute l’ensemble de ses fonctions. L’enjeu du contrôle des temps cellulaires est non seulement d’assurer une certaine « performance », mais surtout de permettre une coordination essentielle entre une multitude de processus élémentaires. Il s’agit là d’un domaine de recherche très riche, que l’on connaît à travers quelques concepts essentiels en physiopathologie : rythmes circadiens et origine de leur précision et de leurs dérèglements ; horloge de segmentation somitique au cours de l’embryogenèse [
1] ; contraction des cellules cardiaques ; régulation du temps de division cellulaire. La régulation de nombreux phénomènes passe également par le couplage entre une réaction activatrice et son inhibition retardée. La compétition cinétique entre ces deux termes conditionne de très nombreux processus et fournit un mécanisme générique combinant réactivité et homéostasie. Ces processus biologiques complexes impliquent de nombreuses molécules dont les concentrations varient avec le temps mais aussi dans l’espace. L’évolution de ces concentrations obéit à la loi de la diffusion [
2] qui régit la façon dont une molécule présente initialement à un endroit donné se répand dans l’espace environnant par mouvements aléatoires (Figure 2A). La rapidité de la diffusion conditionne très largement les cinétiques réactionnelles en biochimie. Si par exemple deux réactions en tandem siègent en deux endroits différents, comme souvent dans la cellule, le produit de la première ne sera disponible comme réactif pour la seconde qu’avec un certain retard, le délai moyen entre les deux réactions étant proportionnel au carré de la distance qui les sépare1. Un exemple célèbre intervenant dans de très nombreux processus est celui des phénomènes dits de « réaction-diffusion » qui met en scène le couple activateur/inhibiteur mentionné plus haut : si la diffusion du premier est plus rapide que celle du second, il peut apparaître une grande variété de structures spatiales [
3]. On voit immédiatement à quel point l’organisation temporelle d’un ensemble de réactions participant à une réponse biologique complexe dépendra de son organisation dans l’espace.
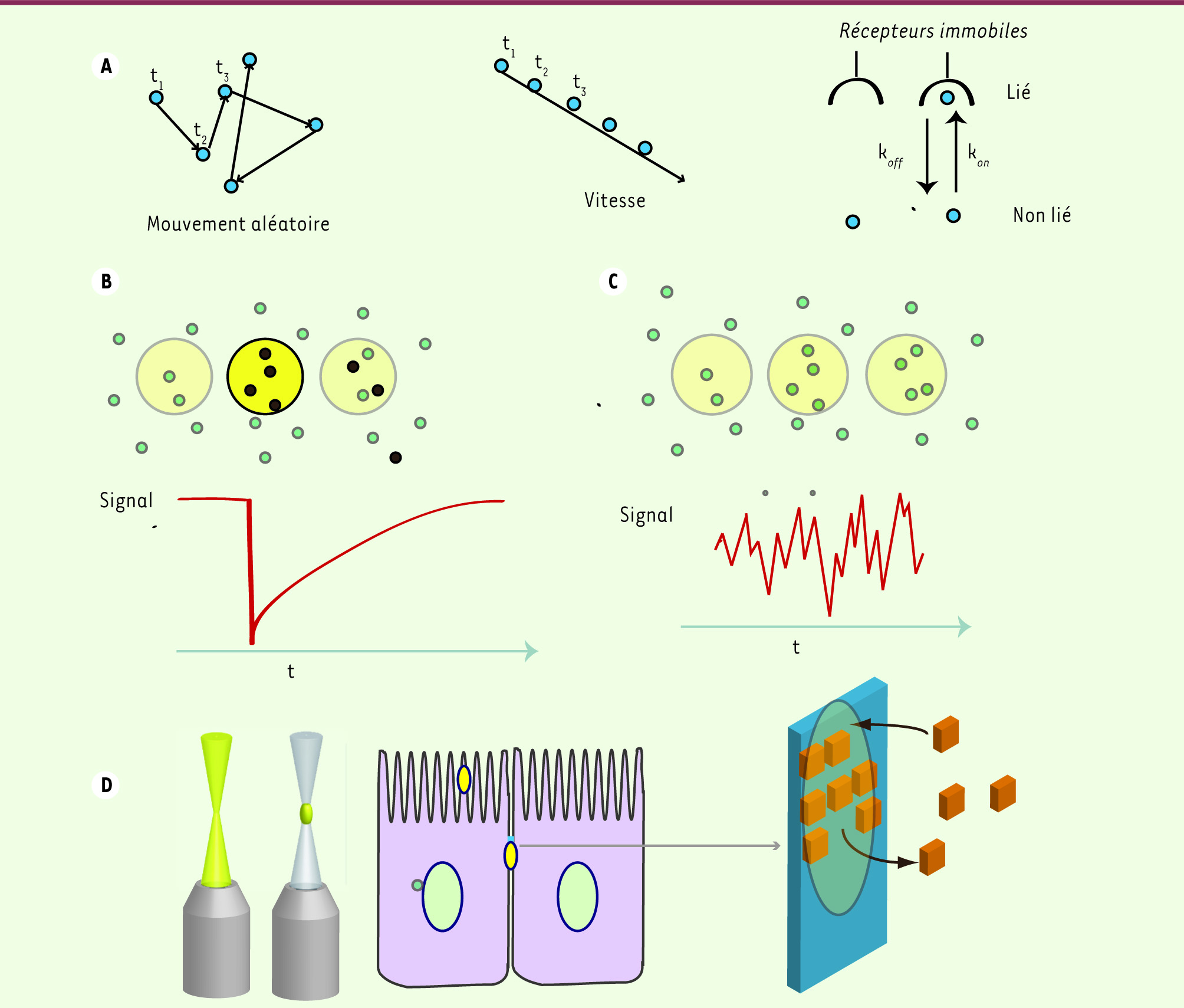
 | Figure 2
Mobilité des molécules et méthodes d’analyse. A. La mobilité des molécules relève schématiquement de trois processus élémentaires : (1) mobilité entretenue par la diffusion, conséquence inévitable de l’agitation thermique en solution, caractérisée par un coefficient de diffusion ; (2) mouvement directionnel, produit soit par un écoulement, soit par les moteurs moléculaires, et caractérisé par une vitesse ; et (3) possibilité pour la molécule considérée de se fixer sur un ligand immobile, ce qui bloque son mouvement pendant une durée dépendante de la cinétique de liaison/dissociation. Ce changement d’état chimique répond en général à une cinétique du premier ordre, et on peut le décrypter quantitativement à travers la mesure de la mobilité. B-D. FRAP (fluorescence recovery after photobleaching) et FCS (fluctuation correlation spectroscopy), deux méthodes de mesure de la mobilité des protéines dans la cellule, basées sur la variation du signal de fluorescence produit par des molécules préalablement marquées - protéines fluorescentes semblables à la green fluorescent protein ou marqueurs divers. B. La méthode de FRAP consiste à supprimer irréversiblement par photoblanchiment la fluorescence des molécules situées dans le volume choisi grâce une illumination locale intense. La fluorescence ne réapparaît dans ce volume que si des molécules non blanchies y pénètrent de nouveau. La cinétique de ce retour est très riche en informations quantitatives sur la nature de la mobilité, et permet de distinguer les trois scénarios mentionnés plus haut (A), voire des comportements hybrides, et d’extraire les caractéristiques quantitatives associées - constante de dissociation, coefficient de diffusion, ou encore la vitesse et la direction du transport. C. La méthode de FCS consiste à mesurer les fluctuations spontanées de la fluorescence dans un volume choisi, typiquement de l’ordre de 1 µm3, défini par la focalisation d’un faisceau laser. Si les molécules observées se déplacent, ces fluctuations accompagnent leur entrée et leur sortie du volume et nous renseignent donc quantitativement sur la mobilité. D’autres méthodes existent, basées sur la mesure des fluctuations d’intensité de fluorescence sur une série d’images - image correlation spectrosopy, speckle microscopy. D. Contrairement à la microscopie classique (faisceau coloré en jaune à gauche), l’excitation de la fluorescence en microscopie biphotonique est strictement restreinte au volume de focalisation, ce qui permet une mesure très locale de la dynamique (image de droite, mouvements vers et hors du volume photoblanchi). La mesure du retour de fluorescence par vidéomicroscopie 3D rapide fournit en outre des informations spatiales. |
|
Nouveaux outils d’investigation des propriétés dynamiques des protéines Les considérations précédentes mettent en lumière de nombreux problèmes : les échelles de temps rencontrées jouent-elles des rôles physiologiques importants, et lesquels ? Comment sont-elles contrôlées ? Comment le temps organise-t-il l’espace et réciproquement, lors de la morphogenèse et dans l’homéostasie cellulaire et tissulaire ? Autant de raisons pour élaborer de nouveaux outils d’investigation des propriétés dynamiques des protéines et de leurs assemblages. Quels sont-ils à ce jour ? Les objets qui nous intéressent sont d’abord les protéines, dont la localisation dans la cellule s’observe par microscopie de fluorescence avec une résolution spatiale de l’ordre de 0,2 à 0,3 µm à raison de 1 à 10 images par seconde en routine. De telles images donnent accès à la concentration des protéines, ou plus précisément au flux des photons émis par les protéines fluorescentes. D’une image à l’autre, cette concentration peut varier, sous l’effet d’un mouvement aléatoire ou dirigé des objets fluorescents, ou d’une variation du nombre de molécules émettrices en chaque point. L’exploration de ces deux types de phénomènes relève de l’analyse d’image que nous ne traitons pas ici, et dont les méthodes fournissent des informations très fines et quantitatives sur l’évolution et les mouvements des structures intracellulaires, voire des protéines individuelles (➜) [
15].
(➜) Voir l’article de I. Izeddin et al. m/s n 5 mai 2011, sous presse
Mais une série d’images de fluorescence ne reflète en général que la concentration des protéines, grandeur collective ne donnant aucune réponse directe à la question suivante : les protéines individuelles qui se « cachent » derrière l’ensemble mesuré sont-elles engagées dans des liaisons qui les immobilisent ou continuellement transportées par diffusion ou d’une autre manière, et remplacées rapidement par d’autres ? Nous entendrons par dynamique l’ensemble de ces comportements (Figure 2A). Plusieurs techniques permettent la mesure de cette dynamique. La méthode FRAP (abréviation de l’anglais pour : recouvrement de fluorescence après photoblanchiment) est la plus ancienne et la plus répandue [
13] : son principe consiste schématiquement à mesurer la rapidité du retour des molécules fluorescentes dans une région dont on les a préalablement fait « disparaître » par photoblanchiement (Figure 2B). Cette méthode [
4] proposée dès 1976 a connu un regain de popularité avec l’avènement des protéines fluorescentes dont la découverte et les applications ont valu le prix Nobel à Tsien, Chalfie et Shimomura en 2008 [
14]. Par fusion d’une séquence d’ADN codant pour une protéine donnée avec celle codant pour une protéine fluorescente dont on peut choisir la couleur (green fluorescent protein [GFP], red-, blue-, etc.), la protéine étudiée est rendue visible et peut être photoblanchie avec une perturbation minime de son activité. Nous avons exploité largement cette approche pour la mesure de la dynamique de protéines du cytosquelette - actine, myosine, ezrine, cadhérine, caténines [
5–
7] - en la combinant avec trois ingrédients nouveaux. Le premier ingrédient est l’excitation de la fluorescence par un processus d’absorption biphotonique2 [
8], qui a lieu dans un volume très restreint autour du point focal, d’environ 1 µm3
(Figure 2D). De ce fait, la mobilité est mesurée de façon locale, ce qui rend possible une cartographie 3D de la dynamique avec la même résolution que l’imagerie microscopique. Le deuxième ingrédient consiste à observer le retour de fluorescence, non pas au point photoblanchi seulement, mais dans tout l’espace 3D qui l’entoure. De cette façon, nous savons si la mobilité est organisée dans l’espace du fait de structures de transport (microtubules, vésicules, écoulements). Enfin, par l’usage de protéines de fusion vertes et rouges présentes dans un volume restreint et grâce à l’excitation biphotonique, nous pouvons mesurer très précisément la mobilité de deux protéines prises dans un volume rigoureusement identique. Il est ainsi possible de comparer la dynamique de protéines en interaction, dans une jonction cellulaire par exemple. La mesure de la mobilité de protéines est aussi possible entre autres méthodes par la technique dite de fluctuation de fluorescence (FCS en anglais), dont le principe est de mesurer le coefficient de diffusion d’une molécule fluorescente à partir de la rapidité des fluctuations relatives de l’intensité qu’elle produit dans un volume donné (Figure 2C, D). Cette méthode est performante dans certaines circonstances, mais d’un emploi plus limité que la méthode par FRAP. D’un point de vue pratique, par FRAP, on peut cartographier le comportement dynamique moyen d’une protéine donnée, à savoir : le temps de résidence en chaque endroit, la constante d’association/dissociation si elle s’engage dans un lien qui la retient, le coefficient de diffusion, ou encore la vitesse et la direction de son mouvement si elle est transportée activement par des moteurs moléculaires (Figure 2A). |
Un ballet moléculaire incessant Grâce à cet ensemble de méthodes, il est maintenant établi que de très nombreuses protéines sont mobiles. C’est évidemment vrai de protéines cytosoliques, dont les coefficients de diffusion sont maintenant bien connus - typiquement de 102 à 103 µm2/s suivant la taille. Mais c’est aussi le cas pour diverses protéines, indépendamment de la permanence de la structure à laquelle elles appartiennent. Ainsi, les monomères d’actine présents dans le cytosquelette apical stable de cellules épithéliales n’y résident qu’environ 2 minutes, tout comme les molécules d’ezrine qui assurent la liaison actine-membrane [5]. Par ailleurs, dans les jonctions adhérentes interépithéliales, les cadhérines qui en sont le principal ingrédient ne restent pas plus de 4 à 5 minutes, alors que ces structures adhésives sont stables bien au-delà de l’heure [7]. D’autre part, les pores nucléaires offrent une très belle structure avec une particularité dynamique remarquable : le pore existe comme un assemblage relativement stable en dehors de la mitose, tandis que ses composants élémentaires, plusieurs dizaines de nucléoporines, n’y résident que pendant des temps limités. Suivant les nucléoporines, ce temps va de 20 s à 70 h [
9]. Enfin, la touffe ciliaire qui assure la détection acoustique à l’apex des cellules de l’oreille interne montre une structure en étages remarquablement précise (Figure 3A), et on sait maintenant que la longueur des microvillosités est directement liée à la vitesse de polymérisation de l’actine par laquelle se renouvellent ses microfilaments [
10].
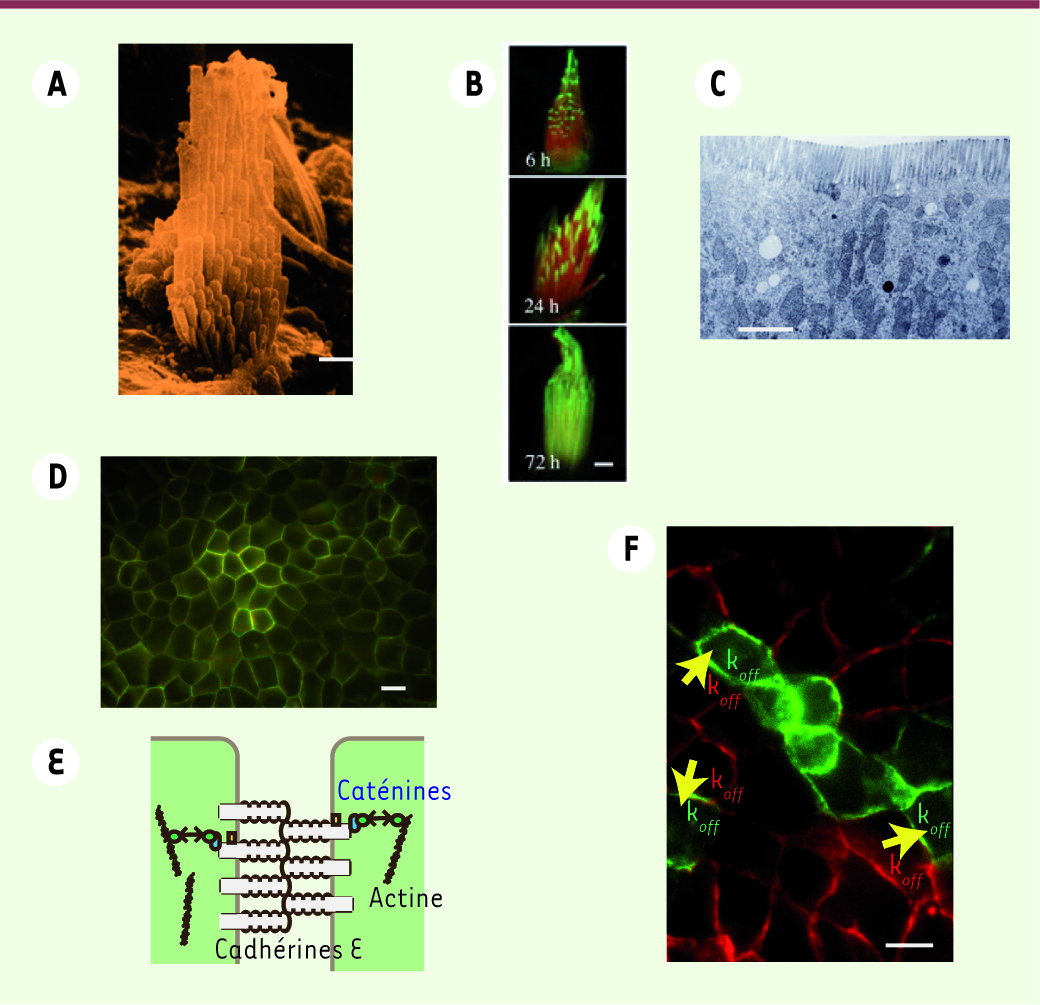
 | Figure 3.
Dynamique des protéines, morphologie et stabilité. A-C. Certaines cellules épithéliales comportent à leur apex des protrusions membranaires très précisément construites : (A, B) les stéréocils formant la touffe ciliaire des cellules de l’oreille interne (barre : 2,5 µm) ( B, d’après [ 10]), et (C) les microvillosités de l’épithélium intestinal (barre : 1 µm). B. Chaque stéréocil possède une longueur précise et stable et contient un faisceau de microfilaments d’actine, en permanence renouvelé par polymérisation au bout libre et dépolymérisation à la base. Le renouvellement, matérialisé par le remplacement d’une actine fluorescente rouge par une actine verte, s’effectue à une vitesse d’autant plus grande que le stéréocil est long [ 10]. D-F. Les cellules des épithéliums, jointives, exercent des tensions mécaniques les unes sur les autres par l’intermédiaire de jonctions adhésives visualisées ici en fluorescence (D) (barre : 10 µm), et dont la E-cadhérine est la protéine d’adhésion clé (voir
E
). La stabilité du tissu requiert l’équilibre de ces forces, sans quoi les jonctions se déplacent de façon désordonnée. E. Les cadhérines tout comme les autres composants -actine, caténines, sont en permanence renouvelées et ne résident typiquement dans la jonction que quelques minutes. Nous avons observé que l’équilibre est maintenu de façon active par l’ajustement permanent des cadhérines, et que c’est l’endocytose qui contrôle le temps passé par la cadhérine à la jonction. F. Grâce à la construction d’épithéliums exprimant des cadhérines vertes et rouges, nous avons observé que l’équilibre entre deux cellules exige que l’endocytose ait lieu avec la même rapidité dans les deux partenaires. Le renouvellement de la cadhérine aux jonctions s’effectue par des réactions du premier ordre, avec des constantes de temps égales de part et d’autre de la jonction à l’équilibre ; les flèches correspondent à l’endroit de la mesure (données personnelles). (Photos A et C, © Inserm-Serimedis). |
Ce ne sont là que quelques exemples d’une liste de faits maintenant très longue, laissant entrevoir que la plupart des structures biologiques stationnaires dans la cellule sont le lieu d’un ballet moléculaire incessant par lequel les molécules sont remplacées sur des échelles de temps beaucoup plus courtes que la durée de vie de la structure elle-même. Énoncées sous cette forme, ces observations ne devraient pas nous surprendre en première analyse. En effet, les interactions entre protéines étant la plupart du temps non covalentes, elles sont réversibles et soumises à la loi d’action de masse3,. On s’attend donc naturellement à ce que les composants d’un complexe multiprotéique s’en détachent au bout d’un temps donné, suivant leur cinétique propre de dissociation. Mais cette façon de voir suppose implicitement que les structures en question sont à l’équilibre thermodynamique, ce qui est rarement le cas. En effet, qu’il s’agisse de la polymérisation du cytosquelette, l’activation ou l’inactivation des protéines, ou encore du maintien de complexes multimoléculaires, de multiples processus irréversibles sont en jeu avec une dépense énergétique constante : phosphorylation, déphosphorylation, clivages, modifications post-traductionnelles. Nous ne pouvons donc pas dissocier l’existence d’une structure biochimique dynamique, même stationnaire, de la présence d’un flux de molécules pour l’alimenter et d’un flux inverse de dissociation. Ces deux flux ne sont en général pas symétriques du point de vue chimique, et la structure est le produit de leur compétition cinétique. Le meilleur exemple, classique, est celui d’un microfilament d’actine, connu pour la dissymétrie des processus de polymérisation et dépolymérisation qui se distinguent par une étape d’hydrolyse d’ATP. Les observations suggèrent que ce schéma est extrêmement général. Nous avons disséqué un mécanisme de ce type dans le cas d’une protéine d’architecture du cytosquelette épithélial, l’ezrine, dont l’engagement repose sur un cycle rapide - 2 minutes - constitué d’une succession d’étapes dont plusieurs sont irréversibles [5]. Une observation analogue a été faite pour les E-cadhérines, protéines d’adhésion entre cellules épithéliales : ces molécules entrent puis sortent des jonctions adhérentes par des processus distincts [7]. Dans ces exemples, bien que les structures observées, cytosquelette apical et jonctions, soient stationnaires et n’évoluent pas à l’échelle du temps de l’expérience, une dépense énergétique importante a lieu pour entretenir la régénération permanente des structures. |
Question centrale et pistes de travail Ces observations conduisent à une question centrale, quelque peu finaliste. À quelle nécessité fonctionnelle répond la dépense énergétique constatée dans ces diverses situations pourtant stationnaires ? En quoi cette dépense serait-elle physiologiquement avantageuse pour la cellule, voire optimisée ? Une première piste de réflexion est celle du contrôle de la morphologie des structures cellulaires sous l’effet direct de leurs propriétés temporelles. Raisonnons sur la touffe ciliaire déjà mentionnée (Figure 3A). Dans les stéréocils qui la composent, les microfilaments polymérisent à l’extrémité libre (Figure 3B) puis dépolymérisent à la base, et leur longueur est donc le résultat direct de la compétition cinétique entre ces processus. Leur longueur stationnaire est corrélée précisément et individuellement à leur dynamique : les stéréocils les plus longs correspondent à une vitesse d’assemblage plus grande à leur extrémité libre. La dynamique dicte ici la géométrie. Dans le cas des microvillosités parfaitement régulières du tissu intestinal (Figure 3C), un autre scénario est envisageable. Supposons que la polymérisation à l’extrémité libre soit d’autant plus rapide que la concentration de monomères y est élevée et que le flux de monomères nécessaire pour l’approvisionner soit limité par la diffusion longitudinale. Alors, la concentration de monomères à l’extrémité sera d’autant plus faible que sa distance à la base est grande. La conséquence immédiate est que la vitesse de polymérisation est d’autant plus grande que la structure est courte, et inversement. Par ce couplage inverse entre longueur et cinétique d’assemblage, la taille des microvillosités peut se stabiliser spontanément autour d’une valeur fixée par les cinétiques du transport et de la biochimie. En bref, la dépense énergétique associée à la dé/polymérisation fournit un mécanisme de stabilisation de l’objet et détermine sa taille. La géométrie étonnamment régulière des microvillosités intestinales résulterait directement de ce couplage entre temps et forme, mais cette hypothèse reste à démontrer. La dépense énergétique, bien supérieure en général au coût thermodynamique de l’assemblage des structures biologiques, serait le prix à payer pour un contrôle morphologique très fin, probablement nécessaire au bon fonctionnement de l’épithélium. Dans d’autres expériences, nous observons que la stabilité des jonctions intercellulaires d’un épithélium est étroitement corrélée avec le fait que le renouvellement des E-cadhérines se fait à la même vitesse de part et d’autre de la jonction (Figure 3F) (de Beco et al., manuscrit en préparation) Plus précisément encore, le temps de résidence des E-cadhérines à la jonction semble plus déterminant pour le contrôle des forces mécaniques transmises par l’adhésion que leur nombre à la jonction. Ce point de vue nous fait passer d’un raisonnement de type « structure-fonction » à la notion du contrôle de la fonction par la dynamique. Nous nous éloignons du cadre de la thermodynamique à l’équilibre et il convient probablement de remplacer le principe de minimisation de l’énergie libre, encore souvent employé de façon explicite ou implicite pour raisonner sur la biochimie et sur les structures cellulaires, par un principe d’optimisation de la dynamique, asservie à la fonction au sens large. Eu égard à la diversité considérable des comportements dynamiques, il s’agit là d’un réel défi pour la biologie, d’autant plus que les lois mathématiques et physiques sous-jacentes n’ont été étudiées que dans des situations relativement simples par rapport à la complexité du vivant. De façon très étonnante, cette vision du vivant structuré comme un flux plutôt que comme un ensemble de structures équilibrées a nourri l’intuition précoce de Engels dans sa dispute avec le naturaliste Dühring [
11]. À l’échelle de l’organisme en développement, notons que ce point de vue est voisin de celui de d’Arcy Thompson qui donne aux aspects dynamiques un rôle déterminant dans la morphogenèse des organismes [
12]. Sur une base encyclopédique d’observations disponibles au début du xx
e siècle, d’Arcy Thompson a pu formuler une description parfois mathématique et quantitative du lien entre forme et dynamique, mais seulement à une échelle macroscopique. Reprendre cette perspective dans le contexte microscopique de la biologie cellulaire et de la biochimie moderne est une belle aventure sur un terrain presque vierge. La seconde hypothèse s’appuie sur l’analogie suivante : si un ensemble de réactions chimiques successives organisées en cycle atteint un régime stationnaire, alors le temps caractéristique du cycle, fixé en général par son étape lente, sera aussi un temps d’adaptation. En effet, si une perturbation est apportée dans ce cycle, en modifiant une de ses constantes cinétiques par exemple, alors l’état stationnaire peut se déplacer vers un nouvel état stationnaire ; et le temps de ce déplacement sera d’autant plus bref que le cycle est rapide. Autrement dit, si le maintien d’une structure biologique passe par un processus de renouvellement donné, le temps typique associé conditionnera le temps de réponse de la structure à une modification des conditions biochimiques de son maintien. La dépense énergétique entraînée pourrait ainsi assurer une capacité d’adaptation rapide et en déterminer le temps caractéristique. L’adaptation étant une caractéristique essentielle en physiologie, on s’attend à une forte pression de sélection sur la dynamique et les constantes de temps clés, même si le coût énergétique en est élevé. Si la dissipation d’énergie métabolique est à ce point importante, on conçoit aisément qu’un dérèglement pathologique affectant les constantes de temps aient des conséquences adverses, même si les concentrations des molécules impliquées varient peu. Ce point de vue pourrait s’avérer utile pour explorer une question difficile : en quoi le partage des ressources métaboliques entre cellules tumorales - plutôt plus actives - et tissus sains conduit à une cohabitation bénigne ou au développement malin ? En conclusion, les observations relatées ici indiquent clairement l’importance des propriétés temporelles associées aux différentes voies de dépense d’énergie métabolique. En outre, les hypothèses proposées pointent vers la nécessité d’un cadre conceptuel et méthodologique nouveau pour comprendre comment la multitude organisée des temps caractéristiques assure le fonctionnement optimal de la cellule. Conflit d’intérêts
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Footnotes |
1.
Aulehla
A
,
Pourquié
O
. Oscillating signaling pathways during embryonic development . Curr Opin Cell Biol.
2008; ; 20 : :632.-637. 2.
Berg
HC
. Random walks in biology . Princeton: : Princeton University Press; , 1993 : :164 p.. 3.
Murray
JD
. Mathematical biology II spatial models and biomedical applications. (3e ed) , 3e ed . Berlin : : Springer Verlag; , 2003 : :736 p.. 4.
Axelrod
D
,
Koppel
DE
,
Schlessinger
J
, et al.
Mobility measurement by analysis of fluorescence photobleaching recovery kinetics . Biophys J.
1976; ; 16 : :1055.-1069. 5.
Coscoy
S
,
Waharte
F
,
Gautreau
A
, et al.
Molecular analysis of microscopic ezrin dynamics by two-photon FRAP . Proc Natl Acad Sci USA.
2002; ; 99 : :12813.-12818. 6.
Waharte
F
,
Brown
CM
,
Coscoy
S
, et al.
A two-photon FRAP analysis of the cytoskeleton dynamics in the microvilli of intestinal cells . Biophys J.
2005; ; 88 : :1467.-1478. 7.
de Beco
S
,
Gueudry
C
,
Amblard
F
,
Coscoy
S
. Endocytosis is required for E-cadherin redistribution at mature adherens junctions . Proc Natl Acad Sci USA.
2009; ; 106 : :7010.-7015. 8.
Amblard
F
. La microscopie à deux photons . Pour la science.
2005; ; 338 : :110.-114. 9.
Rabut
G
,
Doye
V
,
Ellenberg
J
. Mapping the dynamic organization of the nuclear pore complex inside single living cells . Nat Cell Biol.
2004; ; 6 : :1114.-1121. 10.
Rzadzinska
AK
,
Schneider
ME
,
Davies
C
, et al.
An actin molecular treadmill and myosins maintain stereocilia functional architecture and self-renewal . J Cell Biol.
2004; ; 164 : :887.-897. 11.
Engels
F
. Anti-Dühring . Paris: : Éditions sociales; , 1877 : :502 p.. 12.
Thompson d’Arcy
W
. On growth and form . Cambridge: : Cambridge University Press; , 1917 ; ; XVI : :794 p.. 13.
Trugnan
G
,
Fontanges
P
,
Delautier
D
, et al.
FRAP, FLIP, FRET, BRET, FLIM, PRIM… De nouvelles techniques pour voir la vie en couleur ! . Med Sci (Paris).
2004; ; 20 : :1027.-1034. 14.
Pasquier
H
, Prix Nobel de Chimie . (Osumo Shimomura, Martin Chalfie et Roger Y. Tsien) : lumière sur les protéines fluorescentes . Med Sci (Paris).
2008; ; 24 : :985.-986. 15.
Izeddin
I
,
Darzacq
X
,
Dahan
M
. Microscopies cellulaires à l'échelle de la molécule individuelle . Med Sci (Paris).
2011; ; 27 ( sous presse.). |