| |
| Med Sci (Paris). 2011 April; 27(4): 357–359. Published online 2011 April 28. doi: 10.1051/medsci/2011274008.ZNF703 : un nouvel oncogène du cancer du sein Christophe Ginestier,1* Fabrice Sircoulomb,1 Emmanuelle Charafe-Jauffret,1 Max Chaffanet,1 and Daniel Birnbaum1 1Centre de recherche en cancérologie de Marseille, Laboratoire d’oncologie moléculaire, UMR891 Inserm ; Institut Paoli-Calmettes, 27, boulevard Leï Roure, BP 30059, 13273Marseille, France MeSH keywords: Antinéoplasiques hormonaux, pharmacologie, usage thérapeutique, Tumeurs du sein, traitement médicamenteux, génétique, anatomopathologie, Protéines de transport, physiologie, Lignée cellulaire tumorale, Transformation cellulaire néoplasique, Chromosomes humains de la paire 8, ultrastructure, Résistance aux médicaments antinéoplasiques, Femelle, Amplification de gène, Régulation de l'expression des gènes tumoraux, Humains, Modèles biologiques, Complexes multiprotéiques, Cellules souches tumorales, Protéines nucléaires, Oncogènes, Protéines de fusion recombinantes, Transcription génétique |
Le cancer du sein est une maladie hétérogène dans laquelle on distingue plusieurs formes histocliniques, plusieurs sous-types moléculaires définis par les analyses d’expression génique (par exemple basal, ERBB2 et luminal [
1]), et de multiples altérations génétiques comme des mutations, pertes ou amplifications de gènes. Pour proposer des pronostics et des traitements appropriés, il est important de diviser cette maladie en entités homogènes. On a toujours considéré qu’une telle hétérogénéité reflètait les différentes combinaisons d’anomalies génétiques que peut accumuler une cellule cancéreuse au cours de la progression tumorale. On associe notamment la perte de la cadhérine E aux formes lobulaires, l’amplification du gène ERBB2 (v-erbb2 erythroblastic leukemia viral oncogene homolog 2, un récepteur tyrosine kinase de la famille du récepteur à l’EGF, epithelial growth factor) au sous-type ERBB2 ou les mutations du gène BRCA1 (breast cancer antigen) au sous-type basal. Depuis le début des années 2000 et l’émergence du concept des cellules souches cancéreuses (CSC) [
2,
3], les fondements moléculaires de cette hétérogénéité intertumorale peuvent être interprétés différemment [
4]. Plusieurs études ont montré que les cellules tumorales sont organisées selon une hiérarchie dont le sommet est représenté par une petite population de cellules, les CSC, capables de générer l’ensemble des cellules de la masse tumorale. Les CSC seraient issues de la transformation de cellules souches ou progénitrices (plus matures que les précédentes dont elles sont issues) de la glande mammaire et conserveraient, au moins en partie, les caractéristiques de leurs cellules d’origine. Les CSC seraient à l’origine de l’émergence et de la progression des cancers du sein, mais aussi de la résistance thérapeutique. Le déclenchement du processus tumoral à partir d’une cellule souche ou d’un progéniteur plus mature pourrait ainsi contribuer à l’hétérogénéité intertumorale. Cette dernière trouverait donc sa source à la fois au niveau cellulaire (cellule d’origine) et moléculaire (anomalies génétiques). Les classifications histocliniques, cellulaires et moléculaires sont de plus en plus précises, il reste maintenant à comprendre les relations qui les unissent. Deux études récemment publiées [4,
5] contribuent à faire le lien entre les sous-types moléculaires, les altérations géniques et les cellules d’origine. |
L’énigme de l’amplification 8p12 L’amplification de la région chromosomique 8p12 est détectée dans environ 15 % des cancers du sein. Il est vraisemblable que dans cette région amplifiée, un ou plusieurs oncogènes se trouvent sélectionnés au cours de l’oncogenèse mammaire. L’identité de ce ou ces gène(s) est longtemps restée une énigme. Plusieurs d’entre eux, comme FGFR1 (fibroblast growth factor receptor 1), RAB11FIP1 (RAB11 family interacting protein 1) ou PPAPDC1P (phosphatidic acid phosphatase type 2 domain containing 1B) ont été proposés, mais aucune de leurs caractéristiques fonctionnelles ne parvenait à expliquer totalement le phénomène [
6–
8]. En effet, l’amplification 8p12 est plus spécifique des cancers du sein exprimant les récepteurs aux oestrogènes (sous-type luminal) et est souvent associée à l’amplification du gène codant la cycline D1 (CCND1), situé en 11q13. L’oncogène maître de la région 8p12 devait donc avoir à la fois une relation avec l’expression du récepteur aux oestrogènes (RE), conférer un potentiel prolifératif et partager une voie commune avec la cycline D1 [
9].
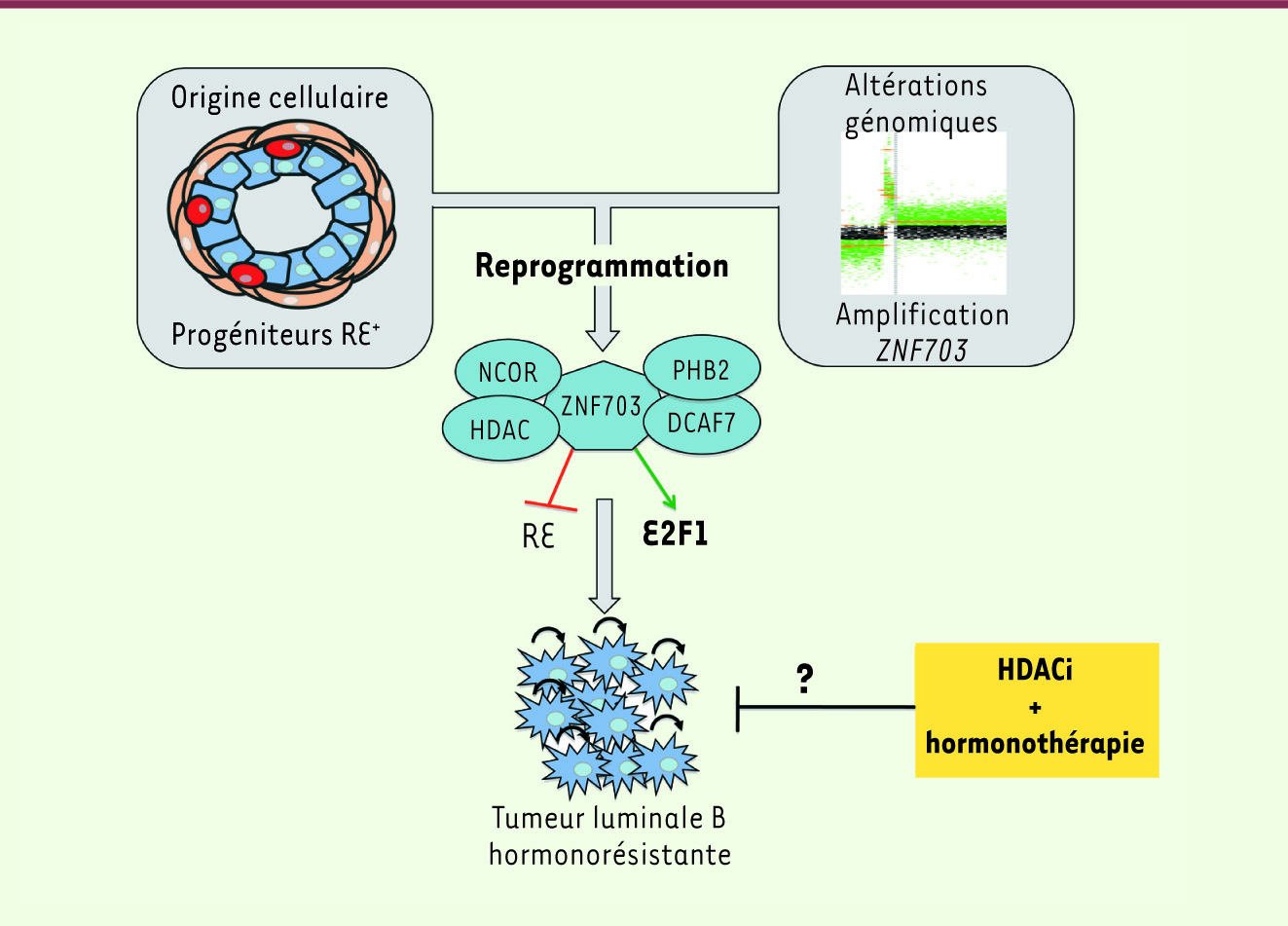
 | Figure 1
ZNF703, oncogène du sous-type luminal B des cancers du sein. L’amplification et la surexpression de ZNF703 dans une cellule progénitrice exprimant le récepteur aux oestrogènes (RE+) pourraient induire une reprogrammation de ces cellules en cellules souches cancéreuses en affectant deux facteurs transcriptionnels majeurs : RE et E2F1. La conséquence fonctionnelle est une perturbation du cycle cellulaire et de l’autorenouvellement des cellules souches cancéreuses. Ces caractéristiques pourraient expliquer l’hormonorésistance associée aux tumeurs luminales B. Ainsi, l’utilisation d’inhibiteurs des HDAC (HDACi) pourrait sensibiliser les tumeurs luminales B à l’hormonothérapie. |
|
Identification de l’oncogène ZNF703 Deux études récentes ont développé des stratégies similaires afin d’identifier le ou les gènes maîtres de l’amplicon 8p12 [4, 5], toutes deux fondées sur l’analyse intégrée des profils génomiques et transcriptionels de plus de 1 000 tumeurs du sein. Ces analyses ont permis de mettre en évidence la plus petite région amplifiée commune à toutes les tumeurs présentant une amplification de la région 8p12. Dans cette région, seul le gène ZNF703 présentait une corrélation systématique entre amplification et surexpression. Dans de rares cas il était le seul gène amplifié [5]. Les deux études ont aussi montré que l’expression de ZNF703 est sous le contrôle des oestrogènes et que sa surexpression est associée aux tumeurs de sous-type luminal B et à une diminution de la survie sans récidive. Toutes ces observations font de ZNF703 un candidat idéal pour le rôle d’oncogène maître de l’amplicon 8p12 des cancers du sein. Ces observations ont été validées in vitro grâce à la construction de vecteurs plasmidiques induisant la surexpression de la protéine ZNF703. Cette dernière augmente la prolifération des cellules de la lignée tumorale mammaire MCF7 et induit la formation de foyers transformés dans les fibroblastes murins NIH3T3. |
ZNF703 est un cofacteur d’un complexe nucléaire Le rôle biologique de ZNF703 n’avait jamais été décrit. Cette protéine appartient à la famille des régulateurs transcriptionels NET (pour Noc/ Elbow/Tlp1) qui jouent un rôle dans la morphogenèse et le processus de différenciation cellulaire [
10]. ZNF703 est localisée dans le noyau sous forme de foyers nucléaires fixés à l’ADN. Une approche d’analyse protéomique par spectrométrie de masse a permis de mettre en évidence des facteurs interagissant avec ZNF703, dont DCAF7 et PHB2, qui régulent l’activité transcriptionnelle en s’associant au complexe de corépresseurs nucléaires NCOR1/NCOR2 [4]. Ce complexe régule l’expression de nombreux gènes cibles via l’activation d’histones déacétylases (HDAC). Cette fonction de répresseur de la transcription via l’activité HDAC a été confirmée par ChIP (chromatin immunoprecipitation) avec la mise en évidence du recrutement de ZNF703 sur les sites régulateurs d’HDAC1 et EP300 [5]. |
ZNF703 régule la biologie des cellules souches cancéreuses Afin de comprendre l’activité oncogénique de ZNF703, les auteurs ont identifié les cibles transcriptionnelles. La comparaison des profils transcriptionnels des cellules MCF7 sauvages et des MCF7 surexprimant ZNF703 ont permis d’identifier des réseaux de gènes impliqués dans la régulation de la biologie des cellules souches tels que les voies de signalisation WNT/caténine β, TGFβ (transforming growth factor) ou NOTCH [5]. Des approches fonctionnelles ont montré que la surexpression de ZNF703 induisait effectivement une augmentation de la population de CSC et favorisait également la différenciation luminale des progéniteurs épithéliaux de la glande mammaire [4, 5]. Ces observations sont en accord avec le rôle connu du complexe nucléaire NCOR2 qui contrôle l’activité transcriptionnelle des gènes maîtres ESR1/RE (récepteur des oestrogènes) et E2F1. Ces deux régulateurs transcriptionels sont impliqués dans le contrôle des processus d’autorenouvellement et de différenciation des cellules souches. Les auteurs ont notamment démontré l’implication de ZNF703 dans la régulation négative de l’activité transcriptionnelle de RE et dans l’activation d’E2F1, contrôlant ainsi la phase G1 du cycle cellulaire qui est une étape cruciale pour le bon déroulement de la division asymétrique, propriété-clé du processus d’autorenouvellement des CSC. Cette balance entre la répression de RE et l’activation d’E2F1 est retrouvée dans les tumeurs du sein de sous-type luminal B, où les cibles transcriptionelles de RE sont fortement inhibées alors que les gènes cibles d’E2F1 sont fortement activés [4]. Ce mécanisme pourrait être à l’origine de l’hormonorésistance des tumeurs luminales B. |
ZNF703, un oncogène reprogrammateur Les oncogènes sont définis comme des gènes qui commandent la synthèse d’oncoprotéines (protéines stimulant la division cellulaire) et déclenchent une prolifération désordonnée des cellules. La nouvelle vision de l’organisation hiérarchique de la masse tumorale implique que les gènes maîtres de l’oncogenèse agissent d’une part sur la prolifération et d’autre part sur les processus d’autorenouvellement et de différenciation des cellules souches. ZNF703 regroupe toutes ces propriétés et pourrait être considéré comme un nouveau type d’oncogène « reprogrammateur ». De plus en plus d’études décrivent des fonctions de reprogrammation pour des suppresseurs de tumeur connus tels que TP53 ou BRCA1 et de plus en plus de mutations dans des régulateurs épigénétiques sont identifiées dans les tumeurs par les séquençages de génomes tumoraux entiers. Le développement de stratégies thérapeutiques visant à inhiber ces oncoprotéines est crucial pour l’éradication des CSC qui sont responsables de l’activité tumorigénique de la tumeur et de sa récidive. Conflit d’intérêts
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
1.
Charafe-Jauffret
E
,
Chaffanet
M
,
Bertucci
F
, et al.
Towards an integrated cellular and molecular : definition of breast cancers . Med Sci (Paris).
2007; ; 23 : :626.-632. 2.
Charafe-Jauffret
E
,
Monville
F
Ginestier
C
, et al.
Cancer stem cells in breast : current opinion and future challenges . Pathobiology.
2008; ; 75 : :75.-84. 3.
Ginestier
C
,
Korkaya
H
,
Dontu
G
,
Birnbaum
D
,
Wicha
MS
,
Charafe-Jauffret
E
. The cancer stem cell : the breast cancer driver . Med Sci (Paris).
2007; ; 23 : :1133.-1139. 4.
Sircoulomb
F
,
Nicolas
N
,
Ferrari
A
, et al.
ZNF703 gene amplification at 8p12 specifies luminal B breast cancer . EMBO Mol Med.
2011; ; 3 : :153.-166. 5.
Holland
D
,
Burleigh
A
,
Git
A
, et al.
ZNF703, a luminal B breast cancer oncogene, is a transcriptional repressor and differentially regulates luminal and basal progenitors in human mammary epithelium . EMBO Mol Med.
2011; ; 3 : :167.-180. 6.
Turner
N
,
Pearson
A
,
Sharpe
R
, et al.
FGFR1 amplification drives endocrine therapy resistance and is a therapeutic target in breast cancer . Cancer Res.
2010; ; 70 : :2085.-2094. 7.
Zhang
J
,
Liu
X
,
Datta
A
, et al.
RCP is a human breast cancer-promoting gene with Ras-activating function . J Clin Invest.
2009; ; 119 : :2171.-2183. 8.
Bernard-Pierrot
I
,
Gruel
N
,
Stransky
N
, et al.
Characterization of the recurrent 8p11-12 amplicon identifies PPAPDC1B, a phosphatase protein, as a new therapeutic target in breast cancer . Cancer Res.
2008; ; 68 : :7165.-7175. 9.
Kwek
SS
,
Roy
R
,
Zhou
H
, et al.
Co-amplified genes at 8p12 and 11q13 in breast tumors cooperate with two major pathways in oncogenesis . Oncogene.
2009; ; 28 : :1892.-1903. 10.
Nakamura
M
,
Runko
AP
,
Sagerstrom
CG
. A novel subfamily of zinc finger genes involved in embryonic development . J Cell Biochem.
2004; ; 93 : :887.-895. |