| |
| Med Sci (Paris). 2011 April; 27(4): 343–345. Published online 2011 April 28. doi: 10.1051/medsci/2011274003.Une approche originale de sélection de nouveaux ARN non codants Mathieu Rederstorff1* AREMS, UHP-CNRS UMR 7214, Faculté des sciences et technologies, Nancy Université, boulevard des Aiguillettes, BP 70239, 54506Vandœuvre-lès-Nancy, France |
Les microARN : des marqueurs pour le diagnostic L’utilisation de plus en plus fréquente des ARNnc, et en particulier des miARN, en tant qu’outils de diagnostic ou de thérapie est toutefois limitée par de nombreuses difficultés techniques. De nombreux miARN sont observés de façon stable dans différents fluides biologiques comme le sang ou l’urine et peuvent ainsi être utilisés comme marqueurs diagnostiques [
3]. Des miARN foetaux circulant dans le sang maternel peuvent même permettre de détecter une grossesse chez la femme [3]. De manière encore plus cruciale, l’expression de nombreux miARN étant dérégulée en cas de cancer [
4,
13,
14], leur identification dans le sang permet des diagnostics de plus en plus précis. Par exemple, la restriction de l’expression de plusieurs miARN à des tissus précis peut renseigner sur l’origine de la tumeur primaire en cas de métastases, ce qui est indispensable à la mise en place du traitement adéquat [
5]. L’ensemble de ces approches diagnostiques a notamment bénéficié des progrès récents des nouvelles techniques de séquençage à haut-débit, très sensibles, et qui peuvent conduire actuellement à obtenir des millions de séquences en une seule expérience, permettant même la détection de miARN très peu abondants. Le point crucial, et souvent limitant, de ces approches à haut-débit demeure toutefois l’étape de génération des banques d’ARN à séquencer [
6]. |
Identification de nouveaux ARN noncodants : les approches à haut-débit Chez les eucaryotes, la grande majorité, si ce n’est la totalité des ARNnc fonctionnels sont contenus dans des complexes ribonucléoprotéiques (RNP) de grande taille. Dans ces complexes, la molécule d’ARN peut être associée à différentes protéines qui se lient à l’ARN pour former des microRNP (avec des miARN) ou des petites RNP nucléolaires (avec des petits ARN nucléolaires ou snoARN). Afin de distinguer, parmi l’ensemble des transcrits présents dans la cellule, ceux qui sont fonctionnels et biologiquement actifs de ceux qui ne le sont pas, une nouvelle approche de génération de banques d’ADN complémentaires (ADNc) des ARNnc [
7,
8]. Jusqu’alors, ce type de banques était généré à partir d’ARN nus extraits au phénol-chloroforme et séparés sur gel de polyacrylamide dénaturant, ce qui permettait une sélection selon leur taille des molécules à inclure dans la banque [
9,
10]. Toutefois, l’inconvénient majeur de ces banques était qu’elles contenaient de façon majoritaire des produits de dégradation non fonctionnels d’ARN ribosomiques ou d’ARN messagers, parfois dans des proportions supérieures à 90 % [10], masquant et empêchant ainsi, lors du séquençage, l’identification de nouvelles molécules plus rares. Notre nouvelle approche est basée sur la sélection de RNP en conditions natives sur gradient de glycérol en amont de l’étape d’isolement des ARN (Figure 1), ce qui permet d’enrichir les banques en ARN fonctionnels inclus dans des RNP, contrairement à l’approche traditionnelle, et d’identifier de nouveaux ARNnc régulateurs [7]. La comparaison des séquences obtenues pour deux banques d’ARNnc issus de cerveaux de souris préparées selon les deux approches est sans ambiguïté (Figure 2). Alors que près de la moitié des séquences obtenues avec une approche classique correspondent à des fragments d’ARN ribosomiques non fonctionnels [9], ce pourcentage n’est que de 2 % avec l’approche par sélection des RNP, ce qui a permis d’obtenir 98 % d’ARNnc fonctionnels dans la banque [7]. Par une approche à haut-débit, le séquençage de cette banque a en outre permis d’identifier environ un millier de nouveaux candidats petits ARNnc dont la caractérisation fonctionnelle est en cours [7].
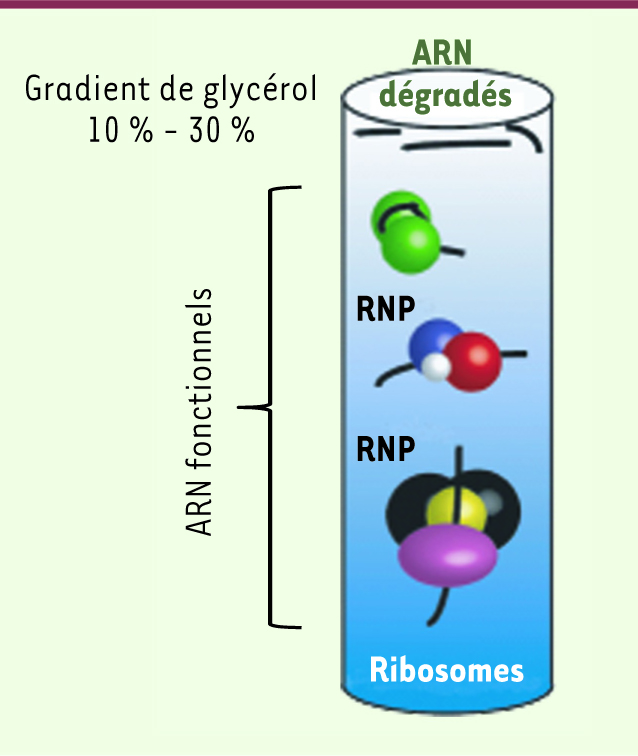
 | Figure 1
Principe de l’enrichissement en particules ribonucléoprotéiques
. La sélection de particules ribonucléoprotéiques (RNP) sur gradient de glycérol permet d’enrichir les banques qui en résultent en ARN fonctionnels. En effet, seuls les ARN associés à des protéines au sein des RNP, et dont la probabilité d’être des ARN fonctionnels est ainsi accrue, sont analysés. Les ARN dégradés non associés à des protéines ou les sousunités ribosomiques que l’on trouve au sommet du gradient ou dans le culot respectivement, sont exclus du processus, ce qui permet d’éviter qu’ils soient ensuite séquencés en majorité. Adapté avec la permission de [ 7]. |
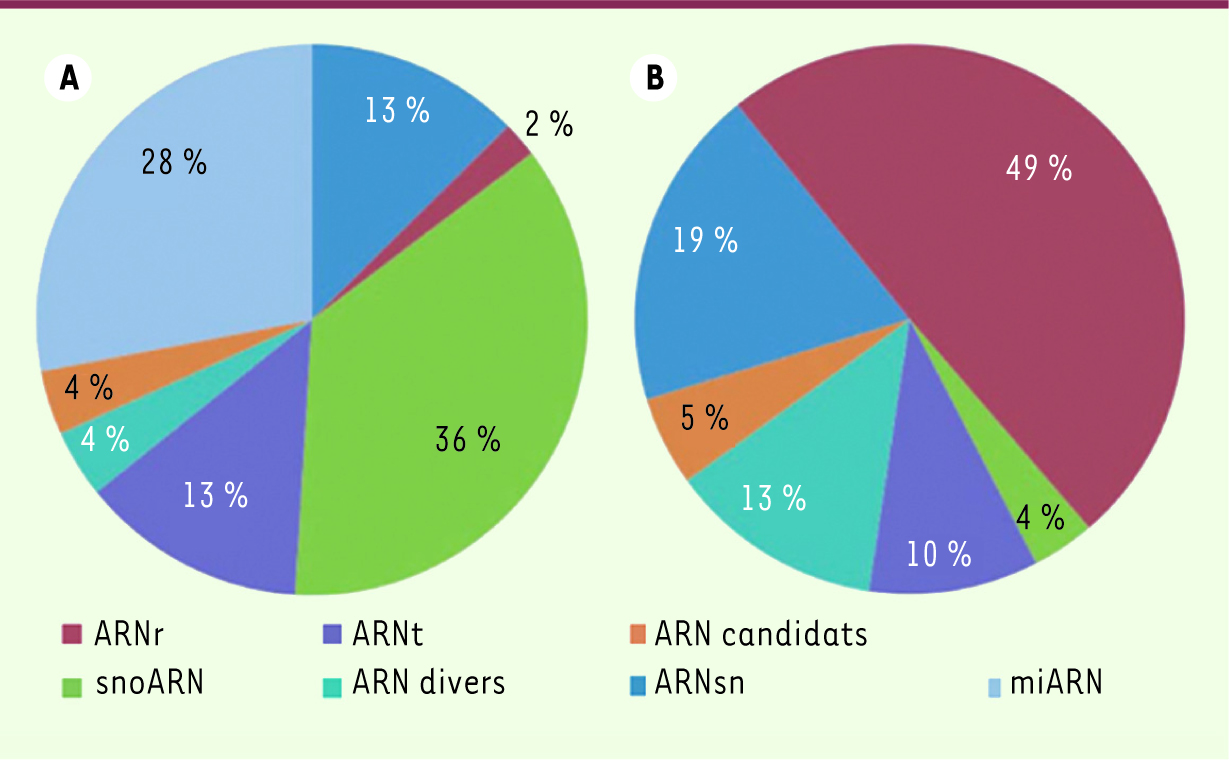
 | Figure 2
Séquençage de deux banques d’ARN non codants de cerveau de souris. A. Distribution des 31 882 séquences obtenues par séquençage à haut-débit d’une banque préparée selon l’approche RNP à partir de cellules de cerveau murin [ 7]. Les proportions d’ADNc correspondant aux différentes catégories d’ARNnc sont rapportées au nombre total de séquences. B. Distribution des séquences de 800 clones choisis au hasard et issus d’une banque d’ADNc classique préparée à partir d’ARN nus de tailles comprises entre 50 et 500 nucléotides [ 9]. Les proportions d’ADNc correspondant aux différentes catégories d’ARNnc sont rapportées au nombre total de séquences. ARNr, ARN ribosomique ; ARNt, ARN de transfert ; miARN, microARN ; snoARN, petit ARN nucléolaire ; ARNsn, petit ARN nucléaire. La proportion d’ARNr pour la même banque atteint 72% si des ARN de tailles comprises entre 110 et 500 nucléotides sont utilisés. Reproduit avec la permission de [ 8]. |
L’obtention de banques à partir des RNP constitue donc une bonne alternative aux protocoles existants, et permet une étude du transcriptome par des approches de séquençage à haut-débit (technologie RNAseq) [8]. Ces approches par séquencage tendent même à dépasser aujourd’hui les approches d’analyse transcriptomique à l’aide des micropuces à ADN qui constituaient la méthode de choix au cours de ces dernières années. En effet, ces dernières approches, bien qu’extrêmement informatives, ne permettent toujours pas l’identification de nouveaux ARNnc, puisqu’elles sont basées sur la connaissance a priori des séquences à étudier qui doivent être déposées sur la puce. |
Les principaux ARN cellulaires non codants et leurs fonctions
|
ARNr 120-4700nt :
|
ARN ribosomique. Composants ARN des ribosomes (5S, 5,8S, 18S, 28S). |
|
ARNt 70-95nt :
|
ARN de transfert. Molécules adaptatrices chargées avec les acides aminés : l’ARNt apporte l’acide aminé au ribosome de manière ARNm dépendante. |
|
miARN 20-25nt :
|
MicroARN. ARN maturés à partir de tiges boucles précurseurs : les miARN réduisent l’expression des protéines en agissant sur la région 3’ non traduite de l’ARNm cible. |
|
siARN 20-25nt :
|
Petits ARN interférents. Produits par clivage de précurseurs double brin : les siARN entraînent le clivage de l’ARN cible. |
|
piARN 26-31nt :
|
ARN associés à Piwi. Petits ARN liés à l’inactivation d’éléments transposables du génome, en particuler dans les cellules germinales. |
|
snARN 100-200nt :
|
Petits ARN nucléaires. Guident l’épissage des ARNm (U1, U2, U4, U5). |
|
snoARN 60-300nt :
|
Petits ARN nucléolaires. Guident la modification des ARNr et des sn ARN. |
|
1.
Huttenhofer
A
,
Schattner
P
,
Polacek
N
. Non-coding RNAs : hope or hype ?
Trends Genet.
2005; ; 21 : :289.-297. 2.
Birney
E
,
Stamatoyannopoulos
JA
,
Dutta
A
, et al.
Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project . Nature.
2007; ; 447 : :799.-816. 3.
Gilad
S
,
Meiri
E
,
Yogev
Y
, et al.
Serum microRNAs are promising novel biomarkers . PLoS One 2008.
3 : :e3148.. 4.
Kahn
A
. L’impérialisme des micro-ARN s’étend maintenant au cancer . Med Sci (Paris) 2005.
21 : :687.-689. 5.
Rosenfeld
N
,
Aharonov
R
,
Meiri
E
, et al.
MicroRNAs accurately identify cancer tissue origin . Nat Biotechnol.
2008; ; 26 : :462.-469. 6.
Saxena
A
,
Carninci
P
. Whole transcriptome analysis : what are we still missing ?
Wiley Interdiscip Rev Syst Biol Med.
2011 ( sous-presse.). 7.
Rederstorff
M
,
Bernhart
SH
,
Tanzer
A
, et al.
RNPomics : defining the ncRNA transcriptome by cDNA library generation from ribonucleo-protein particles . Nucleic Acids Res.
2010; ; 38 : :e113.. 8.
Rederstorff
M
,
Huttenhofer
A
. cDNA library generation from ribonucleoprotein particles . Nat Protoc.
2011; ; 6 : :166.-174. 9.
Huttenhofer
A
,
Kiefmann
M
,
Meier-Ewert
S
, et al.
RNomics : an experimental approach that identifies 201 candidates for novel, small, non-messenger RNAs in mouse . EMBO J.
2001; ; 20 : :2943.-2953. 10.
Jochl
C
,
Rederstorff
M
,
Hertel
J
, et al.
Small ncRNA transcriptome analysis from Aspergillus fumigatus suggests a novel mechanism for regulation of protein synthesis . Nucleic Acids Res.
2008; ; 36 : :2677.-2689. 11.
Derrien
T
,
Guigó
R
. Des longs ARN non codants humains activateurs de la transcription des gènes . Med Sci (Paris).
2011; ; 27 : :359.-361. 12.
Baudry
A
,
Mouillet-Richard
S
,
Schneider
B
, et al.
Le microARN-16 : une clé de l’adaptation des neurones à la fluoxétine . Med Sci (Paris).
2011; ; 27 : :128.-131. 13.
Ladeiro
Y
,
Zucman-Rossi
J
. MicroARN et cancer : le cas des tumeurs hépatocellulaires . Med Sci (Paris).
2009; ; 25 : :467.-472. 14.
Bousquet
M
,
Dastugue
N
,
Brousset
P
. MicroARN et translocations chromosomiques dans les hémopathies malignes . Med Sci (Paris).
2009; ; 25 : :229.-231. |