| |
| Med Sci (Paris). 2011 March; 27(3): 229–231. Published online 2011 March 30. doi: 10.1051/medsci/2011273229.Des communautés de phages divergentes contrôlent-elles la microflore intestinale ? Florent Angly1* and Gene Tyson1 1Australian centre for ecogenomics and Advanced water management centre, The University of Queensland, Brisbane St Lucia, Australie MeSH keywords: Bactéries, virologie, Bactériophages, Humains, Intestins, microbiologie, Métagénome |
Les phages, des virus spécialisés dans l’infection de bactéries, furent découverts par le scientifique franco-canadien Félix d’Hérelle en 1917. Ils furent ensuite trouvés dans toutes sortes d’environnements sur Terre, y compris dans les fèces d’un grand nombre d’animaux [
1]. C’était la première indication que les intestins abritent des phages, et des études récentes ont montré qu’ils sont nombreux dans les intestins de l’homme (Tableau I) [
2]. Même si les phages sont bien plus abondants que les virus qui infectent les eucaryotes, leur présence permanente dans le corps humain n’est pas un signe de maladie en soi. Cependant, les phages pourraient influencer la santé humaine en tant que prédateurs des bactéries intestinales, qui sont responsables de processus métaboliques critiques tels que la digestion de composés complexes et la synthèse de vitamines et acides aminés essentiels [
3]. Tableau I
|
Abondance totale |
Nombre d’espèces |
Stœchiométrie |
Diversité globale** |
Taxons prévalents |
| Phages* |
1012 à 1013 particules |
Environ 110 |
0,91 (maximum 1) |
~ 3,3 |
Podoviridés et phages tempérés |
| Bactéries |
1013 à 1014 cellules |
200 à 800 |
30 à 40 espèces représentent 99 % de la communauté |
~ 3,2 |
Firmicutes et Bacteroidetes |
Comparaison des communautés de phages et bactéries dans l’intestin humain. *Ces chiffres sont basés sur le nombre de phages libres observés. Le nombre total de phages et leur diversité pourraient être plus élevés car la plupart des phages intestinaux sont tempérés et s’intègrent dans leur hôte pendant de longues périodes. **Index de Shannon (utilise l’indice de Shannon qui permet de mesurer la biodiversité). |
|
L’analyse métagénomique : un outil pour l’étude de la diversité des phages dans l’intestin Comme la plupart des bactéries et phages ne peuvent pas pousser en culture pure au laboratoire, les études originelles basées sur leur culture n’offrirent que peu d’informations sur l’identité des phages présents dans les intestins et des bactéries qu’ils infectent. Durant la dernière décennie, le développement de la métagénomique, basée sur le séquençage de fragments d’acides nucléiques extraits d’échantillons environnementaux, a permis de décrire de manière plus précise la composition de communautés de phages et bactéries non cultivés. En 2003, pour la première fois, Breitbart et al. [
4] séquencèrent le métagénome des phages intestinaux humains et identifièrent de nombreuses séquences provenant de phages nouveaux. Les séquences qui pouvaient être classifiées appartenaient principalement à la famille de phages Siphoviridés et à des prophages - des phages lysogéniques1 qui insèrent leur génome dans le génome de leur hôte bactérien pendant une longue période. Pour déterminer si des différences significatives existent entre les communautés de phages d’individus non apparentés et évaluer si l’inné (le génome d’un individu) ou l’acquis (les habitudes alimentaires et l’environnement d’un individu) conditionnent leur composition, Reyes et ses collègues [
5] collectèrent des échantillons fécaux de quatre paires de jumeaux et de leurs mères pendant un an. Les phages furent soumis à une analyse métagénomique et les bactéries de ces mêmes échantillons furent étudiées par le séquençage d’un gène marqueur, l’ARNr 16S. La diversité des phages et bactéries était modérée (entre 10 et 1 000 espèces de phages et 800 espèces de bactéries), et le nombre d’espèces de phages par espèce bactérienne bas comparé à d’autres environnements. Comme dans la première étude métagénomique de phages intestinaux [4], un petit nombre de séquences a pu être classifié et la majorité de ces séquences provenaient de phages lysogéniques. Cependant, des phages à ADN simple brin et des phages Podoviridés furent aussi détectés. D’autre part, les métagénomes furent comparés deux à deux à l’aide d’un algorithme qui peut utiliser toutes les séquences pour estimer les variations entre communautés de phages [
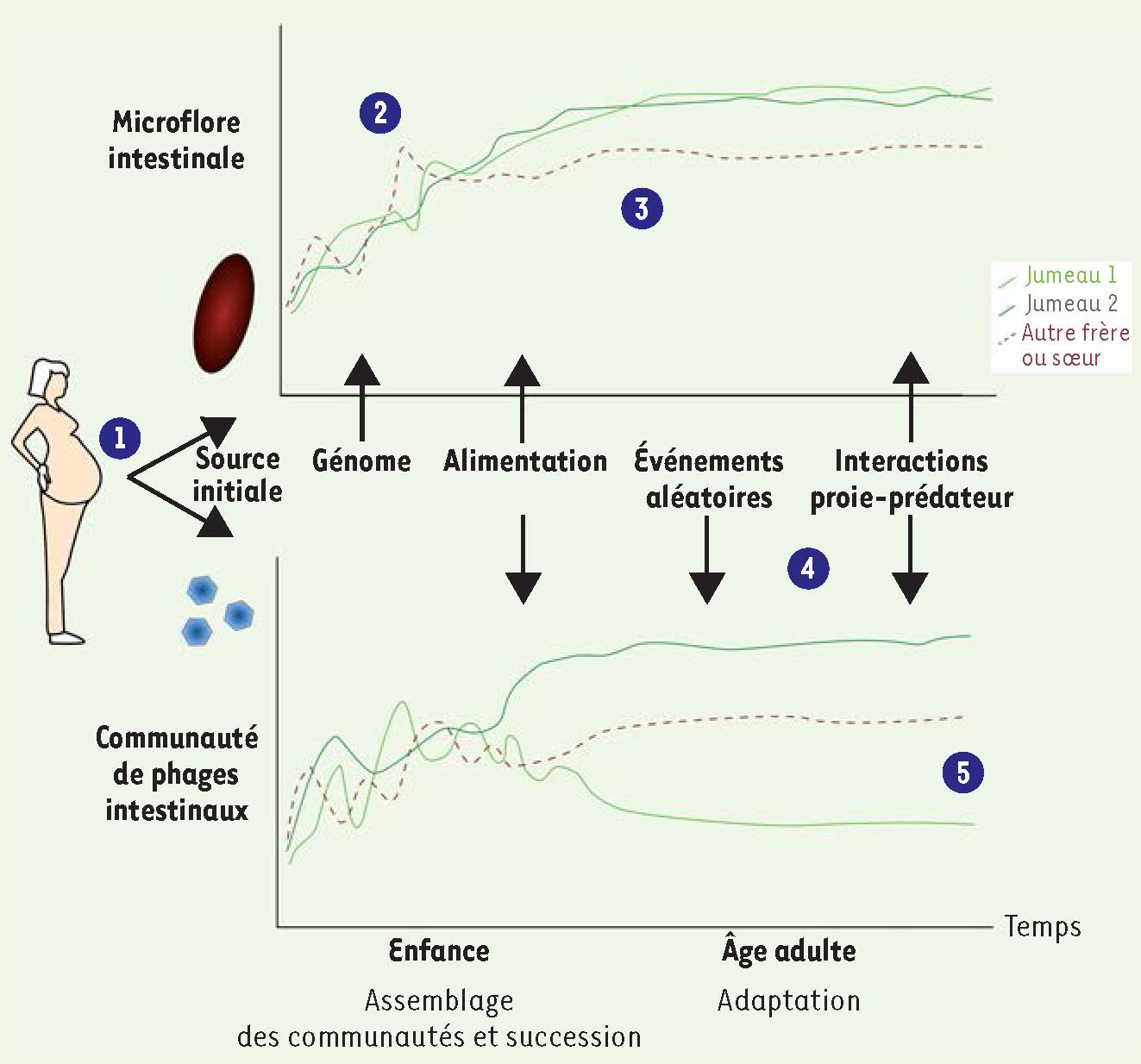
6]. Cette analyse chez les paires de jumeaux - qui ont un patrimoine génétique identique - montra que chacun avait un consortium de phages qui lui était propre, ce qui suggère que l’acquis - et non pas l’inné - décide principalement de la structure des communautés de phages dans les intestins (Figure 1). Ce résultat est surprenant parce que les bactéries, dont les phages dépendent pour leur réplication, sont, elles, sélectionnées par l’inné plutôt que l’acquis. Reyes et ses collègues démontrèrent dans leur analyse temporelle que l’abondance des populations de phages et bactéries ne changeait pas significativement dans les intestins, contrairement à d’autres environnements où ils oscillent rapidement de manière coordonnée en raison de l’intensité des relations proie-prédateur [
7]. Il n’y avait pas non plus de signe de coévolution (dynamique de la Reine Rouge2 [
8]) entre la virulence des phages et les défenses de leurs hôtes durant un an. Une interprétation possible est que la dynamique et l’évolution des phages et bactéries sont lentes dans les intestins, à cause de l’abondance des prophages. Cependant, la raison de la prévalence du style de vie lysogénique reste à expliquer quand on considère que la concentration de bactéries est élevée dans les intestins.  | Figure 1
Un modèle des étapes du développement de la microflore et des phages intestinaux. 1. Les premières bactéries et phages intestinaux du nouveau-né proviennent de sa mère. 2. De grandes fluctuations se produisent dans la microflore intestinale du bébé alors que ses intestins sont colonisés par des bactéries pionnières (et leurs phages) en vagues successives. 3. Lors du développement de l’enfant, sa communauté de bactéries se stabilise sur une trajectoire définie surtout par son génome, mais aussi par son environnement (alimentation, mode de vie). 4. Les phages sont dépendants des bactéries et chaque type de bactérie peut être infecté par plusieurs espèces de phages. Des événements aléatoires décident quels sont les phages qui persistent. 5. Chez l’adulte, la communauté de bactéries et phages spécialisés s’adapte lentement. Les communautés bactériennes d’individus apparentés ont des ressemblances. Les communautés de phages, elles, sont propres à l’individu, même chez les individus apparentés. |
|
Une communauté de phages « personnalisée » : un rôle bénéfique méconnu pour la santé humaine ? Les résultats de l’étude sur les jumeaux révèlent que le tube digestif de chaque individu peut être considéré comme un habitat isolé par des barrières qui freinent l’import de nouvelles espèces dans le tube digestif. À la naissance, des espèces pionnières provenant de la mère s’établissent dans le tube digestif encore vierge en vagues successives. Nous pensons que ces espèces initiales sont suivies par des espèces spécialisées, jusqu’à ce que toutes les niches écologiques des intestins soient occupées. Au cours du temps, l’évolution lente et isolée des espèces dans les intestins d’individus différents forge des communautés divergentes (Figure 1). Comme chaque individu a une communauté de phages unique, trouver une signature virale représentative d’un état sain ne sera pas aussi évident que pour les bactéries. En effet, les Firmicutes et Bacteroidetes représentent la quasi-totalité de la microflore bactérienne d’individus en bonne santé et ce profil métabolique est stable [
9]. Pour cette raison, il sera sûrement difficile d’utiliser les communautés de phages comme outils diagnostiques pour suivre la santé de la microflore dans le futur. Pour autant, bien que les phages ne causent pas une mortalité importante de bactéries dans les intestins, les utiliser en médecine peut avoir un intérêt, car les phages peuvent conférer une protection bénéfique contre des bactéries pathogènes spécifiques. Cette caractéristique a été exploitée par des scientifiques de l’ex-Union soviétique et de l’Europe au cours des soixante dernières années. Ainsi, avant même que les antibiotiques n’existent, certaines infections bactériennes étaient déjà combattues par l’administration de combinaisons de phages capables de lyser diverses bactéries communes. Dans les intestins, les communautés de phages et bactéries semblent s’être adaptées étroitement les unes aux autres pendant de nombreuses années. Si une bactérie pathogène apparaît, elle aura donc de grandes chances d’être neutralisée par l’émergence d’un phage qui peut la tuer, réduisant ainsi les risques de maladie. Dans le modèle que nous proposons, les phages intestinaux maintiennent naturellement une flore bactérienne équilibrée en agissant comme un cocktail « personnalisé » de phages. Des infections intestinales par la bactérie pathogène Clostridium difficile ont récemment été traitées par la greffe de la microflore intestinale d’une personne saine [
10] et il est probable que les phages transplantés de façon concomitante ont joué un rôle important dans la rémission de ces patients, rôle qui devrait être exploré plus en détail dans le futur. La fonction lysogénique des phages intestinaux a de profondes implications pour le métabolisme des bactéries et donc la santé humaine. De nombreuses bactéries et phages sont avalés durant l’ingestion de nourriture. L’acidité de l’estomac empêche probablement la majorité d’entre eux de parvenir à l’intestin, mais les phages qui y parviennent ont certaines chances d’infecter un nouvel hôte bactérien [
11]. Comme les jumeaux de l’étude de Reyes, qui partagent en grande partie le même style alimentaire, ont des communautés de phages intestinaux différents, ce phénomène est relativement peu fréquent, mais a des conséquences potentiellement importantes. Les phages lysogéniques qui ont acquis des gènes dans l’environnement (par exemple associé au métabolisme du fer, du sulfure et des glucides) ont la possibilité de transférer ces gènes horizontalement à leur nouvel hôte bactérien dans les intestins. L’environnement est donc comparable à un réservoir de gènes que les phages pourraient transporter dans d’autres biomes3 comme les intestins. Ce processus permet vraisemblablement l’adaptation de populations humaines entières aux aliments qu’ils consomment, ce qu’illustrent les enzymes porphyranases et agarases marines présentes dans les intestins de la population japonaise et nécessaires à la digestion d’algues qu’ils consomment abondamment dont médecine/sciences s’est récemment fait l’écho [
12]. Alors que les virus d’eucaryotes sont sous les projecteurs parce qu’ils entraînent des maladies humaines, les phages sont souvent ignorés, alors même qu’ils semblent contribuer à la maintenance de fonctions critiques du corps humain dans la vie quotidienne. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Footnotes |
1.
Dhillon
TS
,
Dhillon
EK
,
Chau
HC
, et al.
Studies on bacteriophage distribution: virulent and temperate bacteriophage content of mammalian feces . Appl Environ Microbiol.
1976; ; 32 : :68.-74. 2.
Breitbart
M
,
Haynes
M
,
Kelley
S
, et al.
Viral diversity and dynamics in an infant gut . Res Microbiol.
2008; ; 159 : :367.-73. 3.
Bäckhed
F
,
Ley
RE
,
Sonnenburg
JL
, et al.
Host-bacterial mutualism in the human intestine . Science.
2005; ; 307 : :1915.-20. 4.
Breitbart
M
,
Hewson
I
,
Felts
B
, et al.
Metagenomic analyses of an uncultured viral community from human feces . Bacteriol.
2003; ; 185 : :6220.-3. 5.
Reyes
A
,
Haynes
M
,
Hanson
N
, et al.
Viruses in the faecal microbiota of monozygotic twins and their mothers . Nature.
2010; ; 466 : :334.-8. 6.
Angly
FE
,
Felts
B
,
Breitbart
M
, et al.
The marine viromes of four oceanic regions . PLoS Biol.
2006; ; 4 : e368.. 7.
Rodriguez-Brito
B
,
Li
L
,
Wegley
L
, et al.
Viral and microbial community dynamics in four aquatic environments . ISME J.
2010; ; 4 : :739.-51. 8.
Weitz
JS
,
Hartman
H
,
Levin
SA.
. Coevolutionary arms races between bacteria and bacteriophage . Proc Natl Acad Sci USA.
2005; ; 102 : :9535.-40. 9.
Turnbaugh
PJ
,
Hamady
M
,
Yatsunenko
T
, et al.
A core gut microbiome in obese and lean twins . Nature.
2009; ; 457 : :480.-4. 10.
Grehan
MJ
,
Borody
TJ
,
Leis
SM
, et al.
Durable alteration of the colonic microbiota by the administration of donor fecal flora . Clin Gastroenterol.
2010; ; 44 : :551.-61. 11.
Sano
E
,
Carlson
S
,
Wegley
L
, et al.
Movement of viruses between biomes . Appl Environ Microbiol.
2004; ; 70 : :5842.-6. 12.
Czjzek
M
,
Correc
G
,
Michel
G
. Sommes-nous ce que nous mangeons ? Le transfert de gènes du milieu marin vers la microflore intestinale chez les Japonais . Med Sci (Paris).
2010; ; 26 : :811.-3. |