| |
| Med Sci (Paris). 2010 December; 26(12): 1056–1060. Published online 2010 December 15. doi: 10.1051/medsci/201026121056.mTOR, la cible fonctionnelle de la rapamycine Louis-André Julien and Philippe P. Roux* Institut de recherche en immunologie et en cancérologie (IRIC), Département de pathologie et biologie cellulaire, Faculté de médecine, Université de Montréal, CP 6128, Succursale Centre-ville, Montréal (Québec), H3C 3J7 Canada |
La découverte de la rapamycine remonte à 1964. Elle a été isolée à partir d’échantillons de sol recueillis sur l’île de Pâques (Figure 1A) qui ont été envoyés au laboratoire du Dr Suren N. Seghal à Montréal. La bactérie Streptomyces hygroscopicus (Figure 1B) fut isolée et les propriétés antibiotiques d’un de ses produits, un macrolide nommé rapamycine en référence à Rapa Nui, le nom indigène de l’île de Pâques, furent étudiées [
1]. D’abord utilisée comme agent antifongique, la rapamycine (Figure 1C) fut reléguée aux oubliettes en raison de ses propriétés immunosuppressives. On sait qu’au début des années 1980, l’arrivée des agents immunosuppresseurs que sont la ciclosporine et le tacrolimus (FK506) causa une révolution dans le domaine des transplantations d’organes. Les recherches sur la rapamycine, composé qui possède une structure chimique similaire à celle du FK506, furent relancées en 1987 et la molécule fut commercialisée en 1999 sous le nom de Rapamune® (sirolimus).  | Figure 1
De Rapa Nui à la rapamycine. A. Un moaï, statue gigantesque que l’on trouve par centaines sur l’île de Pâques (photographie d’Artemio Urbina). B. La rapamycine est un produit de la bactérie Streptomyces hygroscopicus qui fut isolée à partir d’un échantillon de sol de l’île de Pâques. Photographie d’une souche de bactérie Streptomyces prise par le Dr Ramon Santamaria, IMB/CSIC-USAL, Espagne. C. Structure chimique de la rapamycine (C51H79NO13). |
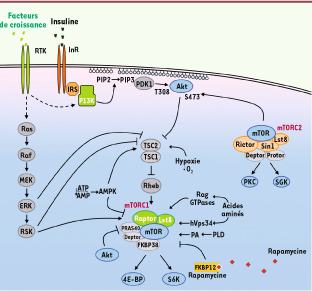
 | Figure 2
Représentation schématique de la voie mTOR. La sérine/thréonine mTOR fait partie de deux complexes fonctionnellement distincts définis par leurs sous-unités spécifiques, Raptor (mTORC1) et Rictor (mTORC2). Les facteurs de croissance qui activent les récepteurs à activité tyrosine kinase (RTK) et le récepteur de l’insuline (InR) activent PI3K, ce qui entraîne la formation de PIP3 dans la membrane plasmique et l’activation de mTORC2 et d’Akt. mTORC2 régule également PKC et SGK. Une fois activé, Akt phosphoryle TSC2 et PRAS40, ce qui induit l’activation de Rheb et de mTORC1. mTORC1, la cible du complexe FKBP12-rapamycine, régule la synthèse protéique en phosphorylant 4E-BP et S6K. L’activation de la voie Ras/MAPK mène également à l’activation de mTORC1 après la phosphorylation de TSC2 par ERK et RSK et celle de Raptor par RSK. L’acide phosphatidique (PA) semble être un régulateur important de mTORC1. Enfin, l’absence d’acides aminés, l’hypoxie et de faibles niveaux d’ATP inhibent l’activation de mTORC1. |
Entre-temps, les propriétés antiprolifératives de la rapamycine furent démontrées envers une diversité de tumeurs solides. En 1989, le groupe du Dr Stuart L. Schreiber établit que le FK506 et la rapamycine pouvaient lier l’immunophiline FKBP12 (FK506 binding protein 12) [
2]. En 1991, Michael N. Hall et N. Rao Movva identifièrent plusieurs levures mutantes de la levure Saccharomyces cerevisiae résistantes à la rapamycine ; la plupart des mutations étaient situées dans le gène FPR1, l’orthologue de FKBP12, mais deux d’entre elles ciblaient de nouveaux gènes, qui furent nommés TOR1 et TOR2, pour target of rapamycin [
3]. En 1994, plusieurs groupes isolèrent, à partir de cellules humaines, une protéine qui s’associait au complexe FKBP12-rapamycine, qui fut nommée FRAP (FKBP rapamycinassociated protein) et RAFT (rapamycin and FKBP12 target), et dont la séquence était similaire à celle du produit des gènes TOR1 et TOR2 identifiés chez la levure [
4,
5]. Bien qu’elle soit ciblée par un complexe FKBP12-rapamycine, la protéine FRAP/RAFT chez les mammifères est couramment appelée mTOR pour mammalian TOR. Dans cet article de synthèse, nous décrirons brièvement les mécanismes de régulation et les fonctions cellulaires de mTOR. |
mTOR, la cible pharmacologique du complexe rapamycine-FKBP12 La délétion homozygote du gène FRAP, situé sur le chromosome 1p36.2 chez l’humain, est létale très tôt au cours du développement embryonnaire murin (E5,5). L’ARN messager (ARNm) codant pour mTOR est exprimé de façon ubiquitaire dans la plupart des tissus et types cellulaires. Le produit du gène FRAP est une sérine/thréonine kinase atypique de la famille des phosphatidylinositol-3-kinase (PI3K)-related kinase (PIKK) [
6]. En plus du domaine kinase, mTOR contient une vingtaine de répétitions HEAT (Huntingtin, elongation factor 3, PR65/A subunit of protein phosphatase 2A, TOR). Leur fonction demeure incomprise mais pourrait régir des interactions protéine-protéine. Pour sa part, mTOR contient aussi des domaines FAT (FRAP/ATM/TRRAP) et FATC (FAT carboxy-terminal) communs à tous les membres de la famille PIKK et importants pour la phosphorylation de substrats par mTOR via un mécanisme inconnu [6]. De plus, mTOR possède un domaine FRB (FKBP12-rapamycin binding) qui est la cible du complexe FKBP12-rapamycine, mais les mécanismes par lesquels cette interaction inhibe l’activité de mTOR demeurent incompris. Enfin, une forme fonctionnelle de mTOR (mTORβ) provenant d’un épissage alternatif a récemment été identifiée et pourrait agir en tant que proto-oncogène [
7]. |
mTOR forme deux complexes intracellulaires La protéine kinase mTOR est le centre catalytique de deux complexes multiprotéiques fonctionnellement distincts, conservés chez tous les eucaryotes et nommés mTORC1 et mTORC2 [
8,
9] (Figure 2). Lorsqu’elle est associée à Raptor (regulatory associated protein of TOR) et à mLST8 (mammalian lethal with sec13 protein 8), mTOR forme le complexe mTORC1. Ce complexe interagit avec Deptor (DEP domaincontaining mTOR-interacting protein), FKBP38 et PRAS40 (prolinerich Akt substrate of 40 kDa), régulateurs négatifs de mTORC1. Pour former mTORC2, mTOR interagit avec les protéines Rictor (rapamycininsensitive companion of TOR), Sin1 (stress-activated map kinaseinteracting protein 1) et mLST8. De plus, mTORC2 s’associe aussi avec Deptor, qui réprime son activité, ainsi qu’avec PPR5/Protor, dont la fonction demeure inconnue. Lorsqu’elle est liée à FKBP12, la rapamycine inhibe spécifiquement mTORC1, mais la cause exacte de cette spécificité demeure encore incomprise. |
mTORC1 et la synthèse protéique Régulation de l’activité de mTORC1 L’activité de mTORC1 est modulée par la disponibilité en nutriments, l’état énergétique de la cellule et par les facteurs de croissance (mitogènes et hormones) [
10] (Figure 2). De plus, régulée par plusieurs proto-oncogènes et suppresseurs de tumeurs, mTORC1 est activée dans un nombre croissant de néoplasmes. Liée au GTP, la petite GTPase Rheb (Ras homologue enriched in brain) interagit avec mTOR et active spécifiquement la signalisation mTORC1 [
11]. Le complexe dimérique formé de TSC1 (hamartine) et TSC2 (tubérine) est un régulateur négatif important de mTORC1 puisque son activité GAP (GTPase activating protein) hydrolyse la molécule de GTP liée à Rheb activée. Le complexe TSC1/2 constitue un carrefour ciblé par plusieurs voies de signalisation. L’activation de PI3K par les facteurs de croissance mène à l’accumulation dans la membrane plasmique de phosphatidylinositol-3,4,5-triphosphate (PIP3), phospholipide important pour le recrutement et l’activation de PDK1 (phosphoinositi-dedependent kinase-1) et de l’oncogène Akt [11]. Une fois activé, Akt phosphoryle TSC2 et inhibe son activité GAP, permettant l’activation de mTORC1 par Rheb. Akt active également mTORC1 en phosphorylant PRAS40. De plus, les kinases ERK (extracellular signal-regulated kinase) et RSK (p90 ribosomal S6 kinase), en aval de la voie de signalisation de la petite GTPase Ras, activent mTORC1 en phosphorylant et inactivant TSC2 [
12]. Enfin, les kinases RSK augmentent l’activité de mTORC1 en phosphorylant directement Raptor [
13]. Lorsque les niveaux énergétiques de la cellule sont faibles, l’AMPK (AMP-activated protein kinase) diminue l’activité de mTORC1 en phosphorylant TSC2 et Raptor. De son côté, mTORC1 est également sensible à la disponibilité en nutriments, notamment en acides aminés, qui l’activent indépendamment du complexe TSC1/2. En effet, les acides aminés envoient mTORC1 à la surface d’un compartiment membranaire intracellulaire qui contient Rheb. Ce mécanisme, qui requiert les petites GTPases Rag, est nécessaire pour l’activation de mTORC1 par Rheb [
14]. De plus, les acides aminés activent mTORC1 grâce à une PI3K de classe III, hVps34 (human vacuolar protein sorting 34), activée par la voie Ca2+/calmoduline [
15]. En outre, l’hypoxie diminue l’activité de mTORC1 via un mécanisme requérant REDD1 (regulated in development and DNA damage response gene 1) et REDD2 [
16]. L’inhibition de mTORCl en réponse à l’hypoxie, à l’absence de nutriments ou à de faibles niveaux énergétiques, est dominante sur les signaux qui l’activent, empêchant son activation lorsque les conditions ne sont pas adéquates pour la croissance cellulaire. Enfin, l’acide phosphatidique (PA), produit lipidique de la phospholipase-D (PLD), lie le domaine FRB de mTOR et semble être un régulateur important de mTORC1 [
17]. La traduction des ARNm La traduction des ARNm est une étape déterminante de l’expression génique dont dépendent la différenciation, la croissance et la prolifération cellulaires. Il faut souligner que mTORC1 est un régulateur majeur de la synthèse protéique puisqu’il régit la formation du complexe d’initiation de la traduction eIF4F (elongation initiation factor 4F) [ 10], formé des sous-unités eIF4E, eIF4A et eIF4G. La sous-unité eIF4E est un oncogène surexprimé dans plusieurs types de néoplasmes car il promeut la traduction des ARNm en liant directement la structure de la coiffe. La formation de eIF4F à la coiffe est inhibée par les protéines 4E-BP (4E-binding proteins), qui déplacent eIF4G pour se lier à eIF4E. En phosphorylant les 4E-BP, mTORC1 inhibe leur liaison à eIF4E et favorise la formation du complexe eIF4F. Le complexe mTORC1 régule un autre effecteur de la croissance cellulaire en phosphorylant le motif hydrophobique de S6K (p70 ribosomal S6 kinase), une kinase de la famille AGC (protéine kinase A, G et C). Une fois activée, S6K phosphoryle eIF4B, favorisant son association au complexe de pré-initiation de la traduction [10], et d’autres substrats qui régissent la synthèse protéique, dont la protéine ribosomale S6 de la sous-unité 40S du ribosome, eEF2K (eukaryotic translation elongation factor 2 kinase) et PDCD4 (programmed cell death protein 4).
La biogenèse des ribosomes mTORC1 coordonne la biosynthèse des ribosomes en entraînant la traduction d’ARNm possédant une séquence terminale en 5’ de 6 à 12 pyrimidines, nommés 5’TOP (5’ terminal oligo-pyrimidine) [ 10]. Cette classe d’ARNm code pour des facteurs d’élongation de la traduction, certains facteurs d’initiation ainsi que la majorité des protéines ribosomales. La façon dont mTORC1 conduit la traduction de ces ARNm reste inconnue. De plus, mTORC1 module l’activité de l’ARN polymérase I, qui transcrit les ARNr, en favorisant l’activité du régulateur transcriptionnel TIF-1A (transcriptional intermediary factor 1A). Même si les mécanismes ne sont pas connus, il semble que mTORC1 module la synthèse de l’ARN 5S et des ARN de transfert (ARNt) par l’ARN polymérase III. Enfin, chez la levure, l’homologue de mTORC1 (TOR1) lie le promoteur du gène de l’ARNr 35S, ce qui est important pour sa synthèse et pour la croissance cellulaire [
18]. Autres fonctions importantes de mTORC1 Le complexe mTORC1 participe à la régulation de l’autophagie, mécanisme de dégradation non spécifique provoqué par une carence en nutriments et qui permet à la cellule de recycler de vieux organites et protéines. Chez la levure, TOR1 bloque l’association entre ATG13 (autophagic specific gene 13) et ATG1 [
19], processus essentiel à la formation d’autophagosomes, tandis que la rapamycine déclenche l’autophagie dans des tissus murins et humains, indiquant que mTORC1 est un inhibiteur de l’autophagie [
20]. L’hypothèse selon laquelle l’autophagie pourrait permettre à la cellule cancéreuse de survivre et de contourner l’apoptose en absence de nutriments demeure sujette à débat et le rôle entre l’autophagie et mTOR dans la tumorigenèse reste à déterminer. Le complexe mTORC1 module l’expression de plusieurs transporteurs de nutriments de façon transcriptionnelle et post-traductionnelle, dont celle du transporteur de glucose GLUT1 (glucose transporter 1), de plusieurs transporteurs d’acides aminés et du récepteur de la transferrine [
21]. Le transport du glucose et de nutriments étant un processus souvent accéléré dans les cellules cancéreuses, ces fonctions de mTORC1 pourraient jouer un rôle dans la tumorigenèse. |
Fonctions et régulation de mTORC2 Régulation de l’oncogène Akt L’une des preuves de l’importance du rôle que joue mTOR dans le cancer est attestée par la découverte que l’oncogène Akt, régulateur majeur de la prolifération et de la survie cellulaires, est directement phosphorylé et activé par mTORC2 [
22] (Figure 2). En effet, l’activation d’Akt par les facteurs de croissance nécessite la phosphorylation d’une thréonine dans la boucle d’activation (Thr308) par PDK1 et d’une sérine située dans un site hydrophobe (Ser473) par mTORC2 [ 22]. D’ailleurs, mTORC2 phosphoryle un site similaire sur les kinases SGK1 et PKCα, ce qui signifie qu’une des fonctions de mTOR consiste à activer plusieurs kinases de la famille AGC [
23]. De plus, des études récentes indiquent que mTORC2 pourrait jouer un rôle dans la phosphorylation du turn motif de plusieurs kinases AGC lors de leur synthèse de novo ; ce phénomène serait important pour leur stabilité [ 23]. Enfin, mTORC2 joue un rôle dans la structuration du cytosquelette d’actine puisque la diminution de son expression empêche la polymérisation de l’actine et l’expansion cellulaire [ 9]. Régulation de l’activité de mTORC2 Les mécanismes qui modulent l’activité de mTORC2 sont peu connus. Ce complexe semble être sous la domination de PI3K, car les facteurs de croissance qui l’activent provoquent la phosphorylation d’Akt dépendante de mTORC2. Plusieurs mécanismes de rétrocontrôle déclenchés par mTORC1 semblent inhiber l’activité de mTORC2 [ 11]. Ainsi, bien que Rheb n’interagisse pas avec mTORC2, il semble qu’il soit un antagoniste de son activité : il s’agit probablement d’une conséquence de l’induction de mécanismes de rétro-inhibition lancés par mTORC1. Toutefois, le complexe TSC1/2 semble être un activateur de mTORC2 indépendamment des mécanismes de rétrocontrôle amorcés par mTORC1 car il se lie directement à mTORC2 et l’active par un mécanisme encore méconnu [
24]. L’activation de mTORC1 et de S6K joue un rôle important dans la mise en œuvre de mécanismes de rétrocontrôle qui diminuent l’activation de PI3K et d’Akt [11]. La phosphorylation d’Akt sur la Ser473 est inhibée et insensible à l’insuline dans des cellules embryonnaires de souris TSC2
~/~
dans lesquelles l’activité mTORC1 et S6K est constitutivement élevée. Un traitement par la rapamycine rétablit la sensibilité de ces cellules à l’insuline et permet la phosphorylation d’Akt, indiquant que les mécanismes de rétro-inhibition sont provoqués par mTORC1. En effet, la phosphorylation des protéines adaptatrices IRS (insulin receptor substrate 1) par mTORC1 et S6K a pour effet de diminuer leur stabilité et d’empêcher le recrutement de PI3K [
25]. De plus, des études plus récentes ont démontré que Rictor est phosphorylé par S6K1, ce qui inhibe l’activité de mTORC2 et diminue la phosphorylation et l’activation d’Akt [
26-
28]. |
Nouvelle génération d’inhibiteurs de mTOR La délétion du gène FRAP semble avoir un effet plus marqué que la rapamycine sur la croissance et la prolifération cellulaires, suggérant que certaines fonctions de mTOR ne sont pas entièrement inhibées par la rapamycine [
29]. La deuxième génération d’inhibiteurs mTOR est constituée d’antagonistes de l’ATP, dont la Torin1 et le PP242 [9], qui inhibent mTOR indépendamment du fait qu’il fasse partie de mTORC1 ou mTORC2. De plus, ces inhibiteurs du domaine catalytique de mTOR ont permis d’identifier une activité mTORC1 résistante à la rapamycine ; d’ailleurs, ils bloquent plus fortement la croissance et la prolifération cellulaire que la rapamycine à elle seule. Somme toute, ces inhibiteurs seront utiles pour déterminer les rôles biologiques exacts de mTOR et se profilent comme des agents thérapeutiques prometteurs pour le traitement du cancer. |
Conclusions et perspectives Le rôle qu’exerce mTOR dans plusieurs pathologies justifie la mise au point d’agents thérapeutiques ciblant son activité. Certes la rapamycine et ses analogues représentent des agents dotés d’un fort potentiel pour le traitement de plusieurs cancers, cependant ces molécules semblent inefficaces lorsqu’elles sont utilisées seules, probablement parce qu’elles ne ciblent que mTORC1. La deuxième génération d’inhibiteurs de mTOR, constituée d’antagonistes de l’ATP de son domaine kinase, ciblent mTORC1 et mTORC2 : ils inhibent ainsi plus efficacement les fonctions cellulaires de mTOR. Dès lors, ces nouvelles molécules pourraient être plus efficaces pour le traitement de certains cancers. La rapamycine a été découverte il y a une quarantaine d’années ; nos connaissances sur la régulation et les fonctions de mTOR sont plus récentes et demeurent limitées. Le défi des prochaines années consistera à mieux caractériser cette voie de signalisation afin d’améliorer les stratégies contre le cancer fondées sur l’inhibition de mTOR. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
1. Vezina C, Kudelski A, Sehgal SN. Rapamycin (AY-22,989), a new antifungal antibiotic. I. Taxonomy of the producing streptomycete and isolation of the active principle. J Antibiot (Tokyo) 1975 ; 28 : 721-6. 2. Harding MW, Galat A, Uehling DE, Schreiber SL. A receptor for the immunosuppressant FK506 is a cis-trans peptidyl-prolyl isomerase. Nature 1989 ; 341 : 758-60. 3. Heitman J, Movva NR, Hall MN. Targets for cell cycle arrest by the immunosuppressant rapamycin in yeast. Science 1991 ; 253 : 905-9. 4. Sabatini DM, Erdjument-Bromage H, Lui M, et al. RAFT1: a mammalian protein that binds to FKBP12 in a rapamycin-dependent fashion and is homologous to yeast TORs. Cell 1994 ; 78 : 35-43. 5. Brown EJ, Albers MW, Shin TB, et al. A mammalian protein targeted by G1-arresting rapamycin-receptor complex. Nature 1994 ; 369 : 756-8. 6. Lempiainen H, Halazonetis TD. Emerging common themes in regulation of PIKKs and PI3Ks. EMBO J 2009 ; 28 : 3067-73. 7. Panasyuk G, Nemazanyy I, Zhyvoloup A, et al. mTORbeta splicing isoform promotes cell proliferation and tumorigenesis. J Biol Chem 2009 ; 284 : 30807-14. 8. Dunlop EA, Tee AR. Mammalian target of rapamycin complex 1: signalling inputs, substrates and feedback mechanisms. Cell Signal 2009 ; 21 : 827-35. 9. Guertin DA, Sabatini DM. The pharmacology of mTOR inhibition. Sci Signal 2009 ; 2 : pe24. 10. Ma XM, Blenis J. Molecular mechanisms of mTOR-mediated translational control. Nat Rev Mol Cell Biol 2009 ; 10 : 307-18. 11. Huang J, Manning BD. A complex interplay between Akt, TSC2 and the two mTOR complexes. Biochem Soc Trans 2009 ; 37 : 217-22. 12. Carriere A, Ray H, Blenis J, Roux PP. The RSK factors of activating the Ras/ MAPK signaling cascade. Front Biosci 2008 ; 13 : 4258-75. 13. Carrière A, Cargnello M, Julien LA, et al. Oncogenic MAPK signaling stimulates mTORC1 activity by promoting RSK-mediated raptor phosphorylation. Curr Biol 2008 ; 18 : 1269-77. 14. Kim E, Guan KL. RAG GTPases in nutrient-mediated TOR signaling pathway. Cell Cycle 2009 ; 8 : 1014-8. 15. Gulati P, Gaspers LD, Dann SG, et al. Amino acids activate mTOR complex 1 via Ca2+/CaM signaling to hVps34. Cell Metab 2008 ; 7 : 456-65. 16. Brugarolas J, Lei K, Hurley RL, et al. Regulation of mTOR function in response to hypoxia by REDD1 and the TSC1/TSC2 tumor suppressor complex. Genes Dev 2004 ; 18 : 2893-904. 17. Fang Y, Vilella-Bach M, Bachmann R, et al. Phosphatidic acid-mediated mitogenic activation of mTOR signaling. Science 2001 ; 294 : 1942-5. 18. Li H, Tsang CK, Watkins M, et al. Nutrient regulates Tor1 nuclear localization and association with rDNA promoter. Nature 2006 ; 442 : 1058-61. 19. Kamada Y, Funakoshi T, Shintani T, et al. Tor-mediated induction of autophagy via an Apg1 protein kinase complex. J Cell Biol 2000 ; 150 : 1507-13. 20. Paglin S, Lee NY, Nakar C, et al. Rapamycin-sensitive pathway regulates mitochondrial membrane potential, autophagy, and survival in irradiated MCF-7 cells. Cancer Res 2005 ; 65 : 11061-70. 21. Edinger AL. Controlling cell growth and survival through regulated nutrient transporter expression. Biochem J 2007 ; 406 : 1-12. 22. Bhaskar PT, Hay N. The two TORCs and Akt. Dev Cell 2007 ; 12 : 487-502. 23. Alessi DR, Pearce LR, Garcia-Martinez JM. New insights into mTOR signaling: mTORC2 and beyond. Sci Signal 2009 ; 2 : pe27. 24. Huang J, Dibble CC, Matsuzaki M, Manning BD. The TSC1-TSC2 complex is required for proper activation of mTOR complex 2. Mol Cell Biol 2008 ; 28 : 4104-15. 25. Zick Y. Ser/Thr phosphorylation of IRS proteins: a molecular basis for insulin resistance. Sci STKE 2005 ; 2005 : pe4. 26. Dibble CC, Asara JM, Manning BD. Characterization of Rictor phosphorylation sites reveals direct regulation of mTOR complex 2 by S6K1. Mol Cell Biol 2009 ; 29 : 5657-70. 27. Julien LA, Carrière A, Moreau J, Roux PP. mTORC1-activated S6K1 phosphorylates Rictor on threonine 1135 and regulates mTORC2 signaling. Mol Cell Biol 2010 ; 30 : 908-21. 28. Treins C, Warne PH, Magnuson MA, et al. Rictor is a novel target of p70 S6 kinase-1. Oncogene 2010 ; 29 : 1003-16. 29. Murakami M, Ichisaka T, Maeda M, et al. mTOR is essential for growth and proliferation in early mouse embryos and embryonic stem cells. Mol Cell Biol 2004 ; 24 : 6710-8. |